Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.55 n.3 Ciudad de la Habana sep.-dic. 2003
Aplicación de la enzima SensiScript a un sistema RT-RCP para la obtención de ARN de VIH-1 a partir de muestras de suero almacenadas a -20 oC durante una década
Dra. Sonia Resik Aguirre,1 Dra. Li-Hua Ping,2 Dr. Julio A. Barrios Olivera,3 Dra. Vivian Kourí Cardellá4 y Dr. Ronald Swanstrom5
Resumen
Se demostró la eficacia de la enzima transcriptasa inversa SensiScript para obtener material genético útil en la secuenciación de ácido nucleico de VIH-1, a partir de sueros colectados entre 1989 y 1998 y conservados a temperatura subóptima. Con el empleo de la enzima SensiScript se obtuvo amplificación del ARN del VIH-1 en 86,5 % de las muestras estudiadas, comparado con 20 % al utilizar la enzima AMV-RT. En 13,5 % de las muestras no se obtuvo amplificación con ninguna de las 2 enzimas empleadas.
DeCS: REACCIONES EN CADENA POR POLIMERASA DE TRANSCRIPTASA REVERSA/ métodos; VIH-1; ANALISIS DE SECUENCIA DE ARN; TESTS ENZIMATICOS.
La aplicación de la reacción en cadena de la polimerasa (RCP) en la biología molecular ha tenido un impacto sorprendente. El proceso de RT-RCP (reverso transcripción- reacción en cadena de la polimerasa) es un procedimiento altamente sensible que ha sido utilizado para detectar moléculas de ARN, obtener material útil para secuenciación de ácidos nucleicos y como un paso en el clonaje de copias de ADN complementario (ADNc) a partir de ARN. La amplificación exitosa por RT-RCP requiere de la confluencia de una serie de factores, entre los cuales la calidad de la muestra y el proceso de extracción del ARN desempeñan un papel fundamental.1
En el caso del virus de la inmunodeficiencia humana tipo 1 (VIH-1), agente causal del SIDA, por ser un virus ARN, el uso de la RT-RCP está bien difundido, aplicándose al diagnóstico y la investigación. La capacidad para analizar ARN de VIH a partir de muestras con bajos niveles del virus reviste una gran relevancia,2,3 por lo que los ensayos que incrementan la sensibilidad para la obtención y cuantificación de ARN de VIH están ganando cada vez más en importancia, con la introducción de técnicas de medición de la carga viral utilizadas en el monitoreo de la terapia antirretroviral, la detección temprana en los pacientes de variantes resistentes y el elevado número de investigaciones moleculares que se están realizando para conocer la evolución del virus, la aparición y circulación de nuevos subtipos y recombinantes, y dilucidar el origen de la epidemia.4
Múltiples son los parámetros a tener en cuenta para poder amplificar ARN a partir de muestras clínicas. Entre otros se han estudiado el uso de diferentes anticoagulantes, variados procesos de extracción del material genético, el tiempo que media entre la toma de la muestra y el procesamiento, y la temperatura de conservación, entre otros.5-7
En el presente trabajo se propone la utilización de la enzima transcriptasa inversa SensiScript para obtener ADNc con la calidad necesaria, para ser empleado en la RCP y posterior secuenciación de ácidos nucleicos de VIH-1, a partir de sueros conservados a temperatura subóptima por un período largo de tiempo.
Se estudiaron un total de 74 muestras de suero de pacientes fallecidos, infectados con el VIH-1, colectadas entre 1989 y 1998 y que fueron conservadas a - 20 °C en la seroteca del Laboratorio de Enfermedades de Transmisión Sexual del Departamento de Virología del Instituto de Medicina Tropical "Pedro Kourí". Se realizó RT-RCP con el objetivo posterior de estudiar, mediante secuenciación de ácidos nucleicos, 2 fragmentos del gen gag y 2 del gen env. El ARN viral fue extraído del suero mediante QIAamp Viral RNA Mini Kit (QIAGEN) y el ADN complementario se sintetizó con el empleo de 2 enzimas diferentes: en un primer ensayo la Reverse Transcriptase, AMV (Boeringer-Mannheim) y posteriormente la SensiScript (QIAGEN), a punto de partida de los resultados obtenidos con la AMV. Se utilizaron cebadores random (QIAGEN). Para la amplificación por RCP-semianidada de 2 fragmentos del gen gag y 2 del env se diseñaron 7 juegos de cebadores (tabla) basados en las cepas de referencia de VIH-1 incluidas en la base de datos de Los Álamos (g1up/g1d, g1.1u/g1d, g2.1u/g2d, g2u/g2d, e1u/e2d, e1u/e1d, e2u/e2d).8 Los productos de RCP fueron purificados utilizando MinElute PCR Purification Kit (QIAGEN) y secuenciados automáticamente mediante el sistema de terminadores de cadena fluorescentes (Applied Biosystems Division, Perkin-Elmer). Todas las secuencias fueron comprobadas utilizando el sistema BLAST (Los Álamos).8
Tabla. Secuencia y posición de los cebadores utilizados
| Cebador | Secuencia (5´-3´) | Posición* |
| g1u | ATGGGTGCGAGAGCGTCAGTATTA | 790-813 |
| g1.1u | GGTGCGAGAGCGTCAGTATTA | 793-813 |
| g2.1u | AAAGACACAGCAGGCAGCAG | 1128-1147 |
| g2u | CACCTAGGACTTTGAATGCATG | 1232-1253 |
| g1d | TGGGGTGGCTCCTTCTGATAATGC | 1309-1332 |
| g2d | GGCATGCTGTCATCATTTCTTC | 1819-1840 |
| e1u | CAATTCCCATACATTATTGTGC | 6859-6880 |
| e1d | CAATTAAAACTATGTGTTGTAATTTC | 7332-7357 |
| e2u | GATATGAGGGACAATTGGAGAAGTG | 7644-7668 |
| e2d | GCCTGGAGCTGTTTAATGCCC | 7937-7957 |
* Según cepa HIVHXB2 (F033819)
Con el empleo de la enzima SensiScript se obtuvo amplificación del ARN del VIH-1 en 86,5 % de las muestras estudiadas (64 sueros), comparado con 20 % (15 sueros) al utilizar la enzima AMV-RT (estos sueros también amplificaron con la SensiScript). En 13,5 % de las muestras (10 sueros) no se obtuvo amplificación con ninguna de las 2 enzimas empleadas. Estas muestras que resultaron negativas fueron colectadas entre los años 1989 y 1995. De las secuencias obtenidas a partir de los productos de RCP, 100 % fue útil y homologaron con VIH-1 al ser comparadas con las secuencias de referencia de la base de datos de Los Álamos.
En la figura se muestra el porcentaje de recobrado del material genético según el año de toma de la muestra y el sistema enzimático empleado. Como es de esperar para ambos sistemas, se logra una mayor recuperación de ARN de las muestras que menos tiempo estuvieron conservadas a - 20 °C; aunque con el sistema SensiScript el recobrado es ostensiblemente mayor. Puede observarse que solo mediante el uso de la enzima SensiScript pudo obtenerse amplificación de un grupo de muestras colectadas entre los años 1989 y 1991.
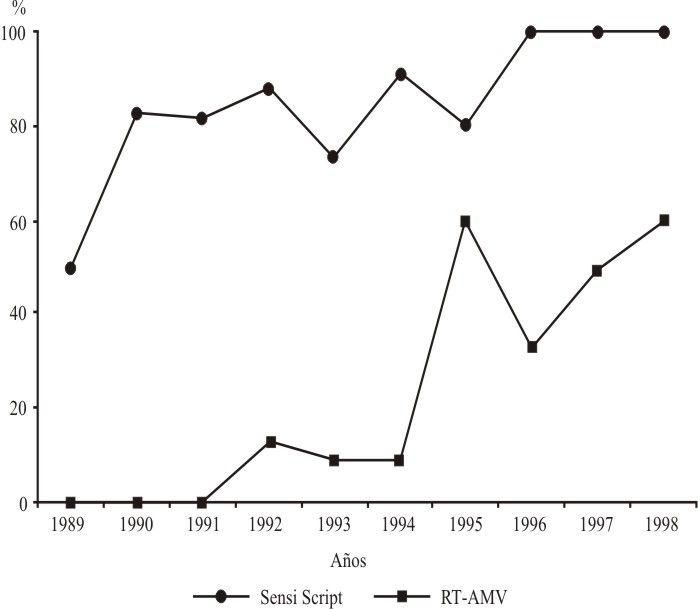
Fig. Porcentaje de recobrado de material genético según el año de toma de muestra y el sistema enzimático empleado.
La enzima SensiScript es una reverso transcriptasa nueva, única, heterodimérica recombinante (expresada en E. coli), diferente de la del virus maloney de la leucemia murina (MMLV) o de la del virus de la mieloblastosis aviar (AMV), y se recomienda para muestras que contengan < 50 ng de ARN. Ha sido utilizada en la técnica de expresión diferencial (differential display) para el estudio de ARN mensajero.9
Al obtenerse solo 20 % de positividad al emplear la enzima AMV y debido a la limitada cantidad de suero con la cual se contaba y la certeza de que las condiciones de almacenamiento no habían sido las idóneas, es que se decide, posteriormente, utilizar la enzima SensiScript; para garantizar un incremento del recobrado de un ARN, que además de degradado en su mayoría, debía estar en muy bajas concentraciones.
Como se ha planteado anteriormente, en este estudio fueron utilizadas muestras de suero para, a partir de estas, extraer material genético. El obtener la secuencia de ácidos nucleicos de las muestras de estos pacientes era de vital importancia y por ser fallecidos no podía contarse con una muestra de plasma fresco y conservado a una temperatura óptima. Un gran número de autores recomienda el uso de plasma libre de células para la obtención de ARN de VIH a utilizar en los estudios moleculares. No obstante, existen reportes que reflejan un coeficiente de variación en la cantidad de copias de ARN/100 uL de muestra de entre 0,09 a 0,8 al comparar el recobrado del plasma con el del suero.7,10 Además, muestras de suero sometidas a ciclos de congelación y descongelación muestran un incremento en el número de copias de ARN (+ 0,073 unidades log).7,11 Por otra parte, la mayoría de las muestras de sangre utilizadas en estudios moleculares de VIH son transportadas en congelación y mantenidas a - 70 °C como plasma libre de células hasta su estudio. Ginocchio7 demostró que la disminución en las cantidades de virus en el plasma al cabo de los meses no fue significativa, pero observó un decrecimiento de 0,3 unidades log al realizar la comparación con el suero.
Con estos resultados se puede concluir que la enzima Sensiscript constituye una herramienta valiosa en el recobrado de ARN de VIH-1, para el análisis genotípico a partir de sueros conservados a temperatura subóptima; esto resulta de vital importancia, no solo para el VIH, sino para otros virus ARN y da la posibilidad de utilizar muestras de suero mantenidas en los laboratorios, bajo estas condiciones de temperatura, hasta por una década.
Summary
The efficiency of SensiScript reverse transciptase to obtain useful genetic material in the sequencing of the nucleic acid from HIV-1, starting from sera collected between 1989 and 1998 and kept at suboptimal temperatures, was proved. On using the SensiScript enzyme it was obtained an amplification of the RNA of the HIV-1 in 86.5 % of the studied samples, compared with 20 % on using the AMV-RT enzyme . No amplification was obtained in 13.5 % of the studied samples with any of the 2 enzymes used.
.
Subject headings: REVERSE TRANSCRIPTASE POLYMERASE CHAIN REACTION/ methods; HIV-1; SEQUENCE ANALYSIS, RNA; ENZYME TESTS.
Referencias bibliográficas
- Niubo J, Li W, Henry K, Erice A. Recovery and Analysis of Human Immunodeficiency Virus Type 1 (HIV) RNA Sequences from Plasma Samples with Low HIV RNA Levels. J Clin Microb 2000;38:309-12.
- Natarajan V, Bosche M, Metcalf JA, Ward DJ, Lane HC, Kovacs JA. HIV-1 replication in patients with undetectable plasma virus receiving HAART. Lancet 1998;353:119-20.
- Zhu T, Korber BT, Nahmias AJ, Hooper E, Sharp PM, Ho DD. An African HIV-1 sequence from 1959 and implications for the origin of the epidemic. Nature 1998;391:594-7.
- Rodrigo AG. HIV evolutionary genetics. Proc Natl Acad Sci 1999; 96:10559-61.
- Verhofstede C, Fransen K, Marissen D, Verhelst R, van der Groen G, Lauwers S, et al. Isolation of HIV-1 RNA from plasma: evaluation of eight different extraction methods. J Virol Methods 1996;60:155-9.
- Fransen K, Mortier D, Heyndrickx L, Verhofstede C, Janssens W, van der Groen G. Isolation of HIV-1 RNA from plasma: evaluation of seven different methods for extraction (part two). J Virol Methods 1998;76:153-7.
- Ginocchio CC, Wang X-P, Kaplan MH, Mulligan G, Witt D, Romano JW, et al. Effects of Specimen Collection, Processing, and Storage Conditions on Stability of Human Immunodeficiency Virus Type 1 RNA Levels in Plasma. J Clin Microbiol 35:2886-93.
- Human Retroviruses and AIDS 2000: A Compilation and Analysis of Nucleic Acid and Amino Acid Sequences. In: Kuiken CL, Foley B, Hahn B, Korber B, McCutchan F, Marx PA, Mellors JW, Mullins JI, Sodroski J, Wolinksy S, eds. Theoretical Biology and Biophysics Group, Los Alamos National Laboratory: Los Alamos, NM.
- Bosch I, Melichar H, Pardee AB. Identification of differentially expressed genes from limited amounts of RNA. Nucleic Acids Research 2000;28:E27.
- Holodniy M, Mole L, Yen-Lieberman B, Margolis D, Starkey C, Carroll R, Spahlinger T, Todd J, Jackson JB. Comparative stabilities of quantitative human immunodeficiency virus RNA in plasma samples collected in VACUTAINER CPT, VACUTAINER PPT, and standard VACUTAINER tubes. J Clin Microbiol 1995;33:1562-6.
- Moudgil T, Daar ES. Infectious decay of human immunodeficiency virus type 1 in plasma. J Infect Dis 1993;167:210-2.
Recibido: 4 de abril de 2003. Aprobado: 8 de mayo de 2003.
Dra. Sonia Resik Aguirre. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana, Cuba. Correo electrónico: sresik@ipk.sld.cu
1 Máster en Virología. Especialista de II Grado en Microbiología. Investigadora Auxiliar. Profesor Instructor. Instituto de Medicina Tropical "Pedro Kourí" (IPK).
2 Investigadora. Center for AIDS Research. Universidad de Chapel Hill, Carolina del Norte, EE. UU.
3 Especialista de I Grado en Microbiología (IPK).
4 Máster en Virología. Especialista de II Grado en Microbiología. Investigadora Auxiliar, Profesor Instructor (IPK).
5 Director. Center for AIDS Research, Universidad de Chapel Hill, Carolina del Norte, EE. UU.














