Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.56 n.2 Ciudad de la Habana Mayo-ago. 2004
Instituto de Medicina Tropical Pedro Kourí
Uso del análisis de restricción en el control de calidad de cepa del biolarvicida cubano BACTIVEC
Lic. Odelaisy Suárez-Moreno,1 Dra. Grisel Montero Lagos,2 Dr. Jorge Luis Maestre Mesa,3 Téc. Manuel Díaz Pérez4 y Est. Odisney Lugo Suárez5
Resumen
Se mostró la caracterización genómica de la cepa utilizada en la producción del biolarvicida Bactivec. Se compararon los patrones de digestión de la cepa de referencia 266 de Bacillus thuringiensis de la colección del Instituto Pushkin del antiguo Leningrado y 2 cepas aisladas de 2 lotes del biolarvicida. Las digestiones del ADN cromosomal se realizaron con las enzimas HindIII y EcoRI. En todos los casos se observó el mismo patrón de restricción, lo que muestra la estabilidad genética del ingrediente activo del biolarvicida Bactivec.
Palabras clave: Bacillus thuringiensis, genética, vectores de enfermedades, control de enfermedades transmisibles; control de calidad, biología molecular.
Es indiscutible que para el control de muchas de las enfermedades tropicales, el factor primordial es la lucha contra los mosquitos, vectores de los agentes causales de estas (Progress report 1997). En los últimos años se ha intensificado el desarrollo de los biocontroles en la lucha contra estos vectores. Entre estos productos los biolarvicidas sobre la base de Bacillus thuringiensis H-14 (Bt H-14) han tenido gran aceptación con un impacto considerable en el control de enfermedades como la malaria, la fiebre amarilla, el dengue y la filariasis, que ocurren en el trópico donde las condiciones ambientales favorecen al vector. Muchas son las investigaciones que confirman la eficacia de los tratamientos con Bacillus thuringiensis en los estudios de campo realizados; de hecho diferentes formulaciones de Bt H-14 han sido evaluadas en India, Tailandia, Cuba, Filipinas y China (TDR, 1995).
En Cuba, la producción y comercialización del biolarvicida Bactivet, la realiza LABIOFAM S.A. siendo objeto de interés comercial en muchos países. Para su introducción en nuevos mercados se hace necesario presentar un aval de la pureza y estabilidad de la cepa, ingrediente activo de la formulación; para ello se emplean diferentes técnicas bioquímicas y bacteriológicas y también pueden utilizarse técnicas que brindan información sobre el comportamiento genético, como es el caso del análisis con enzimas de restricción del ADN cromosomal.
El objetivo del presente trabajo es demostrar que la cepa empleada en el producto cubano, presenta idéntico patrón genético al de la cepa de referencia 266 de Bacillus thuringiensis del Instituto Pushkin del antiguo Leningrado y con ello aportar una herramienta más para el control de calidad del producto.
Métodos
Se estudiaron 3 cepas 266 de Bacillus thuringiensis H-14. Una es la cepa de referencia de la colección del Instituto Pushkin del antiguo Leningrado y las otras 2 cepas fueron aisladas de diferentes lotes del biolarvicida que se elaboraron en los años 2001 y 2002. Para facilitar el trabajo se codificaron las cepas de la forma siguiente: Cepa No. 1 es la cepa 266 de referencia, la cepa No. 2 es la cepa 266 extraída directamente del producto Lote del 2001 y la No. 3 corresponde a la cepa 266 extraída directamente del producto Lote del 2002.
Se cultivaron las cepas en caldo soja/triptona por 18 h a 30 ºC, la masa bacilar fue concentrada por centrifugación y se procedió a la extracción del ADN cromosomal según lo recomendado en el manual de laboratorio Molecular Cloning de Sambrook y otros.
El ADN cromosomal de cada cepa fue digerido con las enzimas HindIII y EcoRI (Promega), para ello se realizó el ensayo siguiendo las recomendaciones del proveedor para cada enzima. La electroforesis del producto digerido se llevó a cabo en gel de agarosa a 0,8 % a 30 V durante 16 h, al término de las cuales se fotografió el gel que contenía los patrones de digestión obtenidos.1
Resultados y comentarios
Las técnicas de análisis de restricción del ADN cromosomal junto con otras técnicas de biología molecular, han sido ampliamente utilizadas en la caracterización y taxonomía de este bacilo (Fergus, 1994).2
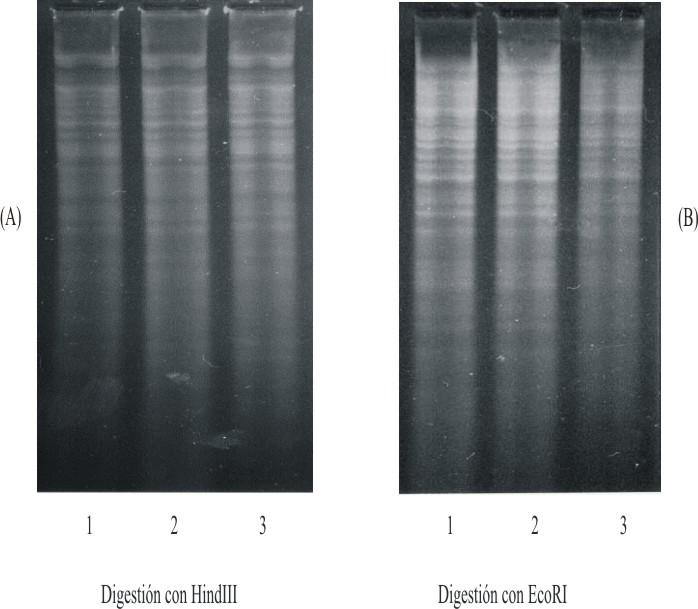
Fig.(A) Digestiones obtenidas con la enzima Hind III, el carril 1 corresponde a la cepa de referencia liofilizada; el carril 2 a la muestra tomada del lote de 2001 y el 3 a la muestra correspondiente del lote de 2002. (B) Digestiones obtenidas con la enzima EcoRI, el carril 1 corresponde a la cepa de referencia liofilizada; el carril 2 a la muestra tomada del lote de 2001 y el 3 a la muestra correspondiente del lote de 2002
Si los organismos son genéticamente iguales, el análisis de restricción permite obtener patrones idénticos de fragmentos polimórficos del ADN, y para incrementar el rigor de discriminación, se recomienda la digestión con una segunda enzima, lo que ayuda a confirmar la identidad de sus patrones (Suárez OM, Maestre JL, Ocampo A. Folleto docente. Introducción a la Biología Molecular. Subdirección Docente. Instituto Pedro Kourí, La Habana, Cuba. 1998). Estudios en los que se ha utilizado la técnica de análisis de restricción con propósitos similares a este son los realizados por Kwang-Bo Joung en el 2000.3-5
Como puede observarse en la figura, con el empleo de las enzimas de restricción HindIII y EcoRI se logró obtener la digestión del ADN de las cepas estudiadas, con un único patrón para cada enzima. Estos resultados indican que las huellas genéticas de las cepas aisladas del producto se han mantenido y por otro lado estos patrones pueden ser utilizados como punto de comparación para estudios posteriores.
Summary
The genomic characterization of the strain used in manufacturing biolarvicide BACTIVET was shown in this paper. Digestion patterns of Bacillus thuringiensis reference strain 266 from the collection of Pushkin Institute located in former Leningrad and of two strains isolated from 2 batches of biolarvicide were compared. Chromosomal DNA digestion was performed by enzymes Hindlll and EcoRI. The same restriction pattern was observed in all cases, which is indicative of genetic stability of the active ingredient of biolarvicide BACTIVET.
Key words: Bacillus thuringiensis, geneics, disease vectors, communicable disease control, molecular biology.
Referencias bibliográficas
- CTD. Division of control of tropical disease. Progress report 1997. 184-5.
- Fergus GP, Denise AK, Yoko BR, Vanderlai PC. Characterization of Bacillus thuringiensis and related bacteria by ribosomal RNA gene restriction fragment lingth polymorphisms. Microbiology 1994;140:1015-22.
- Kwang-Bo J. Test d'ADN pour la classification des variétés de Bacillus thuringiensis. Artículo del año 2000. Revisado el 1 de julio de 2003. http//res2.agr.ca/stjeanl/publicatiom/resume9900/cote1_f.htm. .
- Sambrook J, Frisch EF, Maniatis T. Molecular Cloning . A laboratory manual. 2.ed. 1990.
- TDR. Tropical disease research. Progress 1975-94 Highlights 1993-94. 1995. OMS 1995;147-153.
Recibido: 25 de agosto de 2003. Aprobado: 24 de noviembre de 2003.
Lic. Odelaisy Suárez- Moreno. Instituto de Medicina Tropical Pedro Kourí. Apartado 601, Marianao 13, Ciudad de La Habana, Cuba. Teléf: 2020426. Correo electrónico: odelaisy@ipk.sld.cu
1 Máster en Ciencias. Profesora e Investigadora Auxiliar.
2 Doctora en Ciencias. Investigadora Titular.
3 Especialista de II Grado en Microbiología. Profesor e Investigador Auxiliar.
4 Técnico A.
5 Estudiante de Medicina.