My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Medicina Tropical
Print version ISSN 0375-0760On-line version ISSN 1561-3054
Rev Cubana Med Trop vol.56 no.3 Ciudad de la Habana Sept.-Dec. 2004
Instituto de Medicina Tropical Pedro Kourí
Perfil plasmídico y resistencia antimicrobiana en cepas de Shigella aisladas en Cuba
Dra. Margarita Ramírez,1 Dra. Neysi Valdés,2 Lic. Laura Bravo3 Téc. Anabel Fernández4 y Téc. Nelsydeismi Castañeda5
Resumen
Se investigó un total de 240 cepas de Shigella, procedentes de los Centros Provinciales de Higiene y Epidemiología, durante el período de enero a diciembre de 2002. Estas especies constituyen los microorganismos aislados con mayor frecuencia de infecciones diarreicas agudas en los países en vías de desarrollo y en Cuba. Mediante el estudio de diferentes marcadores fenotípicos y genotípicos como: serotipaje, estudios de resistencia a drogas antimicrobianas y perfil plasmídico, se investigó la relación epidemiológica de las cepas en estudio. Los serogrupos predominantes fueron: S. flexneri 142 (59 %) y S. sonnei 76 cepas (32 %). El comportamiento de la susceptibilidad antimicrobiana mostró 79,2 % de cepas resistentes. Al analizar el comportamiento de S. flexneri según la región de origen, el perfil de resistencia y el perfil plasmídico se realizaron 2 agrupamientos y en el serogrupo S. sonnei se encontraron 5 agrupamientos. Los resultados obtenidos demuestran la heterogeneidad genética en las cepas de Shigella que circulan en el país.
Palabras clave: Shigella, perfil de resistencia, perfil plasmídico.
La cifra anual de infecciones causadas por el género Shigella en todo el mundo es de 164 700 000, de los cuales 163 200 000 se producen en los países en desarrollo y 1 500 000 de casos en los países industrializados. En total, 69 % de todas las infecciones y 61 % de todas las defunciones atribuidas a shigelosis afectan a menores de 5 años.1,2
En los últimos 50 años, Shigella ha demostrado una extraordinaria capacidad para adquirir resistencia, codificada por plásmidos, a los antibióticos que antes constituían el tratamiento de primera línea. Al principio las sulfamidas, la tetraciclina, la ampicilina y el trimetoprim/sulfametoxazol, eran fármacos muy eficaces, pero esto ha ido disminuyendo ante la aparición de cepas multirresistentes.3,4
El análisis de los patrones plasmídicos ha sido útil para la caracterización de cepas de Shigella sp. que albergan plásmidos; la identificación de estos es útil mientras exista una cantidad mínima de presión selectiva en el medio ambiente, la que puede provocar una ganancia; una pérdida de estos hace que sean inestables, no obstante, constituye un potente método en el estudio de marcadores epidemiológicos.4,5
En los estudios sistemáticos de vigilancia de la susceptibilidad a las drogas antimicrobianas realizados en el Laboratorio Nacional de Referencia de Enfermedades Diarreicas Agudas (LNR EDA) del Instituto de Medicina Tropical Pedro Kourí (IPK), durante la última década, se ha comprobado el incremento de la resistencia en las cepas de Shigella, sobre todo a drogas como ampicilina, trimetoprim/sulfametoxazol, tetraciclina, cloranfenicol y han comenzado a surgir cepas resistentes al ácido nalidíxico.6,7
En los últimos años se ha adquirido cada vez más conciencia de la enorme repercusión de Shigella como patógeno intestinal y de las devastadoras consecuencias que podrían derivarse de un ritmo de aparición de cepas multirresistentes, que desbordase la disponibilidad de tratamientos antibióticos accesibles y eficaces.8 Por este motivo se decidió investigar la susceptibilidad antimicrobiana y el perfil plasmídico en un grupo de cepas de Shigella aisladas en Cuba, para conocer el comportamiento epidemiológico de este microorganismo en el medio cubano.
Métodos
Se seleccionaron mediante un muestreo simple aleatorio 240 cepas de Shigella enviadas al LNR –EDA del IPK, procedentes de los Centros Provinciales de Higiene y Epidemiología del país, durante el período enero-diciembre de 2002. Estas cepas fueron aisladas de heces diarreicas de niños menores de 5 años. Para el análisis de la situación en Cuba se agruparon las provincias en 3 regiones: Occidente, Centro y Oriente; 82 de las cepas provenían de la región Occidental, 49 fueron aisladas en el centro del país y 109 de la región oriental.
Serotipaje
Para realizar el serotipaje las cepas se cultivaron en medio de agar soya trypticasa y las reacciones serológicas se determinaron por la técnica de aglutinación en láminas, utilizándose antisueros de la casa Comercial Murex Diagnostic.9
Prueba de susceptibilidad antimicrobiana
La determinación de la susceptibilidad antimicrobiana se realizó por el método de difusión en discos en agar (Kirby-Bauer),10 empleando 11 drogas antimicrobianas (recomendadas por el Comité Nacional de Estándares de Laboratorio Clínico [NCCLS] para la familia Enterobacteriaceae:11 ampicilina AM [10 µg], ampicilina/sublactam, SAM [30 µg]; amoxicilina/ácido clavulánico, AML [30 µg]; ceftriaxona, CRO [30 µg]; aztreonam, ATM [30 µg]; gentamicina, CN [10 µg]; ciprofloxacina CIP, [5 µg]; tetraciclina, TE [30 µg]; ácido nalidíxico, NA [30 µg]; trimetoprim-sulfametoxazol, SXT [25 µg]; y cloranfenicol, C [30 µg]). Se utilizaron como cepas controles: Escherichia coli ATCC 25922, Escherichia coli ATCC 35218, Pseudomonas aeruginosa ATCC 27853 y Staphylococus aureus ATCC 25923.
Extracción del ADN plasmídico
Se seleccionaron para el análisis de los perfiles plasmídico, aquellas cepas que mostraron resistencia a 3 antimicrobianos o más (multidrogorresistentes) y por un muestreo simple aleatorio, se escogieron 30 cepas de S. flexneri y 20 cepas de S. sonnei. No se escogieron cepas de otros serotipos por su pobre representación estadística en la muestra estudiada. Se utilizó el método de lisis alcalina de Kado y Liu.12 Los plásmidos obtenidos fueron visualizados y analizados en electroforesis en gel de agarosa 0,8 %. El peso molecular de los plásmidos fue determinado por el método de la curva logarítmica de regresión lineal.13 El poder de discriminación para el análisis del perfil plasmídico fue determinado por el índice de diversidad de Simpson (D).13
Procesamiento estadístico
El análisis estadístico se realizó al aplicar la prueba de chi cuadrado (X²), las pruebas F de Fisher y de Duncan a 5 %, las cuales permitieron establecer la asociación entre las variables analizadas, las diferencias entre las proporciones obtenidas así como la relación entre estas.
Resultados
Distribución de los serogrupos de Shigella
De las 240 cepas analizadas, la mayoría correspondió a S. flexneri (142 cepas), para 59 %, difiriendo significativamente (p< 0,001) este porcentaje con respecto a los mostrados por los otros serogrupos encontrados: S. sonnei (76 cepas) fue el siguiente en frecuencia con 32 % y difirió significativamente (p< 0,001) de los demás serogrupos, le siguieron S. dysenteriae (12 cepas), con 5 % y S. boydii (10 cepas), con 4 %.
Comportamiento de las infecciones por Shigella en función del serogrupo y resistencia antimicrobiana según las diferentes regiones del país
Como se aprecia en la tabla 1, se obtuvieron esos resultados al analizar la distribución de los serogrupos de Shigella por regiones del país; se detectaron diferencias altamente significativas (p< 0,001) al aplicar las pruebas de Fisher y las pruebas de Duncan a 5 %, en las proporciones presentadas por los serogrupos A (Shigella dysenteriae), B (Shigella flexneri), C ( Shigella boydii) y D (Shigella sonnei) en las diferentes regiones del país; se mostró que el serogrupo A fue más frecuente en el Centro y el serogrupo D en las regiones de Occidente y Oriente. El serogrupo B no presentó diferencias estadísticamente significativas (ns) en cuanto a su distribución por las regiones del país.
Tabla 1: Distribución de los serogrupos de Shigella por regiones del país
| Serogrupos | Regiones | F | ||
| Occidente % | Centro % | Oriente % | ||
| A | 1 (1,3)b | 9 (18,4)a | 2 (1,9)b | 11,60 (p< 0,001) |
| B | 46 (56,1)a | 31(63,2)a | 65 (59,6)a | 0,34 ns |
| C | 0 (0)b | 5 (10,2)a | 5 (4,5)ab | 4,04 (p< 0,05) |
| D | 35 (42,6)a | 4 (8,2)b | 37 (33,9)a | 8,68 (p< 0,001) |
Nota: Proporciones con letras comunes no difieren significativamente según prueba de Fisher.
Análisis de la susceptibilidad antimicrobiana.
Los resultados del comportamiento de la resistencia antimicrobiana de las cepas estudiadas, distribuidas por serogrupos y por regiones del país, se muestran en la tabla 2. Las cepas correspondientes al serogrupo B, mostraron altos porcentajes de resistencia específicamente en la región oriental (61 %), al igual que las del serogrupo D, 32 % en Occidente y 27 % en Oriente. La resistencia de las cepas de los serogrupos A y C no mostró cifras significativas.
Tabla 2. Comportamiento de la resistencia antimicrobiana de los serogrupos de Shigella según las regiones del país
| Serogrupo | Occidente | Centro | Oriente | F |
| Resistentes % | Resistentes % | Resistentes % | ||
| A | 0 (0,0)b | 3 (6,1)a | 1 (0,92)b | 3,85 (p< 0,05) |
| B | 30 (36,6)b | 26 (53,1)b | 61 (55.9)a | 3,75 (p< 0,05) |
| C | 0 (0,0)a | 3 (6,1)a | 5 (4,6)a | 2,27 ns |
| D | 32 (39,02)a | 2 (4,08)b | 27 (24,8)a | 9,90 (p< 0,001) |
Nota: proporciones con letras comunes no difieren significativamente según prueba de Fisher
Fenotipos de resistencia
Se detectó un total de 28 patrones de resistencia en las 190 cepas de Shigella multirresistentes. En la tabla 3 se muestran los 9 patrones que se encontraron en mayor proporción en estas cepas.
Tabla 3: Patrones de resistencia antimicrobiana más frecuentes mostrados por las cepas de Shigella multirresistentes encontradas en nuestro estudio.
| Patrones de resistencia | No de cepas | % |
| S, TE, SXT | 71 | 37,4 |
| AM, SAM, AML, S, TE, SXT, C | 24 | 12,7 |
| AM, SAM, AML, S, TE, SXT | 18 | 9,5 |
| S, TE, NA, SXT | 13 | 6,9 |
| AML, S, TE, SXT | 8 | 4,3 |
| AM, SAM, AML, TE, SXT | 7 | 3,7 |
| AM, SAM, AML, TE, SXT, C | 6 | 3,2 |
| AM, AML, S, TE, SXT, C | 5 | 2,6 |
| AM, AML, S, TE, SXT | 5 | 2,6 |
AM: ampicilina, TE: tetraxiclina, SXT: trimetoprim-sulfametoxazol, SAM: ampiclina-sulbactam, S: estreptomicina, C: cloranfenicol , NA: ácido nalidíxico, AML: amoxicilia-ácido clavulánico.
Perfil plasmídico
En 100 % de las cepas analizadas de Sh. flexneri y Sh. sonnei se detectaron plásmidos, los perfiles plasmídicos presentados en estas especies fueron excluyentes y se asociaron a 17 fenotipos de resistencia.
Análisis del perfil plasmídico de S. flexneri
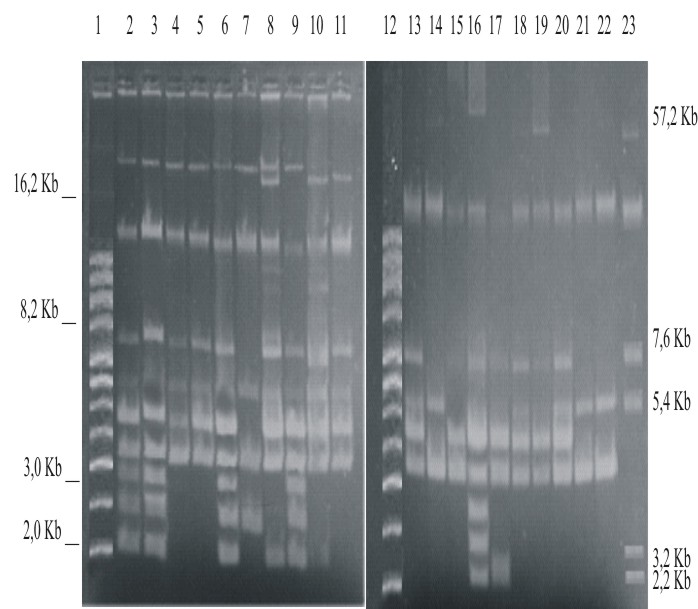
En las 30 cepas de S. flexneri estudiadas, fueron obtenidos 6 perfiles plasmídicos diferentes (fig. 1). Estos perfiles contenían entre 2 y 7 plásmidos con tallas en un rango de 2 a 16 Kb. Se encontró una alta variabilidad del perfil plasmídico (1-3 plásmidos) en este rango. Los perfiles plasmídicos en su mayoría estuvieron presentes en más de una cepa excepto el perfil B III, representado en una sola cepa. El índice de diversidad de Simpson (ID) para esta técnica fue de 0,85.
Líneas 1 y 12, marcador de peso molecular (supercoiled DNA ladder, GIBCO-BRL, invitrogen). Líneas 2, 3, 6, 9 y 16, perfil plasmídico B I. Líneas 4, 5, 10, 11, 13 y 20, perfil plasmídico B II. Línea 7, perfil plasmídico B III. Líneas 8 y 17, perfil plasmídico B IV. Líneas 13, 21 y 22, perfil plasmídico B V. Líneas 15, 18 y 19, perfil plasmídico B VI, línea 23 cepa control Eschericha coli V157 con plásmidos con rango de PM entre 2,1 a 57,2 Kb.
Fig. 1. Perfiles plasmídicos de cepas de S. flexneri.
Al realizar el análisis de los perfiles plasmídicos (tabla 4), se observó que, las cepas con los perfiles B I, II y IV procedían de las 3 regiones del país, los perfiles B V y VI, fueron encontrados en cepas procedentes de Centro y Oriente. Al relacionar los fenotipos de resistencia mostrados por las cepas se encontró que cada perfil plasmídico tenía entre 2 y 5 patrones de resistencia diferentes (excepto en el perfil III) y que estos no eran excluyentes.
Tabla 4. Perfiles plasmídicos de las 30 cepas de S. flexneri analizadas y su relación con los patrones de resistencia y la región de origen de las cepas
| Perfil plasmídico | Total de cepas | Cepas | Región de origen | Fenotipo de resistencia |
| I | 6 | 1 | A | II |
| 14 | B | I | ||
| 5, 8 | A | IX | ||
| 2, 25 | A / C | XVI | ||
| II | 7 | 10 | B | VI |
| 4, 9 | A / C | IX | ||
| 3, 11 | A / B | X | ||
| 18, 22 | C | XII | ||
| III | 1 | 6 | A | X |
| IV | 3 | 7 | A | XIV |
| 15, 27 | B / C | X | ||
| V | 7 | 23 | C | VII |
| 12, 19 | B / C | IX | ||
| 20, 28-30 | C | X | ||
| VI | 6 | 13 | C | V |
| 16, 17 | B / C | X | ||
| 26 | C | XI | ||
| 21 | C | XII | ||
| 24 | C | XIII |
Análisis del perfil plasmídico de S. sonnei
Fueron detectados 8 perfiles plasmídicos diferentes (fig. 2) denominándose como D-I, D-II, D-III, D-IV, D-V, D-VI, D-VII y D-VIII, cada uno albergó entre 2 y 8 plásmidos con tallas en un rango de 2 a 16 Kb. Se encontró una alta variabilidad del perfil plasmídico (de 1 a 3 plásmidos) en este rango. Los perfiles plasmídicos estuvieron presentes en más de una cepa, exceptuando los patrones D-III, VI, VII y VIII. El índice de diversidad de Simpson (ID) para esta técnica fue de 0,86.
Líneas 1 y 13, marcador de peso molecular (supercoiled DNA ladder, GIBCO-BRL, invitrogen). Líneas 2, 3, 10, 11, 14, 16 y 18, perfil plasmídico D I. Líneas 4 y 5, perfil plasmídico D II. Línea 6, perfil plasmídico D III. Líneas 9, 21,22,23, perfil plasmídico D IV. Líneas 8 y 20, perfil plasmídico D V. Línea 15, perfil plasmídico D VI. Línea 17, perfil plasmídico D VII y Línea 19, perfil plasmídico D VIII. Línea 12 cepa control Eschericha coli V157 con plásmidos con rango de PM entre 2,1 a 57,2 Kb.
Fig. 2. Perfiles plasmídicos de cepas de S. sonnei.
Al realizar el análisis de los perfiles plasmídicos (tabla 5), se observó que en el caso de los perfiles D I, D IV y D V, las cepas que los presentaron procedían del Occidente y el Oriente del país, los perfiles DII y DIII estuvieron representados por cepas de la región occidental, mientras que los perfiles DVI, DVII y DVIII, se encontraron solamente en el Oriente del país. Al relacionar los fenotipos de resistencia mostrados por las cepas se encontró que con excepción del perfil DVI, que tiene 2 patrones de resistencia, el resto de los perfiles plasmídicos presentaron un solo patrón de resistencia por perfil. Basándose en el perfil plasmídico, en el patrón de resistencia y en la región de origen de las cepas se realizaron 5 agrupamientos.
Tabla 5. Perfiles plasmídicos de las 20 cepas de S. sonnei analizadas y su relación con los patrones de resistencia y la región de origen de las cepas
| Perfil plasmídico | Total de cepas | Cepas | Región de origen | Fenotipos de resistencia |
| I | 7 | 31, 32, 39, 40, 41, 43, 45 | A (4) / C (3) | XVI |
| II | 2 | 33, 34 | A | III |
| III | 1 | 35 | A | XV |
| IV | 5 | 36, 38 | A | XVI |
| 48, 49, 50 | C | XV | ||
| V | 2 | 37, 47 | A / C | XVI |
| VI | 1 | 42 | C | XVI |
| VII | 1 | 44 | C | XV |
| VIII | 1 | 46 | C | XVI |
Discusión
La estimación exacta del comportamiento de las infecciones por Shigella, en países desarrollados o en vías de desarrollo, tiene 2 importantes dimensiones: una dimensión clínica, que proviene de la magnitud de la morbilidad y mortalidad atribuibles a este patógeno y una dimensión epidemiológica condicionada por la distribución de los serogrupos y serotipos en diferentes áreas geográficas.3 El principal serogrupo de Shigella que circula en una comunidad, parece estar relacionado con el nivel socioeconómico. La mayoría de los aislamientos (60 %) de Shigella en los países en vías de desarrollo corresponde a S. flexneri, S. sonnei.14 Los serogupos encontrados en este estudio coinciden con lo publicado en la literatura para los países de América Latina, Asia y África.14
Entre los marcadores fenotípicos, el serotipaje es uno de los métodos clásicos de tipificación de cepas. Tiene gran importancia desde el punto de vista epidemiológico para los estudios de vigilancia porque permite determinar la prevalencia de serovariedades en diferentes zonas geográficas, ya sea en estudios de brotes o de casos esporádicos.15
En cuanto a los porcentajes de resistencia mostrados por los serogrupos de Shigella en las diferentes regiones del país, de acuerdo con su distribución por regiones, la prevalencia de distintos serogrupos, pudiera estar determinada por factores sociales, ambientales y microbiológicos. Dentro de estos aspectos se incluyen: aumento de las poblaciones de hospederos susceptibles, viajes internacionales, el turismo y el comercio, entre otros.15
La resistencia bacteriana a los antimicrobianos continúa siendo un serio y creciente problema en todo el mundo, especialmente para los países en vías de desarrollo donde muchos factores contribuyen a engendrar, desarrollar y extender esta situación. Los datos del comportamiento de la susceptibilidad a los antimicrobianos, desafortunadamente son escasos y no todos los países pueden monitorear la resistencia.14,16
Hoy día, el principal problema asociado con la shigelosis es indudablemente su tratamiento, esta es una de las infecciones entéricas donde el uso de antimicrobianos está ampliamente recomendado y aceptado, teniendo en cuenta que la terapia antimicrobiana adecuada, acorta la duración y severidad del cuadro diarreico, reduce el tiempo de transmisibilidad del agente infeccioso (excreción) y también reduce las complicaciones potenciales, sobre todo en niños y ancianos.8, 9
De acuerdo con los resultados de este trabajo, un alto porcentaje de las cepas analizadas fueron multirresistentes, lo que coincide con estudios realizados en Cuba, durante la última década, por Ramírez y otros, donde aproximadamente 70 % de las cepas analizadas fueron resistentes a los antimicrobianos de primera línea.6,7 Es importante destacar que la ínfima resistencia a quinolonas y cefalosporinas, puede ser debido a que son antibióticos de nueva generación, que no han tenido un uso indiscriminado por la población cubana.6,7
Dentro de las cefalosporinas, la ceftriaxona constituye un recurso eficaz, en la actualidad su uso en el tratamiento de la shigelosis multirresistente o complicada está muy difundido.8 Los resultados de este estudio se corresponden con lo reportado por autores de diferentes países como Brasil, Bolivia, Argentina, Colombia y Ecuador (Foodborne and Diarrheal Diseases Branch.Shigella surveillance: annual tabulation summaries, 1993-1995 and 1996. Atlanta, Georgia: US Department of Health and Human Services, ,Public Health Service, Centers for Disease Control, National Center for Infectious Diseases, Division of Bacterial and Mycotic Diseases, Foodborne and Diarrheal Diseases Branch, 1996 and 1997, respectively).17,18
Altos porcentajes de resistencia de S. sonnei a agentes antimicrobianos de primera línea como ácido nalidíxico, trimetoprim-sulfametoxazol, tetraciclina y ampicilina, han sido reportados en el mundo. En América, se destacan estudios realizados en países como EE. UU., Argentina y Chile que informan índices elevados de resistencia frente a estos antimicrobianos.15,17
Los patrones de resistencia detectados en este estudio en las cepas de distintos serogrupos se corresponden con el comportamiento observado por otros autores como Lima y otros, Prado y otros, Suárez y otros en investigaciones realizadas en Brasil, Chile y Argentina, respectivamente.17,18
Por causa de que los patrones de resistencia antimicrobiana cambian constantemente, es importante mantener un monitoreo continuo de la susceptibilidad antimicrobiana de las cepas de Shigella, para poder guiar de manera adecuada el tratamiento empírico de los cuadros diarreicos en cada región. La identificación de nuevos o inusuales patrones de resistencia antimicrobiana de cepas aisladas de muchos pacientes es a veces el primer indicio de un brote epidémico.2,14,15
El análisis de las cepas de S. flexneri y S. sonnei incluidas en este estudio, estuvo basado en los perfiles plasmídicos, el patrón de resistencia antimicrobiana y la región de origen de las cepas. La determinación del perfil plasmídico ha sido el método más ampliamente utilizado para la investigación epidemiológica de cepas de casos esporádicos y de brotes, producidos por Shigella sp. Por otra parte este resulta un método económico y rápido, pero, el hecho de que muchos plásmidos son inestables y que pueden perderse o ganarse por la presión selectiva de los antibióticos, puede ser una limitante para el uso de esta técnica. Shigella sp. usualmente alberga una heterogénea población de plásmidos (entre 2 a 10).17-19
Los resultados de este trabajo concuerdan con los obtenidos por Vila J y otros en 199916 en un estudio de caracterización y tipaje de cepas de Shigella sp., donde se estudiaron 78 cepas de S. flexneri y 4 cepas de S. sonnei, encontrándose 8 y 2 perfiles plasmídicos respectivamente y sobre la base de la susceptibilidad antimicrobiana se definieron 6 fenotipos de resistencia.16
En los perfiles plasmídicos detectados en S. flexneri se comprobó una gran variabilidad en los patrones de resistencia asociados a estos, así como poca relación con la región de origen de las cepas. Resultados que concuerdan con los obtenidos por Cabrera y otros en Cuba en el año 2002, donde se estudiaron 20 cepas de S. flexneri, procedentes de 9 provincias del país, encontrándose 11 perfiles plasmídicos diferentes que no guardaban relación con los patrones de resistencia asociados.14 Estudios similares se reportan por Lima y otros en Fortaleza, Brasil, los cuales realizaron un estudio de perfil plasmídico, demostrando la gran variedad de perfiles plasmídicos y patrones de resistencia existentes en el área. Otros estudios realizados por Tornes y otros en 1997 reportan analizando 168 casos de shigelosis, que 70 cepas de Shigella sonnei fueron ubicadas dentro de 7 perfiles diferentes.17 En ninguno de los estudios antes mencionados se encontró asociación de los perfiles plasmídicos con los perfiles de resistencia. Lima y otros, en 1997, encontraron que la mayoría de las cepas tenían idéntico o casi idéntico perfil plasmídico, y ponen de manifiesto que Shigella sonnei, con igual perfil plasmídico puede ser encontrada dentro de una amplia distribución geográfica18 Estos resultados concuerdan con los obtenidos aquí porque algunos de los perfiles detectados para Shigella sonnei, estuvieron distribuidos por las regiones de Occidente y Oriente.
La diversidad de fenotipos de resistencia mostrados por las cepas pudiera deberse a que la resistencia esté siendo mediada por plásmidos y además por integrones o transposones que estén incorporados al cromosoma bacteriano y que confieren también resistencia a múltiples antimicrobianos.19
Summary
240 Shigella strains from the Provincial Centers of Hygine and Epidemiology were investigated from Janaury to December, 2002. These species are the isolated microorganisms with the highest frequency of acute diarrheal infections in the developing countries and in Cuba. By the study of different phenotypic and genotypic markers as serotyping, studies of resistance to antimicrobial drugs and plasmidic profile, the epidemiological relation of the strains under study was analyzed. The predominating serogroups were: 142 S. flexneri (59 %) and 76 S. sonnei strains (32 %). The antimicrobial susceptibility behavior showed 79.2 % of resistant strains. The resistance phenotypes most frequently found were: streptomycin, trimethropim-sulfamethoxazole: 71 (37.4 %); ampicillin, ampicillin-sulbactam, tetracycline, trimethroprim-sulfametoxazole, cloramphenicol: 24 (12.7 %); ampicillin, ampicillin-sulbactram, streptomycin, tetracycline, amoxicyllin- clavulanic acid, trimethropim-sulfametoxazole: 18 (9.5%). The plasmidic profiles detected in the S. flexneri serogrup were 6. Each profile contained between 2 and 7 plasmides and they were excluding. The diversity index was (ID 0.85), whereas in the S. sonnei serogroup there were 8 different profiles. Each one had between 2 and 8 plasmides with ID (0.86). On analyzing the behavior of S. flexneri according to the region of origen, the resistance profile and the plasmidic profile, 2 groupings were created.and 5 groupings were found in the S. sonnei serogroup. The results obtained showed the genetic heterogeneity in the Shigella strains that circulate in the country.
Key words: Shigella, resistance profile, plasmidic profile
Referencias bibliográficas
- López Brea M, Sanz JC, Usera MA, Reina J, Cardeñoso L, Vasallo F. 7. Gastroenteritis bacterianas, víricas, parasitarias y toxiinfecciones alimentarias. En: Procedimientos en Microbiología Clínica. Recomendaciones de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica, España, 1994.
- Lima A, Lima N. Epidemiology, therapy , and prevention of infection with Shigella organism and Clostridium difficile. Curr Op Microbiol Infec Dis 1993;6:63-71.
- Klotoff KL, Winickoff JP, Ivanoff B, Clemens JD, Swerdlow DL et al. Global burden of Shigella infections: Implications for vaccine development and implementation. Bull WHO 1999;77(8):651-66.
- Eisenstein BI. Molecular Techniques for Microbial Epidemiology and the Diagnosis of Infectious Disease. J Infect Dis 1990;161:595-602.
- Marco F, Jiménez de Anta MT. Métodos de tipificación: análisis de plásmidos. Ventajas e inconvenientes. Enferm Infecc Microbiol Clin 1993;11(2):97-101.
- Llop A, Tamargo I, Pérez M, Toraño G, Ramírez M, Bravo L, et al. Antimicrobial resistance and microbiological surveillance in Cuba. En: Salvatierra- González R, Benguigui Y, eds. Antimicrobial resistance in the Americas: magnitude and containment of problem. Washington, DC: Pan American Health Organization; 2000. p. 111-18.
- Ramírez MM, Bravo L, García B, Monté R. Estudio de la susceptibilidad de cepas de Shigella aisladas de niños con enfermedad diarreica aguda. Enferm Inf Microbiol 1996;16(2):112-5.
- Saurina G, Quale JM, Manikal VM. Antimicrobial resistance in Enterobacteriaceae in Brooklyn, NY: epidemiology and relation to antibiotic usage patterns. J Antimicrob Chemother 2000;45:895-8.
- Chin J. El control de las enfermedades transmisibles. 17a ed. Washington DC: OPS; 2001. p. 566-71.
- Bauer AW, Kirby WMM, Sherman TS, Turck M. Antibiotic Susceptibility Testing by Standardized single disk method. Am J Clin Path 1996;45(4):494-5.
- National Committee for Clinical Laboratory Standards. Perfomance Standards for Antimicrobial Susceptibility Testing: Eight Informatical Suplement. M 100-S 10(M2). 2000. p.14-21.
- Kado CI, Liu ST. Rapid procedure for detection and isolation of large and small plasmids. J Bacteriol 1981;145:1365-73.
- Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson's index of diversity. J Clin Microbiol 1988;26:2465-6.
- Herrera S, Cabrera R, Ramírez MM, Usera MA, Echeíta A. Use of AFLP, plasmid typing and phenotyping in a comparative study to asses genetic diversity of Shigella flexneri strains. Epidemiol Infect 2002;129:1-6.
- Guerrant RL, Hughes JM, Lima J. Crane. Diarrhea in developing countries: magnitude, special setting and etiologies. Rev Infect Dis 1999;Suppl 1:S41
- Sosa A Vila J. Infectious disease management in Latin America: a project report. APUA Newsletter 1999;17(2).
- Lima AA, Lima N, Pinho M, Barros E, Texeira M, et al. High frequency of strains multiply resistant to ampicillin, trimethoprim\sulfamethoxazole, streptomycin, chloramphenicol and tetracycline isolates from patients with shigellosis in Northeastern Brazil during the period 1988-1993. Antimicrob Agents Chemother 1995;39(1):256-9.
- Townes JM, Quick R, Gonzales OY, Linares M, Damiani E, et al. Etiology of bloody diarrea in Bolivian children: implication for empiric therapy. J Infect Dis1997;175:1527-30.
- Lima AA, Sidrim JJ, Lima NL, Titlow W, Evans ME, Greenberg RN. Molecular Epidemiology of multiple antibiotic-resistant Shigella flexneri in Fortaleza, Brazil. J Clin Microbiol 1997;35 (5):1061-5.
Recibido: 27 de abril de 2004. Aprobado: 19 de octubre de 2004.
Dra. Margarita Ramírez. Instituto de Medicina Tropical Pedro Kourí. Autopista Novia del Mediodía Km 6 1/2, AP 601, Marianao 13, Ciudad de La Habana. Teléfs. 202-04-36 al 45.
1 Especialista de II Grado en Microbiología. Investigadora Agregada. Instituto de Medicina Tropical Pedro Kourí (IPK).
2 Especialista de I Grado en Microbiología. Centro Nacional de Investigaciones Científicas (CENIC).
3 Licenciada en Biología. Doctora en Ciencias de la Salud. Investigador Auxiliar. IPK
4 Técnica en Farmacia. IPK
5 Técnica en Procesos Biológicos. IPK