Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.59 n.1 Ciudad de la Habana ene.-abr. 2007
Centro de Química Farmacéutica
Identificación fenotípica y molecular de b-lactamasas de espectro extendido TEM y SHV producidas por Escherichia coli y Klebsiella spp. aislados clínicos de hospitales
Lic. Leonora González Mesa,1 Lic. Astrid Ramos Morí,2 Téc. Loreta Nadal Becerra,3 Lic. Janet Morffi Figueroa,4 Dr. Ernesto Hernández Robledo,5 Dra. Ana Berta Álvarez,6 Dr. Juan. J. Marchena Bequer,7 Dra. Mabel González Alemán8 y Dr. Carlos Vallin Plous9
Resumen
Se evaluó la prevalencia de b-lactamasas de espectro extendido (BLEE) tipo TEM y SHV producidas por Escherichia coli y Klebsiella spp. y se determinó la susceptibilidad a otras familias de antimicrobianos. Un total de 326 cepas fueron colectadas entre 2002-2004, procedentes de hospitales en Ciudad de La Habana; los ensayos de susceptibilidad se realizaron de acuerdo con las guías NCCLS y fueron confirmados como productores de BLEE, por el método de doble difusión con discos. La caracterización molecular se realizó mediante la reacción en cadena de la polimerasa, utilizando oligonucleótidos específicos para detectar los genes blaTEM y blaSHV. El fenotipo BLEE fue detectado en 31 de los aislados de Escherichia coli (10 %); de estos 19 (61 %) fueron portadores del gen blaTEM, 5 (16 %) del gen blaSHV; 4 (12 %) portaban ambos genes y 11 (35 %) no portaban ninguno de los genes evaluados. En las cepas de Klebsiella spp. el fenotipo BLEE fue detectado en 10 aislados (36 %) y solo una cepa presentó genotipo blaTEM 1 (10 %). Los antimicrobianos más activos frente Escherichia coli fueron ciprofloxacina (64,5 %) y gentamicina (58,07 %). Los mismos antimicrobianos fueron los más activos para Klebsiella spp., con igual sensibilidad (70 %). Los carbapenémicos aún permanecen activos en las cepas productoras de BLEE, no obstante su uso debe vigilarse cuidadosamente.
Palabras clave: b-lactamasas de espectro extendido (BLEE), Escherichia coli, Klebsiella spp., genotipos TEM y SHV.
Los bacilos gramnegativos son patógenos nosocomiales oportunistas, particularmente importantes en infecciones urinarias, bacteriemias, heridas quirúrgicas y neumonía asociada con ventilador.1 Las infecciones nosocomiales causadas por miembros de la familia Enterobacteriaceae productores de b-lactamasas de espectro extendido (BLEE) representan un importante problema de salud, que se ha incrementado y diseminado rápido en todo el mundo, pues prolongan la duración de las hospitalizaciones y aumentan los costos del cuidado de los pacientes.2
Las b-lactamasas de espectro extendido (BLEE) son enzimas hidrolíticas derivadas de b-lactamasas TEM y SHV, por una o más sustituciones de aminoácidos que confieren resistencia a los antibióticos oximino b-lactámicos. La actividad hidrolítica de las BLEE es inhibida in vitro por el ácido clavulánico, y no afectan las cefamicinas (cefoxitina, cefotetan), ni los carbapenémicos (imipenen, meropenem). Muchas de estas enzimas no muestran un aumento significativo en la concentración mínima inhibitoria (CMI), lo que dificulta su identificación.
El primer aislamiento de BLEE documentado tuvo lugar en Alemania en 1983, a partir de una cepa de Klebsiella ozaenae, y recibió el nombre de SHV-2.3 Estas enzimas están asociadas con megaplásmidos transferibles (>100 kDa), que codifican frecuentemente resistencia cotransferida a aminoglucósidos, cloranfenicol, tetraciclinas y trimetoprim-sulfametoxazol. Estos megaplásmidos se diseminan rápido en los ambientes hospitalarios entre las diferentes especies bacterianas. Las BLEE codificadas por estos elementos móviles pueden ser adquiridas de Enterobacteriaceae resistentes a múltiples antibióticos como K. pneumoniae y Escherichia coli.4
Los métodos de laboratorio para la identificación de BLEE son muy importantes, en la medida en que pueden dirigir el tratamiento de las infecciones causadas por microorganismos productores de estas enzimas. Aunque los métodos para la identificación de BLEE han sido estandarizados solo para K. pneumoniae, K. oxytoca y E. coli, también pueden ser identificadas en otros miembros de la familia Enterobacteriaceae y no-fermentadores. Como existe un aumento en la producción de BLEE en Enterobacteriaceae, esto justifica la necesidad de que los laboratorios de microbiología clínica posean suficiente experiencia para la identificación de esas enzimas en los aislamientos clínicos.5 En este estudio se determinó la prevalencia de BLEE TEM y SHV en bacilos gramnegativos de Escherichia coli y Klebsiella spp. aislados de infecciones nosocomiales en hospitales de Ciudad de La Habana.
Métodos
Cepas bacterianas: este estudio incluyó el análisis de un total de 326 cepas, de los cuales 298 correspondieron a Escherichia coli y 28 a Klebsiella spp., colectadas desde abril de 2002 hasta abril de 2004, de 3 hospitales pediátricos y un hospital ginecoobstétrico de Ciudad de La Habana; las muestras fueron obtenidas a partir de diferentes pacientes con infecciones de heridas quirúrgicas, secreciones vaginales, hemocultivos e infecciones del tracto urinario, fundamentalmente. Los aislados fueron identificados mediante técnicas convencionales,6 y seleccionados como productores de BLEE según la técnica de doble difusión con discos.7
Ensayos de susceptibilidad y confirmación de BLEE: la susceptibilidad a los antibióticos de los diferentes aislados fue determinada por el método de Kirby-Bauer, según los procedimientos descritos en el documento M2-A8 del National Committee for Clinical Laboratory Standards (NCCLS) de 2004,8 con la utilización de los discos de cefotaxima (30 mg), ceftazidima (30 mg), ceftriaxona (30 mg) y aztreonam (30 mg) (Unipath-Oxoid, UK), los cuales son recomendados para aumentar el nivel de detección de aislamientos clínicos que presentan este tipo de enzimas. Fueron confirmadas las cepas productoras de BLEE mediante la técnica de la doble difusión con discos, basada en la sensibilidad de las BLEE, que se manifiesta por la sinergia entre un inhibidor de b-lactamasa (ácido clavulánico) colocado en el centro de una placa de agar de Mueller-Hinton inoculada antes con una suspensión bacteriana del microorganismo a probar y las oximino-cefalosporinas y el aztreonam colocados a una distancia de 25 mm del centro.7 La E. coli ATCC 25922 y K. pneumoniae ATCC 700603 fueron utilizadas como control negativo y positivo, respectivamente, en los ensayos de susceptibilidad.
Técnicas de análisis molecular: la amplificación mediante reacción en cadena de la polimerasa (RCP) de los genes blaTEM se realizó con los oligonucleótidos TEM -1 (5´- ATAAAATTCTTGAAGACGAAA) y TEM- 2 (5´- GACAGTTACCAATGCTTAATCA), descritos por Mabilat y otros.9 La mezcla de reacción se ajustó a un volumen final de 50 mL: 5 mL de buffer de reacción 10x, 5 mL 25mM MgCl2, 100 mM de cada deoxinucleósido trifosfato (Promega), 93,7 pMol del cebador TEM-1, 159,4 pMol del cebador TEM-2, 0,3 mL de 3 U de Taq polimerasa (CIGB, Cuba) y 4 mL del ADN muestra extraído previamente. Las RCP se realizaron en un termociclador Mini Cycler (MJ Research), bajo las condiciones siguientes: 5 min de desnaturalización a 95 °C, 90 s de hibridación a 54 °C y 1 min de extensión a 72 °C, 30 s de desnaturalización a 94 °C, repetir 35 ciclos y una extensión final 5 min a 72 °C. El producto de amplificación se analizó por electroforesis en gel de agarosa 1 % (SunrisetmGibco BRL, Germany), se tiñó con bromuro de etidio (1,0 mg/mL) y se examinó a través de un transiluminador de luz ultravioleta (UV). La corrida electroforética se llevó a cabo a 120 V con solución de Tris Borato EDTA, pH= 8 (TBE). El patrón de peso molecular utilizado fue λ Hind III (Promega).
Para la amplificación de los genes blaSHV se emplearon los oligonucleótidos siguientes: SHV - 1(5´- TCAGCGAAAAACACCTTG) y SHV - 2 (5´- TCCCGCAGATAAATCACCA). descritos por Howard y otros.10 La mezcla de reacción se ajustó a un volumen final de 50 mL: 5 mL de buffer de reacción 10x, 5 mL 25 mM MgCl2, 100 µM de cada deoxinucleósido trifosfato (Promega), 200 pMol del cebador SHV-1, 149 pMol del cebador SHV-2 (CIGB, Cuba), 0,3 mL de 3 U de Taq polimerasa (Promega) y 4 mL del ADN muestra extraído previamente. Las RCP se realizaron en un termociclador Mini Cycler (MJ Research), bajo las condiciones siguientes: 5 min de desnaturalización a 96 °C, 35 ciclos a temperaturas de 96, 55 y 72 °C por 30 s para cada temperatura y un ciclo final de 3 min a 72 °C. El producto de amplificación se analizó por electroforesis en gel de agarosa 1 % (SunrisetmGibco BRL, Germany), se tiñó con bromuro de etidio (1,0 mg/mL) y se examinó a través de un transiluminador de luz ultravioleta (UV). La corrida electroforética se llevó a cabo a 120 V con solución de Tris Borato EDTA, pH= 8 (TBE). El patrón de peso molecular utilizado fue λ Hind III (Promega).
Resultados
De los 326 aislados clínicos colectados de hospitales en Ciudad de La Habana, a partir de muestras de urocultivos, heridas quirúrgicas, exudados vaginales y hemocultivos, se obtuvieron 298 aislamientos pertenecientes a E. coli y 28 a Klebsiella spp. Siguiendo lo establecido en las normas del NCCLS, se realizó un primer descarte teniendo en cuenta los halos de inhibición del antibiograma (tabla 1).
Tabla 1. Zonas de inhibición para detectar posibles BLEE en Escherichia coli y Klebsiella spp.
| Antibiótico | Zona de inhibición para cepas sensibles | Zona de inhibición con posible producción de BLEE |
| Aztreonan 30 mg | ≥ 22 mm | ≤ 27 mm |
| Cefotaxima 30 mg | ≥ 23 mm | ≤ 27 mm |
| Ceftriaxona 30 mg | ≥ 21 mm | ≤ 25 mm |
| Ceftazidima 30 mg | ≥ 18 mm | ≤ 22 mm |
Según los resultados obtenidos en los ensayos de susceptibilidad (tabla 2) fueron resistentes a cefalosporinas de espectro extendido: 140 cepas de E. coli y 25 de Klebsiella spp.
Tabla 2. Porcentaje de cepas con patrones de sensibilidad, intermedio y resistencia a las cefalosporinas de espectro extendido
| Microorganismos | Patrones | CAZ (%) | CRO (%) | CTX (%) | ATM (%) |
| Escherichia coli (n=298) | Sensibilidad | 84,2 | 83,6 | 82,9 | 83,6 |
| Intermedio | 11,1 | 3,4 | 3 | 1,3 | |
| | 4,7 | 13,1 | 14,1 | 15,1 | |
| Klebsiella spp. (n=28) | Sensibilidad | 82,1 | 71,4 | 71,4 | 71,4 |
| Intermedio | 7,1I | 7,10 | 0 | 0 | |
| Resistencia | 10,7 | 21,4 | 28,6 | 28,6 |
Ceftazidima (CAZ), ceftriaxona (CRO), cefotaxima (CTX), monobactam: aztreonam (ATM).
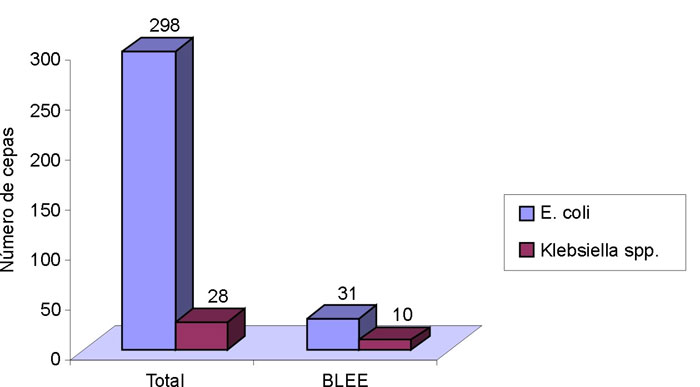
La presencia de b-lactamasas de espectro extendido (BLEE) se manifestó por el efecto sinérgico del inhibidor, bajo la forma de una ampliación del halo de inhibición en uno o varios de los b-lactámicos probados, este fenotipo se observó en 31 aislados de E. coli (10 %) y 10 de Klebsiella spp. (36 %), por tanto estos aislados fueron confirmados como productores de β-lactamasas de espectro extendido (BLEE) (fig. 1).
Fig. 1. Total de cepas aisladas y total de cepas portadoras de b-lactamasas de espectro extendido.
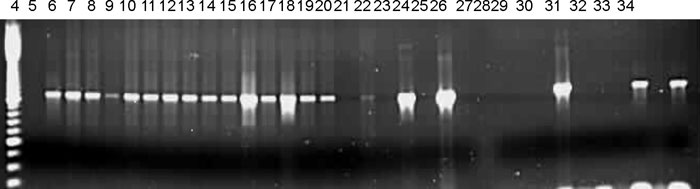
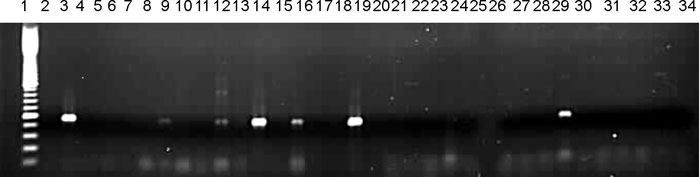
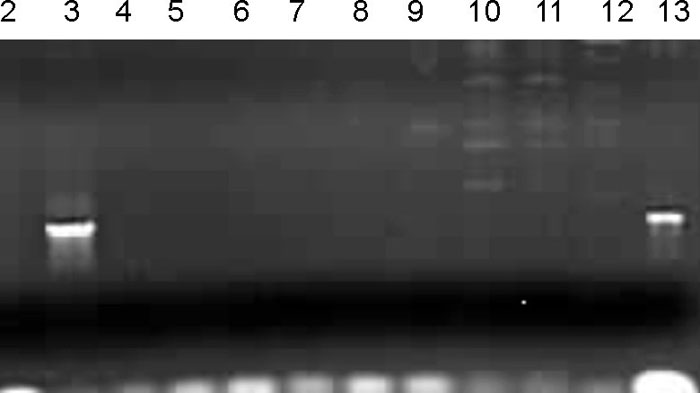
La reacción en cadena de la polimerasa (RCP) realizada a 31 (10 %) de los aislados de Escherichia coli con fenotipo BLEE positivo, dio como resultado que la mayoría 19 (61 %) presentaron genotipo blaTEM (fig. 2), y muy pocos aislados, 5 (16 %), presentaron genotipo blaSHV (fig. 3). Algunos aislados fueron positivos para la combinación de ambos genes y en el caso de Klebsiella spp. solo un aislado presentó genotipo blaTEM (fig. 4).
Línea 1: estándar de peso molecular λ Hind III; línea 2: control negativo E. coli ATCC 25922; línea 3: control positivo E. coli que presenta el gen blaTEM-5; de la línea 4 a la línea 34, aislados de E. coli.
Fig. 2. Electroforesis en gel de agarosa para visualizar el producto de la RCP correspondiente a la amplificación de un fragmento del gen blaTEM.
Línea 1: estándar de peso molecular λ Hind III; línea 2: control negativo E. coli ATCC 25922; línea 3.: control positivo Klebsiella pneumoniae que presenta el gen blaSHV-18; de la línea 4 a la línea 34, aislados de E. coli.
Fig. 3. Electroforesis en gel de agarosa para visualizar el producto de la RCP correspondiente a la amplificación de un fragmento del gen blaSHV.
Línea 1: estándar de peso molecular λ Hind III; línea 2: control negativo E. coli ATCC 25922; línea 3: control positivo E. coli que presenta el gen blaTEM-5; de la línea 4 a la línea 13, aislados de Klebsiella spp.
Fig. 4. Electroforesis en gel de agarosa para visualizar el producto de la RCP correspondiente a la amplificación de un fragmento del gen blaTEM.
El perfil fenotípico de susceptibilidad a otros antimicrobianos en los aislamientos de Klebsiella spp. y E. coli productores de BLEE, es el que se reporta a continuación: cefepime (50,0 %, 77,41 %); gentamicina (30,0 %, 41,93 %); tobramicina (60 %, 87,07 %); ciprofloxacina (30,0 %, 35,48 %); amoxicilina/ac. clavulánico (90,0 %, 90,32 %), respectivamente, 100 % de resistencia a las cefalosporinas de tercera generación y aztreonam; sensibilidad de 100 % para imipenem y meropenem (tabla.3).
Tabla 3. Porcentaje de resistencia de los antibióticos recomendados en aislados clínicos productores de BLEE
| Antimicrobianos | % de resistencia (n) | |
| Klebsiella. spp. (28) | E. coli (298) | |
| Meropenem | 0,0 | 0,0 |
| Imipenem | 0,0 | 0,0 |
| Cefepime | 50,0 | 77,41 |
| Gentamicina | 30,0 | 41,93 |
| Tobramicina | 60,0 | 87,07 |
| Ciprofloxacina | 30,0 | 35,48 |
| Amoxicilina/ácido clavulánico | 90,0 | 90,32 |
n: número de cepas.
Discusión
De las muestras procesadas, 94 % pertenecía a los resultados de urocultivos, esto demuestra la gran prevalencia de esos patógenos en el tracto urinario, lo cual se corresponde con reportes de trabajos recientes de que Escherichia coli es la especie bacteriana más aislada y el sitio donde más frecuente se aíslan es el tracto urinario.11,12
Como se observa en los resultados, 109 aislados de E. coli y 15 de Klebsiella spp. fueron resistentes a cefalosporinas de espectro extendido, pero no expresaron el fenotipo de BLEE; en este caso la técnica de la doble difusión con discos pudo fallar y no detectó la presencia de BLEE si esta enzima está presente junto a otras b-lactamasas que enmascaran el efecto del ácido clavulánico, como la cefalosporinasas de Bush tipo I (AmpC, no inhibidas por el ácido clavulánico) o estos aislados presentan otros mecanismos de resistencia como la disminución de la permeabilidad de la membrana al paso del antibiótico.13
Estudios del National Nosocomial Infections Surveillance Systems14 (NNIS), realizados entre 1986 y 1993 en EE. UU. en cepas de K. pneumoniae, informaron tasas de BLEE de hasta 58 %. En otro estudio realizado en 1998 por el programa de vigilancia epidemiológica, Resisnet15 en América Latina, informó que la prevalencia de BLEE por E. coli en algunos países llega casi a 65 % y en K. pneumoniae la prevalencia es aún más alta pues llega hasta 73 %. Todos estos estudios muestran una variación geográfica en cuanto a la prevalencia de BLEE en K. pneumoniae y E. coli en el mundo.
Los resultados de estudios del Antimicrobial Resistance Surveillance Program (SENTRY)16 que monitorea la frecuencia de aparición y susceptibilidad antimicrobiana de patógenos causantes de infecciones en el mundo, informó en América Latina entre 1997 y 1998 una alta prevalencia de BLEE en E. coli y K. pneumoniae, las cuales superan 40 %.17
En estudios más recientes del programa SENTRY se encontró que los aislamientos de K. pneumoniae con fenotipo BLEE fueron más prevalentes en América Latina (45,4 %), seguidos por la región Pacífica (24,6 %), Europa (22,6 %) y Canadá (4,9 %).18
Coincidiendo con lo que se plantea en la literatura, la mayoría de las BLEE son derivadas de las enzimas TEM y SHV, donde las tipo TEM resultan las más abundantes en E. coli.19, 20 Hoy día existen más de 90 BLEE tipo TEM y más de 45 BLEE tipo SHV.21 La nomenclatura de las BLEE actualmente es monitoreada en un sitio Web dirigido por G. A. Jacoby y K. Bush (http://www.lahey.org/studies/webt.stm), donde están disponibles las secuencias de aminoácidos y las referencias bibliográficas, para todas las variantes TEM, SHV y OXA.22
La resistencia cruzada observada para otros antibióticos no beta-lactámicos ha sido informada en otros estudios,23 en los cuales los porcentajes de esta corresistencia alcanzaron altos niveles, lo que limita el sinergismo terapéutico entre estas familias de antibióticos. La resistencia presentada a los aminoglucósidos por las cepas productoras de BLEE, es probable se deba a la expresión de enzimas modificantes de aminoglucósidos tipo N-6`-acetiltransferasas y N-2`-nucleotidiltransferasas. La presencia de estas enzimas modificantes sugieren el amplio uso de gentamicina y tobramicina en los hospitales estudiados.24 El presente estudio también encontró resistencia a las fluoroquinolonas de últimas generaciones como ciprofloxacina. El porcentaje de resistencia a quinolonas ha venido incrementándose de manera paralela con la producción de BLEE, como lo han informado otros autores;5 sin embargo, no se considera una característica específica en cepas productoras de BLEE dado que esta resistencia se ha presentado en productores y no productores de estas enzimas.
La prevalencia de BLEE en K. pneumoniae y E. coli indica que los antibióticos de primera línea para estos microorganismos debería ser modificada por el uso prudente de imipenem o meropenem, para evitar la selección de mutantes resistentes de P. aeruginosa y Acinetobacter spp. a estos agentes antimicrobianos.25
Sin embargo, la administración de piperacilina y tazobactam sería útil para el tratamiento de las infecciones causadas por K. pneumoniae y E. coli productoras de BLEE, así como el tratamiento de reemplazo a imipenem, considerando que los microorganismos solo expresen la producción de BLEE y no otro tipo de b-lactamasas como AmpC o sus variantes.26, 27
En conclusión, la producción de BLEE en Klebsiella spp. y E. coli en los hospitales involucrados en el estudio y los niveles de resistencia elevados frente a otros antibióticos como los aminoglucósidos, cefepime y amoxicilina/ácido clavulánico, indica posiblemente la presencia de plásmidos transferibles que por causa de la presión selectiva ejercida por los antibióticos estén diseminando la resistencia en estos hospitales. La prevalencia de BLEE en E. coli y Klebsiella spp., sugiere restringir el uso de b-lactámicos de amplio espectro e implementar medidas de higiene para el control de las infecciones y la prevención de la diseminación de microorganismos productores de BLEE.
Phenotypic and molecular identification of extended-spectrum b-lactamase (ESBL) TEM and SHV produced by clinical isolates Escherichia coli and Klebsiella spp. in
Summary
Nosocomial infections caused by gram-negative bacilli which produce extended spectrum β-lactamase (ESBL) are associated with the increase of morbidity and mortality in hospitals. The objective of this study was to evaluate the frequency of ESBL, specifically the TEM and SHV type, produced by Escherichia coli and Klebsiella spp. strains, and also to determine the antimicrobial susceptibility of these isolates in comparison with other antibiotic families. A total of 326 strains were collected between 2002-2004 from hospitals in
Key words: Extended-spectrum beta-lactamase (ESBL), Escherichia coli, Klebsiella spp., genotype TEM and SHV.
Referencias bibliográficas
1. Bergogne BE, Towner KJ. Acinetobacter spp. as nosocomial pathogens: microbiological, clinical and epidemiological features. Clin Microbiol Rev 1996;9:148-65.
2. Medeiros AA. Evolution and dissemination of b-lactamases accelerated by generations of ·-lactam antibiotics. Clin Infect Dis 1997;24(suppl.1):19-45.
3. Knothe HP, Shah V, Kremery M, Mitsuhashi S. Transferable resistance to cefotaxime, cefoxitin, cefamandole and cefuroxime in clinical isolates of Klebsiella pneumoniae and Serratia marcescens. Infection 1983;11:315-7.
4. Wong BA. Therapeutic challenges associated with extended-spectrum, b-lactamases producing E. coli and K. pneumoniae. Pharmacotherapy 2001;21:583-92.
5. Martínez P, Mercado M, Máttar S. Determinación de b-lactamasas de espectro extendido en gérmenes nosocomiales del Hospital San Jerónimo, Montería. Colomb Med 2003;34:130-9.
6. Farmer JJ. Enterobacteriaceae: introduction and identification. En: Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH, eds. Manual of clinical microbiology, 6th ed. Washington, DC: American Society for Microbiology; 1995. p. 438-49.
7. Jarlier V, Nicolas MH, Fournier G, Philippon A. Extended broad-spectrum b-lactamases conferring transferable resistance to newer b-lactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis 1988;10:867-78.
8. NCCLS. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically. Approved Standard M2-A8, M7-A6. Vol. 24 No. 1. Wayne, Pa: National Committee for Clinical Laboratory Standards; 2004.
9. Mabilat C, Goussard S. PCR detection and identification of genes for extended-spectrum ·-lactamases. En: Persing DH, Smith TF, Tenover FC, White TJ ed. Diagnostic molecular microbiology: principles and applications. Washington, DC: American Society for Microbiology; 1993. p. 553-9.
10. Howard C, Van Daal A, Kelly G, Schooneveldt J, Nimmo G, Giffard PM. Identification and minisequencing-based discrimination of SHV b-lactamases in nosocomial infection-associated Klebsiella pneumoniae in Brisbane, Australia. Antimicrob Agents Chemother 2002; 46:659-64.
11. Andrasevic AT, Tambic T, Kalenic S, Jankovic V, the Working Group of the Croatian Committee for Antibiotic Resistance Surveillance. Surveillance for Antimicrobial Resistance in Croatia. Emerg Infect Dis 2002;8(1):14-8.
12. Aubry-Damon HH, Courvalin P. Resistance to antimicrobial agents: selected problems in France, 1996 to 1998. Emerg Infect Dis 1999;5(3):315-20.
13. Sánchez AB. Betalactamasas de espectro extendido (BLEE). Revista Electrónica de Medicina Intensiva. 2004;4 (8):1-16. URL disponible en : ©REMI, http://remi.uninet.edu/.
14. Monnet DL, Biddle JW, Edwards JR. Evidence of interhospital transmission of extended spectrum ·-lactamases resistant Klebsiella pneumoniae in the United States, 1986 to 1993. The National Nosocomial Infections Surveillance Systems. Infect Control Hosp Epidemiol 1997;18:492-8.
15. Sifuentes J, Prado V, Zurita J. Is the antimicrobial resistanse in Pseudomonas aeruginosa y Acinetobacter spp. expanding in Latin-America. The Resisnet Collaborative Group. Abstracts 120 of the IDSA Philadephia:37th Annual Meeting; 1999. p. 60.
16. Sader HS, Pfaller MA, Jones RN. Bacterial pathogens isolated from patient with bloodstream infections in Latin-America, 1997. Frequency of occurrence and antimicrobial susceptibility patterns from the SENTRY antimicrobial surveillance program. Braz J Infect Dis 1999;3:97-110.
17. Jones RN. Global Epidemiology of Antimicrobial Resistance among Community-Acquired and Nosocomial Pathogens: A Five-Year Summary from the SENTRY Antimicrobial Surveillance Program (1997-2001). Semin Respir Crit Care Med 2004;24(1):121-34.
18. Martínez P, Espinal P, Bustos A, Mattar S. Prevalencia de Klebsiella pneumoniae y Escherichia coli productoras de b-lactamasas de espectro extendido (BLEE), en el Hospital San Jerónimo de Montería. Med UNAB 2005;8(1):15-22.
19. Bush K, Jacoby GA, Medeiros AA. A functional classification scheme for ·-lactamases and its correlation with molecular structure. Antimicrob. Agents Chemother 1995;39:1211-33.
20. Jacoby GA, Medeiros AA. More extended-spectrum b-lactamases. Antimicrob Agents Chemother 1991;35:1697-704.
21. Bradford PA. Extended-spectrum b-lactamases in the 21st century: characterization, epidemiology, and detection of this important resistant threat. Clin Microbiol Rev 2001;14:933-51.
22. Bush K. Identifying Resistance. ESBL in Enterobacteriaceae. International Newsletter 2003;4:1-6.
23. Winokur PL, Canton R, Casellas JM, Legakis N. Variations in the prevalence of strains expressing an extended-spectrum b-lactamases phenotype of isolates from Europe, the Americas and the Western Pacific Region. Clin Infect Dis 2001;32:94-103.
24. Millar GH, Sabatelli FJ, Hare RS, Glupczynski N, Mackey P, Sales D, et al. The most frequent aminoglicoside resistance mechanisms-changes with time and geographic area: A Reflection of aminoglicoside usage patterns? Clin Infect Dis 1997;24(Suppl 1):S46-S62.
25. Valverde A, Coque T, Sánchez P, Rollán A, Baquero F, Cantón R. Dramatic increase in prevalence of fecal carriage of extended-spectrum b-lactamase-producing Enterobacteriaceae during nonoutbreak situations in Spain. J Clin Microbiol 2004;42:4769-75.
26. Nasim K, Elsayed S, Pitout J, Conly J, Church D, Gregson D. New method for laboratory detection of AmpC b-lactamases in Escherichia coli and Klebsiella pneumoniae. J Clin Microbiol 2004;42:4799-802.
27. Paterson DL, Mulazimoglu L, Casellas JM, Ko WC, Goznes H, Von Gottberg A, et al. Epidemiology of ciprofloxacin resistance and its relationship to extended-spectrum ·-lactamase production in Klebsiella pneumoniae isolates causing bacteremia. Clin Infect Dis 2000;30:473-8.
Recibido: 15 de marzo de 2006. Aprobado: 18 de noviembre de 2006.
Dra. Leonora González Mesa. Centro de Química Farmacéutica, Calle 200 y Ave. 21, Reparto Atabey, AP 16042, CP 11600, Playa, Ciudad de La Habana, Cuba. Teléf.: 208 52 36, ext. 236. Fax: (537) 33 64 71, 33 04 97. Correo electrónico: leonora.gonzalez@cnic.edu.cu
1 Licenciada en Biología. Especialista en Microbiología. Centro de Química Farmacéutica.
2 Licenciado en Microbiología. Investigador Agregado. Centro de Química Farmacéutica.
3 Técnico Medio en Química. Centro de Química Farmacéutica.
4 Licenciada en Microbiología. Aspirante a Investigadora. Centro de Química Farmacéutica.
5 Máster en Ciencias en Microbiología. Hospital Pediátrico "Juan Manuel Márquez".
6 Doctora en Medicina. Hospital Ginecoobstétrico "Eusebio Hernández".
7 Doctor en Medicina. Hospital Pediátrico de Centro Habana Benjumeda y Morales.
8 Doctora en Medicina. Hospital Pediátrico "William Soler".
9 Doctor en Ciencias Biológicas. Investigador Titular. Centro de Química Farmacéutica.