Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión On-line ISSN 1561-3054
Rev Cubana Med Trop v.61 n.1 Ciudad de la Habana ene.-abr. 2009
ARTÍCULO ORIGINAL
Variabilidad genética de poblaciones de Triatoma flavida (Hemiptera: Reduviidae) en la península de Guanahacabibes
Genetic variability of Triatoma flavida (Hemiptera: Reduviidae) in Guanahacabibes
Jorge Fraga NodarseI; Jinnay Rodríguez RodriguezII; Omar Fuentes GonzálezIII; Mayda Castex RodriguezIV; Aymé Fernández-Calienes ValdésV
I Máster en Parasitología. Licenciado en Bioquímica. Investigador Agregado. Profesor Instructor, Departamento de Parasitología, Instituto de Medicina Tropical "Pedro Kourí" (IPK).
II Máster en Control de Vectores. Licenciada en Biología. Investigadora Agregada. Departamento de Control de Vectores, IPK.
III Licenciado en Biología. Investigador Auxiliar. Departamento de Control de Vectores, IPK.
IV Licenciada en Biología. Investigadora Agregada. Departamento de Control de Vectores, IPK.
V Máster en Bioquímica. Licenciada en Bioquímica. Investigadora Auxiliar. Departamento de Parasitología, IPK.
RESUMEN
INTRODUCCIÓN: la enfermedad de Chagas es una enfermedad transmitida por triatomineos. Triatoma flavida es una especie selvática autóctona de Cuba, que presumiblemente es atraída a las casas por la luz, de las especies encontradas en Cuba es la más abundante.
OBJETIVO: se investigó la variabilidad genética intrapoblacional e interpoblacional de ejemplares de T. flavida colectados en la región occidental de Cuba, utilizando la técnica del ADN polimórfico amplificado al azar, con la determinación además de posibles relaciones genéticas entre las poblaciones.
MÉTODOS: un total de 10 cebadores al azar (OPA-1 al 10) fueron usados para evaluar la variabilidad genética dentro de una población y entre 9 poblaciones diferentes de T. flavida, mediante la técnica de ADN polimórfico amplificado al azar. Además, se evaluó la diversidad genética entre individuos salvajes y de la primera generación (F1) obtenidos en el laboratorio, así como entre los diferentes estadios de esta especie.
RESULTADOS: no se detectaron diferencias en los patrones de amplificación del ADN entre los individuos silvestres y de la F1; al igual que entre los diferentes estadios de esta especie. Se encontró homogeneidad genética dentro de la población estudiada y una variabilidad genética baja entre las diferentes poblaciones de T. flavida. Se obtuvieron 2 grupos bien definidos según el análisis del ADN polimórfico amplificado al azar, mostrando concordancia con el origen geográfico, en las poblaciones capturadas en áreas del occidente y el oriente de Guanahacabibes, Pinar del Río. Entre estas poblaciones se encontró una pequeña diferenciación genética (Fst 0,030) y tasas de migración (N> 1) que revelan flujo genético y homogeneidad genética.
CONCLUSIONES: los resultados presentados en este estudio establecen una aproximación a la estructura genética de T. flavida. La homogeneidad genética encontrada entre los individuos silvestres de T. flavida constituye un aspecto importante para la implementación de las políticas de control de este vector.
Palabras clave: Triatoma flavida, ADN polimórfico amplificado al azar, poblaciones, triatomineos, flujo genético.
ABSTRACT
INTRODUCTION: chagas disease is a Triatomineos-borne disease. Triatoma flavida is an indigenous Cuban species, presumably attracted to houses by light and it is the most abundant species in the country.
OBJECTIVE: the intrapopulatinal and interpopulational genetic variability of T. flavida collected in the western region, using the Random Amplified Polymorphic DNA (RAPD) technique, thus determining possible genetic relationships among the populations.
METHODS: a total of ten random primers (OPA-1 at the 10) were used to evaluate the genetic variability in one population and among 9 populations of T. flavida using the RAPD technique. We also evaluate the genetic diversity among wild individuals and their first generation (F1) obtained in the laboratory as well as different stages of this species.
RESULTS: differences were not detected in the amplification patterns among the wild individuals and the F1. The same results were achieved among different life cycles of this species. Genetic homogeneity of the studied population and low genetic variability among different T. flavida populations were observed. Two well-defined groups were obtained according to random amplified polymorphic DNA data; they matched with the geographical origin in the populations captured in areas from east and west of Guanahacabibes, Pinar del Río. Among these populations, small genetic differentiation (Fst 0,030) was found as well as migration rates (N> 1), which reveals the gene flow and genetic homogeneity.
CONCLUSIONS: The results of this study constitute an approach to the genetic structure of T. flavida. The genetic homogeneity between wild individuals of T. flavida represents an important item for the implementation of the vector control programs.
Key words: Triatoma flavida, RAPD, populations, Triatomineos, genetic flow.
INTRODUCCIÓN
Los triatomineos (Hemiptera, Reduviidae) desempeñan un papel importante en la transmisión de la Enfermedad de Chagas, porque al picar a sus hospederos para alimentarse defecan y depositan en su deyección Trypanosoma cruzi, causante de esta enfermedad. Los cálculos de la Organización Mundial de la Salud indican la existencia de 16 a 18 millones de personas infectadas por T. cruzi, alrededor de 100 millones en riesgo de ser infectadas y 50 000 muertes anuales, por lo cual la enfermedad de Chagas se considera, por amplio margen, la enfermedad parasitaria más grave de las Américas.1 La transmisión por transfusión de sangre parece ir en aumento en muchas ciudades latinoamericanas, pero en general la mayor transmisión todavía se debe a las poblaciones domésticas de insectos vectores, de ahí que eliminar estas poblaciones con insecticidas constituye hoy día la única medida de control efectiva. Estos insectos están agrupados en 6 tribus, 19 géneros y 137 especies; el género Triatoma es el más numeroso, con cerca de 82 especies.2
En Cuba existe el riesgo de la transmisión vectorial al encontrarse en la isla 4 especies de triatomineos: Triatoma flavida (Neiva, 1911), Triatoma bruneri (Usinger, 1944), Bolbodera scabrosa (Valdés, 1990) y Triatoma rubrofasciata (De Geer, 1973). La última especie es un reconocido vector cosmopolita y junto con T. flavida, probados transmisores de T. cruzi, capaces de infectar seres humanos.3
La especie Triatoma flavida es la más abundante de las especies encontradas en Cuba. Fue hallada e identificada por Valdés en 1910 y descrita por Neiva en 1911, es considerada una especie selvática que presumiblemente es atraída a las casas por la luz.4
La tendencia actual en América es el estudio de las especies peridomésticas, porque se ha visto que desempeñan un papel determinante en la transmisión de la enfermedad de Chagas, por no recibir directamente las aplicaciones intradomiciliarias de pesticidas que se dirigen al control de las especies domésticas.
En el caso de Cuba, la especie con una mayor presencia, T. flavida, es una especie silvestre con un aparente proceso de acercamiento a las viviendas, lo que aumentaría el contacto hombre-vector, aun cuando no se han encontrado evidencias del establecimiento de colonias en las casas.3,4
El análisis de la genética poblacional de triatomineos constituye un elemento esencial en el conocimiento de las especies de triatomas. Con esto se pueden diseñar de manera eficiente las políticas de control de estos vectores. Este análisis permite distinguir entre individuos supervivientes e inmigrantes, elemento importante para conocer los motivos de las reinfecciones posteriores al control y permite además identificar poblaciones de insectos con riesgo futuro de ser domiciliarios, así como el potencial de dispersión y flujo genético.5
En el estudio de la estructura genética de varias especies de triatomineos se han utilizado ampliamente diversas técnicas moleculares, como la técnica del ADN polimórfico amplificado al azar (RAPD, por sus siglas en inglés), la secuenciación de genes mitocondriales, microsatélites, y el análisis de genes y espaciadores ribosomales.6-10
La técnica del RAPD provee un método sensible y muy eficaz para obtener marcadores genéticos, aplicables a organismos muy diferentes y capaces de detectar diferencias entre individuos que muestran una relación genética estrecha. El análisis del RAPD no requiere conocimiento previo sobre la secuencia del ADN de las muestras a analizar. Además, se ha demostrado que la información provista por los polimorfos del RAPD es consistente con aquella obtenida por medio de otros ensayos moleculares.11 Todas estas propiedades hacen del RAPD una herramienta muy atractiva para estudios de variabilidad genética en poblaciones.
Esta técnica se ha utilizado para el análisis de la estructura poblacional de varias especies de triatomineos como T. infestans, T. dimidiata, T. brasiliensis, T. venosa, Rhodnius prolixus, R. colombiensis, R. robustus; permite discriminar especies, conocer las variaciones genéticas y estimar el flujo genético entre poblaciones silvestres, peridomiciliarias y domiciliarias, de estos vectores.6-13
En este trabajo se presenta el estudio del polimorfismo genético intrapoblacional e interpoblacional de Triatoma flavida colectados en la Península de Guanahacabibes, Pinar del Río, usando la técnica de RAPD, se determinó además posibles relaciones genéticas entre las poblaciones.
MÉTODOS
Triatomineos
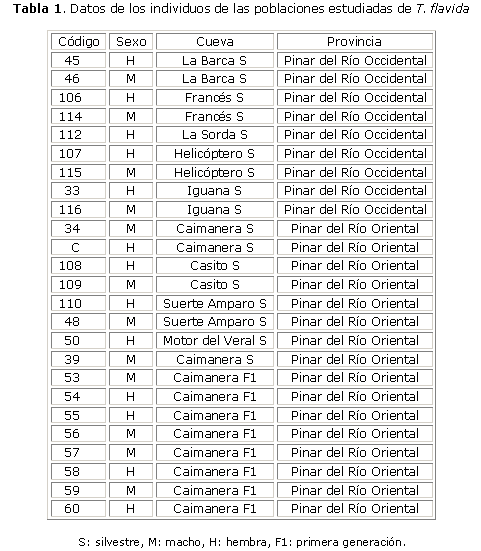
Los triatomineos fueron capturados en cuevas de la Península de Guanahacabibes, provincia Pinar del Río, Cuba (fig. 1). Los insectos se mantuvieron en condiciones de laboratorio a 26 ± 2 °C y 76 ± 2 % de humedad relativa. Todos los vectores fueron clasificados como T. flavida siguiendo las claves de Usinger (1944).14 De las poblaciones colectadas en condiciones de laboratorio se obtuvieron individuos de la primera generación (F1).
Un total de 25 individuos fueron estudiados: 9 hembras y 7 machos silvestres, y 8 individuos obtenidos de la generación F1 en condiciones de laboratorio provenientes de la población de Caimarera (4 hembras y 4 machos) (tabla 1). Además, se estudiaron los diferentes estadios de la embriogénesis de T. flavida (huevo y ninfas del 1ro. al 5to. estadio), tomados de la población de laboratorio proveniente de Caimanera.
Se utilizó además en el estudio un individuo de Triatoma rubrofasciata, procedente de la colección del Departamento de Control de Vectores del Instituto de Medicina Tropical "Pedro Kourí" como especie fuera de grupo.
Extracción de ADN
A los triatomas colectados se le aisló el ADN genómico a partir de una pata utilizando el método de acetato de potasio modificado.15 La concentración de ADN se estimó en un espectrofotómetro mediante su absorbancia a 260 nm. La pureza de la muestra se examinó por electroforesis en un gel de agarosa [0,8 % en tampón TBE (tris-borato 0,045 M; EDTA 0,001 M) que contenía bromuro de etidio (0,5 mg/mL)] y se utilizó un transiluminador UV (Macrovue 2011, LKB, Suecia) para su visualización.
Técnica del ADN polimórfico amplificado al azar (RAPD)
Se usaron 10 cebadores (OPA-1-OPA-10, Kit A, Operon Technologies Inc, California) (EUA) en la amplificación de marcadores al azar de RAPD, para determinar el polimorfismo genético entre individuos de T. flavida. Los reactivos y las concentraciones utilizadas en la técnica de RAPD fueron optimizadas previamente.16 La amplificación del ADN se realizó en un volumen final de 25 mL que contenía 2,5 mL de tampón de amplificación 10 x (Promega, EUA), 200 mM de cada dNTP (Promega, EUA), 5 pmol de cebador, 2,5 mM de MgCl2; 2,0 U Taq ADN polimerasa (Promega, EUA) y 25 ng de ADN molde a amplificar. Se incluyó un control negativo en cada ensayo, donde la mezcla de reacción contenía agua destilada estéril en lugar de ADN, con el objetivo de descartar cualquier contaminación. Posteriormente, se procedió a efectuar la reacción, en un termociclador (Perkin Elmer, EUA) con el siguiente perfil de amplificación: desnaturalización inicial a 94 °C por 5 min, seguido de 45 ciclos de desnaturalización a 94 °C por 1 min, hibridación (annealing) a 36 °C por 1 min y extensión a 72 °C por 2 min, con una extensión final después del último ciclo a 72 °C por 15 min. Para la detección del producto se analizaron 20 mL de cada mezcla resultante, en electroforesis en gel de agarosa 1,2 %, preparado en tampón TBE 0,5x con bromuro de etidio (0,5 mg/mL). La visualización de los productos de amplificación se hizo mediante luz ultravioleta utilizando un transiluminador UV (Macrovue 2011, LKB, Suecia).
Análisis del polimorfismo genético
El análisis se realizó bajo 3 supuestos: 1. las poblaciones están en equilibrio de Hardy-Weinberg, o sea, que se asume que no hay presiones de selección que favorezcan alguna población; 2. los marcadores RAPD segregan en forma mendeliana con tasas constantes de evolución y sustitución; y 3. los alelos recesivos son idénticos entre los individuos.
Las bandas individuales de RAPD se apuntaron como presentes o ausentes (1 o 0) para cada individuo, a partir de lo cual se construyó una matriz binaria y el inverso del coeficiente de similitud de Jaccard (Sneath 1957)17 fue calculado para cada par de individuos analizados, que resultó una matriz de distancia.
Teniendo en cuenta que en los marcadores RAPD el fenotipo dominante de un locus se considera como la presencia de una banda, y el fenotipo recesivo es la ausencia de esa banda, los individuos se compararon fenotípicamente en cada locus.
La relación filogenética se determinó mediante la estrategia de agrupamiento mediante el método de análisis no pareado de media aritmética (UPGMA, del ingles, Unweighted Pair Group Method with Arihtmetical Averages), utilizando el paquete de Programas FreeTree, versión 0.9.1.59.18-22 El dendograma se construyó basado en los resultados obtenidos con los 10 cebadores y el análisis de bootstrap se realizó con 1 000 réplicas.
El estadístico F (Fst) y la tasa efectiva de migración (Nm) entre poblaciones se estimaron utilizando el programa RAPDFST mediante 3 metodologías: Wright,19 Weir y Cockerham20 así como Lynch y Milligan.21 El Fst es la razón de la varianza observada en la frecuencia de un alelo en un locus RAPD entre subpoblaciones y su máxima varianza en su población total; el programa computa los valores Fst para cada locus RAPD asumiendo que las poblaciones están en equilibrio de Hardy-Weinberg y la dominancia de cada loci. Los valores de Fst y Nm son estimaciones indirectas del flujo genético y la migración entre poblaciones, en las cuales se asume que no hay selección ni mutación.
RESULTADOS
Al analizar el polimorfismo genético entre 9 poblaciones de T. flavida colectados en la provincia de Pinar del Río, Cuba, distribuidos en diferentes zonas geográficas dentro de la Península de Guanahacabibes (fig. 1), los 10 cebadores analizados generaron 90 bandas de amplificación de RAPD reproducibles, dentro de las cuales 29 mostraron polimorfismo entre las poblaciones analizadas y 61 marcadores están presentes en todos los individuos analizados.
Para estudiar la variabilidad genética intrapoblacional se analizaron 8 individuos de T. flavida procedentes de la cueva de Caimanera, primera generación (F1) (4 hembras y 4 machos), utilizando los cebadores OPA-1 al OPA-5. Se analizaron 34 bandas de amplificación entre los individuos estudiados y no se encontraron marcadores polimórficos, de igual forma no se encontró polimorfismo de ellos con los individuos salvajes de esta población (datos no mostrados). Lo mismo sucede entre los diferentes estadios de la embriogénesis de T. flavida (huevo, ninfas del 1ro. al 5to. estadio y adulto), donde no se encontraron diferencias en el patrón de RAPD entre los diferentes estadios ni entre ellos con el adulto, al analizar los 5 cebadores (datos no mostrados).
El dendograma de la figura 2 obtenido a partir del patrón de bandas generados al analizar los 10 cebadores, muestra que de acuerdo con la distancia genética entre los individuos se formaron 2 grupos, que coinciden con los individuos colectados en las cuevas de la región occidental y oriental de Guanahacabibes. Las poblaciones del occidente se agruparon en un conglomerado con una distancia genética de 0,074 (coeficiente de similitud 92,6 %), mientras que las del oriente se agruparon en un conglomerado con una mayor distancia genética de 0,104 (coeficiente de similitud 89,6 %). El individuo analizado procedente de la cueva Motor del Veral, presentó la mayor variabilidad genética de los individuos agrupados correspondiente a la región oriental.
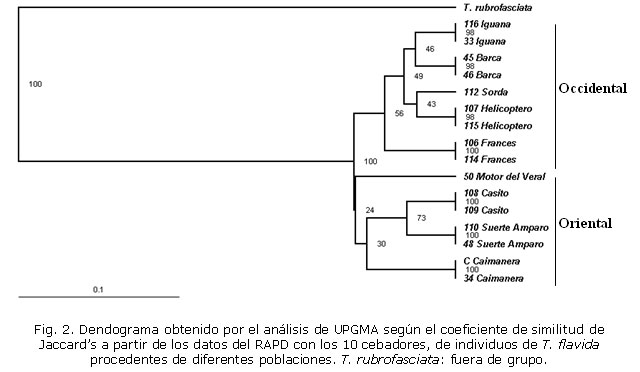
Con los cebadores OPA-3, 5, 6, 8, 9 y 10 no se encontró variabilidad genética entre las poblaciones de T. flavida analizadas. Sin embargo, los cebadores OPA-1, 2, 4 y 7 mostraron polimorfismo en los individuos estudiados (fig. 3). La homogeneidad en el patrón de bandas obtenido por RAPD entre las poblaciones del oriente y el occidente, sugiere una distancia genética pequeña entre los individuos silvestres de T. flavida. Las poblaciones se encuentran separadas con una distancia genética de 0,107 (coeficiente de similitud 89,3 %). Se estimó el índice de fijación o estadístico F (Fst) que mide el grado de diferenciación genética entre las poblaciones del oriente y el occidente, y la tasa de migración (Nm). Los valores de Fst obtenidos por las 3 metodologías indican una diferenciación genética pequeña (Fst< 0,05) y una tasa de migración (Nm> 1) suficiente para mantener homogeneidad genética entre los individuos (tabla 2).

Con el cebador OPA-1 y OPA-4 se encontraron marcadores genéticos capaces de diferenciar las poblaciones del oriente a las del occidente de la península de Guanahacabibes. En la figura 3 A se observa el patrón de bandas de RAPD obtenido al utilizar el cebador OPA-1, donde aparece un marcador de 737,5 pb en los individuos procedentes de poblaciones de la región oriental que no está presente en los de la zona occidental; igualmente, con el cebador OPA-4 aparece un marcador genético de 750 pb que se encuentra presente en los individuos de la zona oriental y no se encuentra en los de la zona occidental (fig. 3 C).
DISCUSIÓN
Los triatomineos parecen representar un grupo genéticamente heterogéneo, con especies generales que registran mayor variación del genotipo, y especies especializadas con una variabilidad poblacional algo reducida, quizás asociada a una serie de "cuellos de botella" genéticos, seguida de una moderada selección.22 La estructura poblacional y el potencial de dispersión de las especies de triatomas son esenciales para establecer el riesgo que representan y así poder realizar una adecuada vigilancia epidemiológica y establecer un óptimo control del vector.
La información sobre la biología, dispersión y estructura poblacional de T. flavida es muy limitada. Se conoce que esta especie se ha encontrado fundamentalmente en hábitat silvestre, en cuevas y en zonas de abundante vegetación en la zona occidental del país; sin embargo, se ha reportado que durante la noche son atraídas a las casas por la luz, aunque no se han encontrado evidencias del establecimiento de colonias en las viviendas.3,4
Los triatomineos fueron en su origen silvestres, en los últimos años se han incrementado los reportes de especies conocidas de triatomineos que han establecido colonias domésticas, después de ser consideradas casi exclusivamente con hábitat silvestre,23 y se ha demostrado que estas poblaciones silvestres resultan un riesgo inminente para la transmisión de la enfermedad.10
La distribución silvestre de T. flavida, junto a su potencial transmisor de T. cruzi y otras características particulares que aumentan su peligrosidad como vector, entre ellas: eclosión durante todo el año, tiempo de deyección corto y hábitos alimentarios que favorecen una rápida dispersión de la enfermedad, así como una rápida dispersión de insectos infectados,4 hacen que sea importante estudiar la estructura genética de las poblaciones de este triatomineo.
La técnica del RAPD constituye una herramienta que permite evaluar las relaciones genéticas entre los triatomineos.6 En diferentes especies se ha utilizado para determinar la estructura genética de poblaciones, de ahí que se haya utilizado para evaluar la variabilidad genética intrapoblacional e interpoblacional de T. flavida.
Se han descrito cambios genéticos y morfológicos asociados a la adaptación de poblaciones silvestres a domésticas o de laboratorio. Al analizar el polimorfismo genético mediante la técnica de RAPD entre los individuos silvestres y la primera generación obtenida en el laboratorio, se encontró homogeneidad en el patrón de bandas obtenido. Estos resultados están en concordancia con la baja variabilidad genética encontrada al analizar diferentes poblaciones de T. flavida de este estudio.
Al analizar el polimorfismo genético entre los individuos de una misma población, se encontró homogeneidad genética entre los individuos de la población de Caimanera, de igual forma sucedió entre los 2 individuos de cada una de las poblaciones estudiadas. Resultados similares obtuvieron Ramírez y otros24 al estudiar la estructura genética de T. dimidiata en Colombia, donde encontraron homogeneidad genética entre los patrones de amplificación de cada grupo analizado (silvestre, peridomiciliado y domiciliado). El comportamiento de T. flavida encontrado en el presente estudio coincide con la baja variabilidad genética encontrada entre las poblaciones estudiadas de T. dimidiata.11
García y otros6 reportaron polimorfismo genético entre los diferentes estadios del ciclo de vida de R. prolixus (huevo, 1ro. al 5to. estadio ninfa y adulto), sin embargo en el presente estudio no se encontró diferencia entre los diferentes estadios ni entre este con el adulto, elementos que pudieran estar relacionados con la homogeneidad genética encontrada entre los individuos de una misma población de T. flavida y que no fue encontrado en R. prolixus en el estudio descrito antes.
Las estimaciones de Fst y Nm indican que entre las poblaciones del oriente y el occidente de la Península de Guanahacabibes, Pinar del Río, existe una diferencia genética pequeña por causa de las tasas de migración de alrededor de 8 individuos por generación, lo que implica un flujo genético importante entre las poblaciones y como resultado una homogeneidad genética entre estas. Este movimiento de insectos entre poblaciones separadas por distancias medias y barreras geográficas, puede ser resultado del transporte pasivo asociado a migraciones de animales.25
Aunque T. flavida presentó baja variabilidad genética, se observaron algunas diferencias genéticas entre las poblaciones del oriente y el occidente de la región de Guanahacabibes, por lo que se puede mencionar una asociación entre el polimorfismo genético y la distribución geográfica. Además, se reportaron marcadores moleculares de RAPD capaces de diferenciar las poblaciones de ambas regiones, que podrán ser analizados en futuros estudios con el fin de determinar posibles genes relacionados con las diferencias dentro de la especie.
Estos resultados están en concordancia con los obtenidos en un estudio previo, en el cual se estudiaron 6 poblaciones de T. flavida silvestres con solo 5 cebadores al azar mediante la técnica de RAPD, donde se encontró una baja heterogeneidad entre las poblaciones estudiadas.16
Las poblaciones silvestres de la mayoría de las especies presentan mayor variabilidad que las especies domiciliadas coespecíficas.7,22,24 El proceso de domiciliación de los triatomineos es de especialización que implica simplificación genética y fenotípica.27 Diferentes estudios han demostrado la reducción progresiva de la variabilidad de la secuencia de genes al comparar poblaciones silvestres y domiciliadas.7,12,24 Teniendo en cuenta estos resultados, se debe esperar que si ocurriera un proceso domiciliar en los individuos de T. flavida, estos tendrían mayor homogeneidad genética que los de las poblaciones silvestres estudiadas en este trabajo; ello favorecería los sistemas de control del vector domiciliado, lo que podría constituir el elemento fundamental para el control vectorial.
Los resultados presentados en este estudio constituyen una aproximación a la estructura genética de T. flavida. La relevancia de este vector en la transmisión de la enfermedad de Chagas es aún desconocida y los niveles de infección con Trypanosoma no han sido reportados, solo la potencialidad de transmitir la enfermedad.3 Por causa del alto flujo genético reportado en el presente estudio, este vector pudiera ser considerado como un vector potencial para la transmisión de T. cruzi, elemento a tener en cuenta en la vigilancia epidemiológica en el futuro.
La homogeneidad genética entre los individuos silvestres de T. flavida constituye un aspecto importante para la implementación de las políticas de control de este vector, que resulta en una mayor susceptibilidad al uso de insecticidas en los programas de control.
REFERENCIAS BIBLIOGRÁFICAS
1. WHO. Control of Chagas Disease. WHO Tech. Rep. Ser. 905. Geneva: World Health Organization; 2002.
1. Galvao C, Carcavallo R, Da Silva Rocha D, Jurberg J. A checklist of the current valid species of the current valid species of the subfamily Triatominae Jeannel, 1919 (Hemiptera, Reduviidae) and their geographical distribution, with nomenclature and taxonomical notes 1. Zootaxa. 2003;202:1-36.
2. Hernández Y, González R. Revisión de la subfamilia Triatominae (Hemiptera: Reduviidae) en Cuba. Boletín de Malariología y Salud Ambiental 2006;47:107-13.
3. Jiménez-Ozete H. Observaciones sobre la biología de Triatoma flavida, Neiva, 1911 en Cuba. Rev Cubana Med Trop. 1981;33:42-50.
4. Zu H, Cecere MC, Gurtler R, Kitron Y, Cohen JE. Re-establishment of local populations of vectors of Chagas disease after insecticide spraying. J Appl Ecol. 2007;44:220-7.
6. García AL, Carrasco HJ, Schofield CJ, Russell J, Frame IA, Valente SAS, et al. Random Amplified Polymorphic DNA as a tool for taxonomic studies of Triatomine Bug (Hemiptera: Reduviidae). J Med Entomol. 1998;35:38-45.
7. Jaramillo C, Montaña MF, Castro LR, Vallejo GA, Guhl F. Differentiation and genetic analysis of Rhodnius prolixus and Rhodnius colombiensis by rDNA and RAPD amplification. Mem Inst Oswaldo Cruz. 2001;96:1043-8.
8. Marcilla A, Bargues MD, Ramsey JM, Magallon-Gastelum E, Salazar-Schettino PM, Abad-Franch F, et al. The ITS-2 of the nuclear rDNA as a molecular marker for populations, species and phylogenetic relationships in Triatominae (Hemiptera: Reduviidae), vector of Chagas disease. Mol Phyl Evol. 2001;18:136-42.
9. Marcet PL, Lehmann T, Groner G, Gurtler RE, Kitron U, Dotson EM. Identification and characterization of micro satellite markers in the Chagas disease vector Triatoma infestan (Heteroptera: Reduviidae). Inf Gen Evol. 2006;6:32-7.
10. López DC, Jaramillo C, Gulh F. Estructura poblacional y variabilidad genética de Rhodnius prolixus (Hemiptera: Reduviidae) procedente de diferentes áreas geográficas de Colombia. Biomédica. 2007;27:28-39.
11. Williams JGK, Kubelik AR, Livak KJ, Rafalski JA, Tingey SV. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res. 1990;18:6531-5.
12. Borges EC, Dujardin JP, Schofield CJ, Romanha AJ, Diotauti L. Dynamics between sylvatic, peridomestic and domestic populations of Triatoma brasiliensis (Hemiptera: Reduviidae) in Ceará State, Norheastern Brazil. Acta Tropica. 2005;93:119-26.
13. Feliciangeli MD, Dujardin JP, Bastrenta B, Mazzarri M, Villegas J, Flores M, et al. Is Rhodnius robustus (Hemiptera: Reduviidae) responsible for Chagas diseases transmission in Western Venezuela? Med Int Health. 2002;7:280-7.
14. Usinger RL. The Triatominae of North and Central America and West Indies and their public health significance. Public Health Bull. 1944;288.
15. Fraga J, Rodríguez J, Fuentes O, Castex M, Fernández-Calienes A. Comparación entre cinco métodos para la extracción de ADN de Triatomíneos: Su utilización en la técnica de ADN polimórfico amplificado al azar (RAPD). Rev Cubana Med Trop. 2004;56(3):208-13.
16. Fraga J, Rodríguez J, Fuentes O, Fernández-Calienes A, Castex M. Optimization of random amplified polymorphic DNA techniques for use in genetic studies of Cuban Triatominae. Rev Inst Med Trop Sao Paulo. 2005;47:295-300.
17. Sneath PHA. Some thoughts on bacterial classification. J General Microbiol. 1957;17:201-26.
18. Pavlícek A, Hrdá S, Flegr J. Free-Tree—freeware program for construction of phylogenetic trees on the basis of distance data and bootstrap/jackknife analysis of the tree robustness. Application in the RAPD analysis of genus Frenkelia. Folia Biol (Praha). 1999;45:97-9.
19. Wright S. Evolution in Mendelian populations. Genetics. 1931;16:97-159.
20. Weir BS, Cockerham CC. Estimating F stadistics for the analysis of population´s structure. Evolution. 1984;38:1358-70.
21. Lynch M, Milligan BG. Analysis of population genetic structure with RAPD markers. Mol Ecol. 1994;3:91-9.
22. Gulh F, Schofield CJ. Population genetics and control of Triatominae. Parasit Today. 1996;12:169.
23. Schofield CJ, Diotaiuti L, Dujardin JP. The process of domestication in Triatominae. Mem Inst Oswaldo Cruz. 1999;94:375-8.
24. Ramírez C, Jaramillo CA, Delgado MP, Pinto NA, Aguilera G, Gulh F. Genetic structure of sylvatic, peridomestic and domestic population of Triatoma dimidiata (Hemiptera: Rediviidae) from and endemic zone of Boyaca, Colombia. Acta Tropica. 2005;93:23-9.
25. Dias JC. Rural resource development and its potential to introduce domestic vector into a new epidemiological situation. Rev Argent Microbiol. 1988;20:81-5.
Recibido: 11 de mayo de 2008.
Aprobado: 6 de junio de 2008.
Lic. Jorge Fraga Nodarse. Laboratorio de Biología Molecular. Departamento de Parasitología. Instituto de Medicina Tropical "Pedro Kourí". AP 601, Marianao 13. Ciudad de La Habana, Cuba. Teléf.: 53-7-202-0633. Correo electrónico: fraga@ipk.sld.cu














