Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760
Rev Cubana Med Trop vol.65 no.2 Ciudad de la Habana abr.-jun. 2013
ARTÍCULO ORIGINAL
Aislamiento de Leptospira borgpetersenii de fuentes de agua en Argentina
Isolation of Leptospira borgpetersenii in water sources in Argentina
Dra. Silvina Francois Barbagelata,I Dra. Bibiana Brihuega Fernández,II Lic. Sylvia Grune Loffler,II Med. Vet. Virginia Gattarello Marcos,I Med. Vet. David Correa Pérez,I Lic. Jessica Petrakovsky Melillo,III Med. Vet. Catalina Gualtieri Serragatta,I Dra. Mirta Arestegui De LucaI
I Universidad Nacional de Rosario. Casilda, Santa Fe, Argentina.
II Organización Mundial de Sanidad Animal, Dirección de Laboratorios-Servicio Nacional de Sanidad Animal, Instituto de Patobiología, Centro de Investigaciones en Ciencias Veterinarias y Agropecuarias, Instituto Nacional de Tecnologia Agropecuaria. Castelar. Buenos Aires, Argentina.
III Organización Mundial de Sanidad Animal, Dirección de Laboratorios-Servicio Nacional de Sanidad Animal. Buenos Aires, Argentina.
RESUMEN
Introducción: las especies del género Leptospira son los agentes causales de la leptospirosis, enfermedad considerada como la zoonosis de mayor distribución en el mundo. En Argentina reviste carácter endémico. La provincia de Santa Fe registra el mayor número de casos humanos. Desde mediados de la década de 1980, las especies patógenas de leptospiras aisladas de animales y humanos fueron diferenciadas sobre la base de estudios de hibridización ADN-ADN, surgiendo nuevas especies: L. interrogans, L. kirschneri, L. weilii, L. noguchii, L. borgpetersenii, L. santarosai, L. meyeri, L. inadai, L. faineri y L. alexanderi.
Objetivo: aislar y caracterizar mediante métodos moleculares, leptospiras de fuentes de agua que vierten en un canal que atraviesa la ciudad de Casilda, Santa Fe, en Argentina.
Métodos: se sembraron 6 muestras de agua de las vertientes al canal, previa filtración con filtros Millipore de 0,22 µm, en medios EMJH y Fletcher para aislamiento de leptospiras. Se incubaron en estufa a 30 °C durante 15 días, se observaron semanalmente mediante microscopia de campo oscuro. Se implementó la técnica de reacción en cadena de la polimerasa bajo las condiciones específicas (Sugathan, 2005), con dos juegos de cebadores (Gravekamp, 1993), que permiten detectar la presencia de ADN de leptospiras patógenas. La técnica molecular utilizada para genotipificar fue multiple-locus variable-number tandem repeats analysis (MLVA).
Resultados: se obtuvieron 5 aislamientos de Leptospira spp., de los cuales 2 resultaron positivos a la reacción en cadena de la polimerasa, determinando que se trataba de leptospiras patógenas. Mediante la genotipificación por MLVA se pudo observar que uno de los aislamientos patógenos mostró un patrón correspondiente a la especie Leptospira borgpetersenii, no siendo identificable la otra cepa.
Conclusiones: en la ciudad del estudio, que tiene alrededor de 40 000 habitantes, se logró identificar por primera vez una cepa de Leptospira borgpetersenii de fuentes de aguas urbanas, con el peligro potencial que esto representa para la población humana y animal.
Palabras clave: Leptospira borgpetersenii, agua, Santa Fe, Argentina.
ABSTRACT
Introduction: the Leptospira genus species are causative agents of leptospirosis, a disease that is considered the most widely spread zoonotic disease worldwide. In Argentina, leptospirosis is endemic and Santa Fe province has the highest number of human cases. Since mid-1980's, the pathogenic leptospira species isolated from animals and humans have been differenciated through DNA-DNA hybridization tests, resulting in new species: L. interrogans, L. kirschneri, L. weilii, L. noguchii, L. borgpetersenii, L. santarosai, L. meyeri, L. inadai, L. faineri y L. alexanderi.
Objectives: to isolate and to characterize by molecular test leptospiras from water poured into a channel that runs through Casilda City in Santa Fe Province, Argentina.
Methods: six samples of water from the channel were cultured after having been filtered through 0.22 µm, Millpore filtres in EMJH and Fletcher media to isolate leptospires. They were incubated at 30 °C for 15 days, and weekly observed through dark field microscopy. Polymerase chain reaction assay was used under specific conditions (Sugathan, 2005), with two sets of primers (Gravekamp, 1993), to determine whether the isolates were pathogenic. The molecular technique for genotyping was Multiple-Locus Variable-number tandem repeats Analysis (MLVA).
Results: five Leptospira spp. isolates were obtained of which 2 were positive to PCR, all of which determined that they were pathogenic leptospiras. MLVA genotyping allowed the observation of a pattern similar to that of L. borgpetersenii species in one pathogenic isolates, but the other isolate was not identified.
Conclusions: in the City where the study was carried out, with a population of about 40,000 inhabitants, a L. borgpetersenii species was identified for the first time in urban water sources, with the potential risk that it may pose for human and animal populations.
Key words: Leptospira borgpetersenii, water, Santa Fe, Argentina.
INTRODUCCIÓN
Las especies del género Leptospira son los agentes causales de la leptospirosis, enfermedad considerada como la zoonosis de mayor distribución en el mundo.1 Esta enfermedad en Argentina reviste carácter endémico. Según datos internacionales, desde mediados de la década de los ochenta, las especies patógenas de leptospiras aisladas de animales y humanos fueron diferenciadas sobre la base de estudios de hibridización ADN-ADN, surgiendo nuevas especies: L. interrogans, L. kirschneri, L. weilii, L. noguchii, L. borgpetersenii, L. santarosai, L. meyeri, L. inadai, L. faineri y L. alexanderi.1 Mayoritariamente, las infecciones producidas por estas bacterias en humanos y animales, son causadas por cepas de L. interrogans y L. borgpetersenii, las que varían en su distribución en la naturaleza y cuentan con modos diferentes de transmisión.2 En los animales de producción, la leptospirosis ocasiona grandes pérdidas económicas por agalactia, abortos y muerte perinatal. Por su parte, en los humanos, la infección puede ser transmitida de manera directa o indirectamente mediante la orina de un animal infectado, causando diversos cuadros clínicos agudos o sistémicos, caracterizados por cefaleas, fiebre elevada, meningitis e ictericia. Si el enfermo no es tratado temprano, la enfermedad puede progresar a cuadros graves que lo pueden llevar hasta la muerte.1 En el medio ambiente, el agua representa uno de los vehículos más importantes para la transmisión indirecta. La exposición a las leptospiras puede estar asociada a la aparición de brotes en temporadas de lluvias e inundaciones en zonas urbanas con condiciones sanitarias deficientes, a ocupaciones laborales que revisten un alto riesgo de contagio como los trabajadores rurales, veterinarios, etc. Recientemente la exposición recreacional asociada con el turismo en áreas endémicas ha provocado la existencia de brotes importantes después de eventos atléticos.3,4 El agua es un factor clave para la supervivencia y la transmisión de las leptospiras. Si presenta un valor de pH entre 7 y 8, esto facilita que las especies patógenas puedan sobrevivir en ella por varias semanas.5
La provincia de Santa Fe, registra el mayor número de casos humanos diagnosticados en Argentina. Este hecho se debe a que posee características medioambientales que son propicias para el desarrollo y la supervivencia de las leptospiras, como un clima templado-húmedo, altos índices pluviométricos y terrenos bajos e inundables. La ciudad de Casilda, como otras áreas de la provincia, presenta las mismas características. Además, está surcada por un canal en el que desembocan las cañerías colectoras de desagües pluviales y el agua que drena desde zanjas colectoras ubicadas en los distintos barrios de la ciudad. En épocas de lluvias abundantes, verano y otoño, el sistema de drenaje es sobrepasado en su capacidad de eliminación. Esto provoca la inundación de la zona, que está habitada por un elevado número de personas en estrecho contacto con pequeños basureros (a pesar de contar con sistemas de recolección semanal de residuos), aguas estancadas, gran cantidad de perros callejeros y roedores. El objetivo del presente estudio consistió en aislar y caracterizar mediante métodos moleculares, leptospiras de las fuentes de agua que vierten en el canal que atraviesa la ciudad de Casilda, Santa Fe, en Argentina.
MÉTODOS
Toma de muestras
En el mes de octubre de 2011 se analizó el largo del canal y sus vertientes mediante el uso de elementos cartográficos. Se midió el pH del agua de las zanjas que desembocan en el canal y se seleccionaron para la toma de muestras, aquellas con pH superior a 7,5. Se procedió a tomar 6 muestras de un volumen equivalente a 100 mL de agua cada una, correspondientes a distintas zanjas, introduciendo un frasco colector estéril a 15 cm de profundidad en la parte de la orilla. Las muestras se transportaron de inmediato al laboratorio, donde se procedió a su cultivo.
Procedimiento para el aislamiento de leptospiras
Cultivo bacteriológico
Las muestras de agua se sembraron directamente por filtración con filtros Millipore de 0,22 µm, en medios EMJH (Difco) y Fletcher (Difco) para aislamiento de leptospiras, adicionados con 5-fluorouracilo (5-FU, 200 µg/mL), 0,1 mL por 5 mL de medio, según la metodología descrita.6 Posteriormente, los tubos sembrados se incubaron en estufa a 30 °C durante 15 días, observándolos semanalmente mediante microscopia de campo oscuro. Los aislamientos de leptospiras fueron enviados al Laboratorio de Referencia para Leptospirosis de la OIE, DILAB-SENASA, para determinar mediante PCR (polymerase chain reaction), si se trataba de leptospiras patógenas.
Determinación de patogenicidad por PCR en los aislamientos de leptospiras
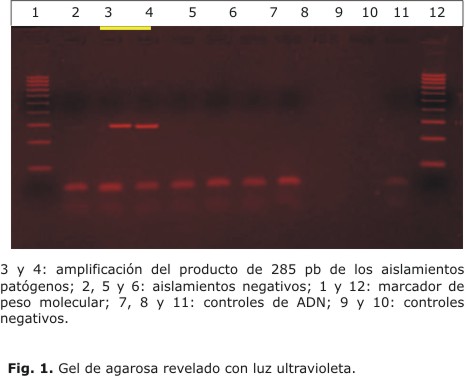
Para la extracción del ADN se empleó un kit comercial con columnas de extracción. La PCR se llevó a cabo bajo las condiciones específicas.7 En la PCR se utilizaron dos juegos de cebadores, que permiten detectar la presencia de ADN de leptospiras patógenas.8 El par de cebadores G1 5'-CTG AAT CGC TGT ATA AAA GT y G2 5'-GGA AAA CAA ATG GTC GGA AG, tamaño del producto 285 bp presente en todas la cepas de leptospiras patógenas exceptuando a L. kirschneri, y el par: B64I 5'-ACT AAC TGA GAA ACT TCT AC y B64II 5'-TCC TTA AGT CGA ACC TAT GA, tamaño del producto 563 bp que identifica a esta última.
Tipificación genotípica
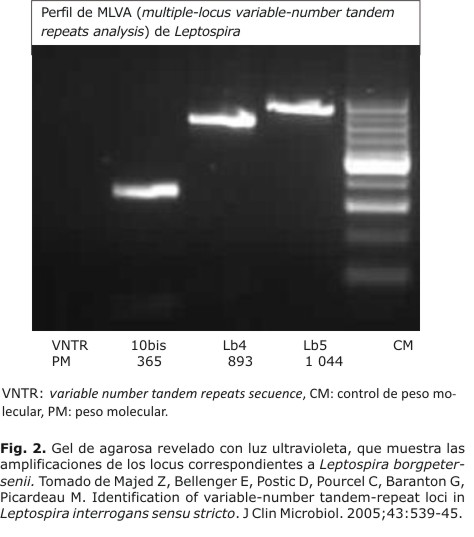
Los aislamientos caracterizados previamente como leptospiras patógenas se enviaron al Centro de Referencia de Leptospirosis de la OIE, del INTA Castelar, Buenos Aires, Argentina; donde fueron genotipificados por la técnica de multiple-locus variable-number tandem repeats analysis (MLVA), según la metodología de Majed Z y otros para amplificar fragmentos del genoma de la especie patógena Leptospira interrogans sensu stricto. Los locus flanqueados para discriminar entre cepas fueron, VNTRS: 4, 7, 9, 10, 19, 23 y 31, descritos por Majed Z y otros, así como Pavan ME y otros.9,10 Para las restantes especies patógenas L. borgpetersenii y L. kirschneri, los oligonucleótidos descritos en los locus flanqueados fueron: VNTR-Lb4, VNTR-Lb5, VNTR-4bis, VNTR-7bis, VNTR-10bis.11 Cada reacción contenía tampón PCR (20 mM Tris-HCl, pH 8,4; 50 mM KCl), 200 µM de cada dNTP, 2 mM MgCl2, 1,25 U de Taq DNA polymerasa (Invitrogen), 2 µM de cada cebador, y 5 µL de templado de ADN en un volumen final de 50 µL.
Las PCR fueron llevadas a cabo en un MiniCycler PTC-150 (MJ Research) de la forma siguiente: 94 °C por 5 min, siguiendo 35 ciclos de desnaturalización a 94 °C por 30 s, templado de 55 °C por 30 s y extensión a 72 °C por 90 s, con un ciclo final de 72 °C durante 10 min. Cada muestra amplificada (14 µL) fue sometida a electroforesis en un gel de agarosa 2,2 % en tampón TAE (40 mM Tris-Acetate, 1 mM EDTA) con 0,2 µg/mL de bromuro de etidio, a 100 V por 50 min. Las bandas de DNA amplificadas se visualizaron con luz UV (DynaLight, Labnet). El tamaño del amplicón fue estimado usando CienMarker (Biodynamics).
RESULTADOS
Del total de las muestras de agua cultivadas, se obtuvieron 5 aislamientos de Leptospira spp. Mediante la PCR, se obtuvieron 2 aislamientos positivos, en los cuales se observó la banda de 285 pb, presente en todas la cepas de Leptospira patógenas, exceptuando a L. kirschneri (Fig. 1).
Mediante la genotipificación por MLVA se pudo observar que uno de los aislamientos patógenos mostró un patrón correspondiente a la especie L. borgpetersenii (Fig. 2). El resto no mostró un perfil identificable.
DISCUSIÓN
En Argentina existen pocos reportes de estudios que hayan utilizado el cultivo bacteriológico de muestras de agua para el aislamiento de leptospiras.12 Este hecho quizá se deba a lo complejo que resulta el aislamiento de estas, cuando las muestras están altamente contaminadas. En este estudio se determinó que la metodología empleada para aislar Leptospira a partir de fuentes de agua contaminada resultó ser una práctica eficaz. Se observó además, que estas bacterias se desarrollan y son capaces de sobrevivir en aguas servidas con valores de pH que van de la neutralidad al lado alcalino de la escala. El porqué de la supervivencia de las cepas aisladas de muestras de aguas tomadas a 15 cm de profundidad en la zona de las orillas, quizá podría explicarse por razones bióticas y abióticas del micro-medioambiente, que favorecen la supervivencia de la bacteria, como son la temperatura del agua, el pH, la presencia de oxígeno y la disponibilidad de nutrientes.
La conservación de leptospiras patógenas en agua y suelo es uno de los problemas importantes a investigar en los estudios geográficos-epidemiológicos, dado que la mayor parte de los brotes de leptospirosis humana que han sido reportados en todo el mundo, son de casos relacionados con la transmisión acuática y al aumento de la exposición a los entornos contaminados.13 En las investigaciones epidemiológicas, los ensayos moleculares constituyen metodologías diagnósticas precisas, gracias a su alto poder de discriminación, reproducibilidad y fácil interpretación.14 En este estudio, los ensayos basados en técnicas de PCR utilizados, resultaron herramientas útiles tanto para caracterizar como para identificar las leptospiras patógenas.
En la ciudad del estudio, que tiene alrededor de 40 000 habitantes, la metodología genotípica implementada permitió identificar por primera vez una cepa de Leptospira borgpetersenii de fuentes de agua urbanas, con el peligro potencial que esto representa para la población humana y animal. Existen numerosos reportes que demuestran la virulencia de esta especie, así como los casos graves de leptospirosis en humanos que ha causado en los últimos años, en diversas partes del mundo.14-16 Estudios realizados sobre el genoma de L. borgpetersenii han revelado que este ha sufrido reducciones en cuanto a genes de adaptación al medio ambiente y que al parecer está evolucionando a depender de manera estricta de un ciclo de transmisión hospedador-hospedador.17 Este hecho es importante de considerar desde el punto de vista epidemiológico en una ciudad densamente poblada como Casilda y, además, conduce a nuevas investigaciones acerca de distintos aspectos de la cepa aislada.
Finalmente, los resultados han permitido conocer la existencia local de una especie patógena del género Leptospira, lo que significa un gran avance para entender la epidemiología de la leptospirosis en esta ciudad y para brindar a las autoridades sanitarias la posibilidad de realizar las tareas correspondientes de prevención y control. No hay que olvidar que la leptospirosis es una enfermedad a controlar porque sus reservorios silvestres impiden su erradicación.
REFERENCIAS BIBLIOGRÁFICAS
1. Stanchi N, Martino P, Gentilini E, Reinoso E, Echeverría M, Leardini N, et al. Microbiología Veterinaria. Buenos Aires: Inter-Médica; 2010.
2. Sakamoto M, Kato T, Sato F, Yoshikawa K, Yoshida M, Shiba K, et al. A case of Leptospirosis caused by Leptospira borgpetersenii serovar sejroe infected in Bali Island, Indonesia. Kansenshogaku Zasshi. 2005;79(4):294-8.
3. Morgan J, Bornstein SL, Karpati AM. Outbreak of Leptospirosis among Triathlon participants and community residents in Springfield, Illinois, 1998. Clin Infect Dis. 2002;34:1593-9.
4. Sejvar J, Bancroft E, Winthrop K. Leptospirosis in "Eco Challenge" athletes, Malasysian Borneo, 2000. Emerg Infect Dis. 2003;9:702-7.
5. Schereier S, Doungchawee G, Triampo D, Wangroongsarb P, Hartskeerl RA, Triampo W. Development of a magnetic bead fluorescence microscopy immunoassay to detect and quantify Leptospira in environmental water samples. Acta Trop. 2012;122(1):119-25.
6. Giraldo de León G, Orrego Uribe A, Santa Cruz M, Yepes E. Leptospirosis. Las aguas de la explotación porcina como vehículo de la leptospira, en la zona central Cafetera de Colombia. Arch Med Vet. 2002;34(1):79-87.
7. Sugathan S, Varghese TP. Multiplex PCR on leptospiral isolates from Kolenchery, Kerala, India. Indian. J Med Microbiol. 2005;23(2):114-6.
8. Gravekamp C, Van De Kemp H, Franzen M, Carrington D, Schoone GJ, Van Eys GJJM. Detection of seven species of pathogenic leptospires by PCR using two sets of primers. J Gen Microbiol. 1993;139(8):1691-700.
9. Majed Z, Bellenger E, Postic D, Pourcel C, Baranton G, Picardeau M. Identification of variable-number tandem-repeat loci in Leptospira interrogans sensu stricto. J Clin Microbiol. 2005;43:539-45.
10. Pavan ME, Cairó F, Pettinari MJ, Samartino L, Brihuega B. Genotyping of Leptospira interrogans strains from Argentina by multiple-locus variable-number tandem repeat Analysis (MLVA). Comp Immunol Microbiol Infect Dis. 2011;34(2):135-41.
11. Salaün L, Mérien F, Gurianova S, Baranton G, Picardeu M. Application of multilocus variable-number tandem-repeat analysis for molecular typing of the agent of Leptospirosis. J Clin Microbiol. 2006;44(11):3954-62.
12. Brihuega B, Auteri C, Romero G, Samartino L. Aislamiento de una cepa patógena de leptospira de un río urbano: Respuesta frente a quinolonas fluoradas. Rev Med Vet. 2006;87(4):144-6.
13. Yang W, Pang J, Li C. An investigation on the distribution of Leptospira interrogans in water and soil in southwest of Yunnan Province. Zhonghua Liu Xing Bing Xue Za Zhi. 1994;15(5):289-91.
14. Moreno N, Agudelo-Flórez P. Application of conventional and multiplex PCR assays for identification of isolates of Leptospira spp. in Colombia. Rev Peru Med Exp Salud Pública. 2010;27(4):548-56.
15. Monte LG, Conceiçáo FR, Coutinho ML, Seixas FK, da Silva EF, Vasconcellos FA, et al. Monoclonal antibodies against the leptospiral immunoglobulin-like proteins A and B conserved regions. Comp Immunol Microbiol Infect Dis. 2011;34(5):441-6.
16. Diniz JA, Félix SR, Bonel-Raposo J, Seixas Neto AC, Vasconcellos FA, Grasmann AA, et al. Highly virulent Leptospira borgpetersenii strain characterized in the Hamster model. Am J Trop Med Hyg. 2011;85(2):271-4.
17. Bulach DM, Zuerner RL, Wilson P, Seemann T, Mc Grath A, Cullen PA, et al. Genome reduction in Leptospira borgpetersenii reflects limited transmission potential. Proc Natl Acad Sci USA. 2006;103(39):14560-5.
Recibido: 13 de julio de 2012.
Aprobado: 24 de enero de 2013.
Silvina Francois Barbagelata. Servicio de Diagnóstico de Leptospirosis de la Facultad de Ciencias Veterinarias, Universidad Nacional de Rosario. Casilda, Santa Fe, Argentina. Correo electrónico: silvinafrancois@yahoo.com.ar