Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760
Rev Cubana Med Trop vol.65 no.2 Ciudad de la Habana abr.-jun. 2013
ARTÍCULO ORIGINAL
Mutaciones puntuales en el gen de la dihidropteroato sintetasa de Plasmodium falciparum en Bolívar, Colombia
Point mutations in the dihydropteroate synthetase gene of Plasmodium falciparum in Bolivar, Colombia
MSc. Dary Luz Mendoza Meza, Biol. Luis Acuña Cantillo, Biol. Fernando De la Cruz López, MSc. Alfredo Lagares Guzmán, Dra. Lourdes Varela Prieto
Universidad del Atlántico. Barranquilla, Colombia.
RESUMEN
Introducción: la resistencia antimalárica dificulta el control del paludismo en Colombia. La vigilancia molecular de mutaciones puntuales en blancos terapéuticos es fundamental en el estudio de la resistencia a los antimaláricos.
Objetivo: identificar mutaciones puntuales en el gen de la dihidropteroato sintetasa de Plasmodium falciparum (pfdhps), asociadas con resistencia in vitro a sulfadoxina.
Métodos: la fuente de ADN de Plasmodium falciparum consistió en láminas de gota gruesa de 55 individuos con infección malárica, reportados en el departamento de Bolívar, Colombia. El ADN se extrajo con solución de Chelex-100 al 5 %. Las mutaciones se identificaron mediante PCR-RFLP (polymerase chain reaction-restriction fragment length polymorphism) y secuenciación.
Resultados: 17 muestras (31 %) amplificaron un fragmento de 438 pb de pfdhps. La PCR-RFLP mostró frecuencia del genotipo mutante G-437 en 65 % de los amplificados, el mixto A/G-437 en 29 % y el silvestre A-437 en 6 %. Los alelos mutantes G-437, F-436 y el alelo silvestre K-540 se identificaron en todas las muestras secuenciadas.
Conclusiones: este es el primer reporte de mutaciones puntuales en el gen dhps de Plasmodium falciparum en el departamento de Bolívar, Colombia, lo cual contribuye al conocimiento de la resistencia a los antimaláricos en el Caribe colombiano.
Palabras clave: Plasmodium falciparum, pfdhps, mutaciones puntuales, gota gruesa.
ABSTRACT
Introduction: antimalarial drug resistance hinders the control of malaria in Colombia. The molecular surveillance of point mutations in therapeutic targets is essential in the study of antimalarial drugs.
Objective: to identify point mutations at dihydropteroate synthetase gene of Plasmodium falciparum (pfdhps) associated with sulfadoxine resistance.
Methods: source of P. falciparum DNA was blood thick smears of 55 individuals with malaria infection, reported in Bolivar, Colombia. The DNA was extracted with 5 %. Chelex-100 solution. The mutations were identified by PCR-RFLP and sequencing.
Results: seventeen samples (31 %) amplified a 438 bp fragment of pfdhps. The PCR-RFLP (polymerase chain reaction-restriction fragment length polymorphism) showed a frequency of mutant genotype (G-437) in 65 % of amplicons, mixed genotype (A/G-437) in 29 % and wild genotype (A/G-437) in 6 %. The mutant alleles G-437, F-436 and the wild allele K-540 were identified in all sequenced samples.
Conclusions: this is the first report of point mutations in the P. falciparum dhps gene in Bolivar, Colombia. This result contributes to the knowledge of antimalarial drug resistance in the Colombian Caribbean region.
Key words: Plasmodium falciparum, pfdhps, point mutations, thick smears.
INTRODUCCIÓN
En Colombia cerca de 25 millones de personas se encuentran en riesgo de adquirir malaria, constituyéndose en un problema de salud pública. Aproximadamente, 48 % de las muertes por malaria, en Colombia, son ocasionadas por Plasmodium falciparum, 35 % por Plasmodium vivax, 15 % por malaria mixta y 2 % por Plasmodium malariae; el P. falciparum es la especie causante de la forma más severa de la enfermedad.1 La resistencia del P. falciparum a los medicamentos antimálaricos es uno de los factores asociado con el incremento de casos y muertes por malaria.2
La sulfadoxina es una sulfonamida, que junto con la pirimetamina (Fansidarâ), se usó en combinación con cloroquina, en Colombia, desde la década de los setenta, para el tratamiento en primera línea de la malaria por P. falciparum; los primeros reportes de resistencia a pirimetamina-sulfadoxina en este país datan de los años ochenta,2 lo cual obligó a cambiar el esquema de tratamiento en las zonas geográficas con reportes de falla terapéutica. Mutaciones puntuales en el gen de la dihidropteroato sintetasa de Plasmodium falciparum (pfdhps), asociadas con resistencia a sulfadoxina, se han identificado en varias poblaciones colombianas;3-5 sin embargo, en el Caribe colombiano los estudios de resistencia y falla terapéutica a este medicamento son escasos, existiendo pocos reportes de prevalencia de mutaciones en pfdhps. El objetivo de este estudio radicó en identificar mutaciones en el gen pfdhps, a partir de casos de malaria reportados en el departamento de Bolívar.
MÉTODOS
Muestra: láminas de gota gruesa de 55 individuos infectados con malaria no complicada por P. falciparum, donadas por el Laboratorio de Salud Pública del departamento de Bolívar, correspondientes a casos reportados entre marzo 2010 y septiembre de 2011, en los municipios de Tiquisio 44%, Achi 18 %, Montecristo 11 %, Arenal sur 9 %, Santa Rosa del Sur 5,4 %, Cartagena 5,4 %, San Jacinto del Cauca 3,6 %, Rioviejo 1,8 % y San Pablo 1,8 %. Todos los pacientes que donaron muestras para el estudio firmaron consentimiento informado, previa explicación de los objetivos de la investigación, sus ventajas y exposición con riesgo mínimo, según resolución 8430 del 4 de octubre 1993 del Ministerio de Salud de la República de Colombia.6
Extracción de ADN: la extracción del ADN desde las láminas de gota gruesa se realizó siguiendo el método descrito por Van Der Zanden,7 con algunas modificaciones; las láminas se lavaron con xilol y etanol absoluto, luego se rasparon con un bisturí estéril, la sangre se recolectó en tubos de polipropileno de 1.5 mL y se trató con 50 µL de solución de lisis (5 % de Chelex-100; 0,01 % de SDS; 1 % de Tween-20 y 1 % de Brij-97), seguido por incubación y calentamiento a 90 °C durante 45 min. La mezcla se centrifugó a 11 000 g por 10 min, el sobrenadante se utilizó directamente en la PCR (polymerase chain reaction).
PCR-RFLP (polymerase chain reaction-restriction fragment length polymorphism): se realizó amplificación de un fragmento de 438pb del gen pfdhps, siguiendo protocolo reportado,8 con algunas modificaciones. Se utilizaron los iniciadores, sentido K (TGCTAGTGTTATAGATATAGGATGAGCATC) y antisentido K/(CTATAACGAGGTATTGCATTTAATGCAAGAA). En un volumen final de 50 µL de reacción, se usaron 0,2 mM de dNTP (Promega), 0,25 µM de cada iniciador, 1,25 U de Taq ADN polimerasa (Promega), 2 mM de MgCl2 (Promega) y 10 µL de ADN extraído. El programa de amplificación consistió de un ciclo inicial de desnaturalización a 94 °C durante 3 min, 40 ciclos a 94 °C durante 1 min, a 45 °C durante 1 min y a 72 °C durante 1 min, y una extensión final a 72 °C durante 10 min.
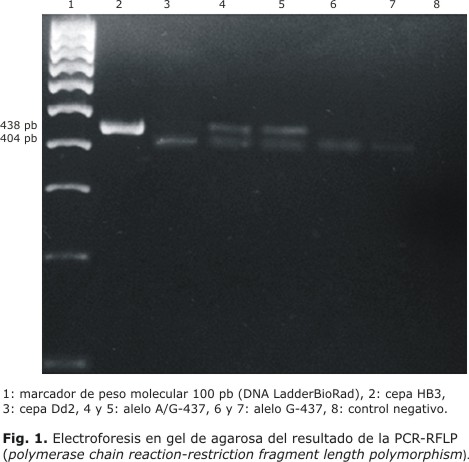
En la PCR se utilizaron como controles, 50 ng de ADN de 4 cepas de referencia de P. falciparum, donadas por el Laboratorio de Bioquímica del Instituto Nacional de Salud de Colombia: 3D7 (África) y Dd2 (Indochina-Lao) (alelo mutante G-437); también, HB3 (Honduras) y 7G8 (Brasil) (alelo silvestre A-437). Los iniciadores K y K/ no hibridan en secuencias complementarias del gen dhps de P. vivax, por lo que el ADN de este parásito se usó como control negativo en la PCR. La amplificación del fragmento se comprobó por electroforesis en gel de agarosa 1,5 % (SeaKem® LE Agarose, Cambrex BioScience Rockland, Inc.). Los amplificados fueron digeridos con la endonuclasa de restricción Ava II (Eco 471 FastDigest®, Fermentas) según las condiciones del fabricante. Ava II digiere al tipo mutante (G-437) en un sitio (GGA/TCC), generando un fragmento de 404 pb y otro de 34 pb; en el tipo silvestre (A-437), Ava II no tiene sitio de corte por lo que se obtiene un solo fragmento de 438 pb. Cuando está presente el tipo mixto (A/G-437) se obtienen fragmentos de 438, 404 y 34 pb.
Secuenciación: los amplificados que mostraron una buena resolución en la electroforesis fueron analizados mediante la técnica de secuenciación por extensión del iniciador, en ambos sentidos, con el equipo 3730XL DNA Analyzer (Applied Biosystems™), usando el kit comercial BigDye® Terminator (Applied Biosystems™) y los iniciadores K y K/. Las secuencias consensos se derivaron de las secuencias obtenidas de cada hebra de ADN, utilizando el programa MEGA 5.05. Con la herramienta BLASTN (http://www.blast.ncbi.nlm.nih.gov), se determinó el porcentaje de similitud y la posición de las secuencias obtenidas con respecto al gen dhps de P. falciparum. La presencia o ausencia de las mutaciones fue determinada por análisis de alineamiento con el algoritmo ClustalW (http://www.ebi.ac.uk/Tools/msa/clustalw2/) usando las secuencias de aminoácidos de DHPS de las cepas de P. falciparum: It.D12, 3D7, FCB1, Tak9/96 y V1/S. Finalmente, todas las secuencias nucleotídicas generadas en este estudio fueron introducidas en el Banco de Genes (GenBank) con los números de acceso del KC473523 al KC473526 (tabla). Los datos se organizaron en Excel 2007-Microsoft Office® para establecer una distribución de frecuencias de las variantes silvestres y mutantes.
RESULTADOS
De las 55 muestras, 17 (31 %) amplificaron el producto de 438 pb de pfdhps, el cual va desde el codón 425 al 571. La baja positividad de la PCR fue atribuida a la calidad de las láminas de gota gruesa, evidenciada por la presencia de artefactos, exceso de coloración y aceite de inmersión; estudios previos argumentan que estos factores influyen en la pérdida de ADN del parásito y son cruciales en la PCR.9,10 Por PCR-RFLP, 11 de las 17 muestras (65 %) presentaron el genotipo mutante G-437, 29 % (5/17) el genotipo mixto A/G-437 y 6 % (1/17) el genotipo silvestre A-437 (Fig. 1).
De los productos de PCR, 4 mostraron concentración y calidad óptima del ADN para el análisis por secuenciación. Las secuencias de nucleótidos obtenidas mostraron porcentajes de similitud mayores que 90 %, al compararlas con el gen pfdhps de cepas reportadas en el GenBank. El análisis de alineamiento múltiple con las secuencias aminoacidicas de DHPS (tabla), mostró la presencia de una mutación doble, correspondiente a los codones F-436 y G-437, en los 4 productos de PCR secuenciados, no detectándose mutación en la posición 540 (Fig. 2).
DISCUSIÓN
Estos resultados son consistentes con lo reportado en otras zonas geográficas de Colombia; por ejemplo, en Córdoba (región Caribe) y en Antioquia (región Andina), las prevalencias de la mutación G-437 son de 90 % y 85 %, respectivamente;3,5 otras regiones reportan prevalencias más bajas para esta mutación, como Chocó (región Pacífica) y la Amazonia colombiana, con 66 % y 69 %, respectivamente.4 Desde el punto de vista epidemiológico, estos hallazgos son relevantes porque no existen antecedentes de prevalencia de mutaciones puntuales en el gen pfdhps en el departamento de Bolívar. Este constituye el primer reporte, lo cual contribuye al conocimiento de la resistencia a los antimaláricos en el Caribe colombiano.
REFERENCIAS BIBLIOGRÁFICAS
1. Padilla JC, Álvarez G, Montoya R, Chaparro P, Herrera S. Epidemiology and control of malaria in Colombia. Mem Inst Oswaldo Cruz, Rio de Janeiro. 2011;106:114-22.
2. Espinal CA, Uribe LM, Eslava A, Rodríguez ME. Resistencia del Plasmodium falciparum a la combinacion sulfa-pirimetamina: descripción de los tres primeros casos en Colombia. Biomédica. 1981;1:213-7.
3. Galindo JA, Cristiano FA, Knudson A, Nicholls RS, Guerra AP. Mutaciones puntuales en los genes dhfr y dhps de Plasmodium falciparum de tres regiones endémicas para malaria en Colombia. Biomédica. 2010;30:56-64.
4. Corredor V, Murillo C, Echeverry DF, Benavides J, Pearce RJ, Roper C, et al. Origin and dissemination across the Colombian Andes mountain range of sulfadoxine - pyrimethamine resistance in Plasmodium falciparum. Antimicrob Agents Chemother. 2010;54:3121-5.
5. Villa A, Carmona-Fonseca J, Benito A, Martínez A, Maestre A. Status of dhps and dhfr genes of Plasmodium falciparum in Colombia before artemisinin based treatment policy. Infectio. 2012;16:37-44.
6. Ministerio de Salud, República de Colombia. Resolución 8430 de 1993: por la cual se establecen las normas científicas, técnicas y administrativas para la investigación en salud [citado 10 Ene 2013]. Disponible en: http://www.minsalud.gov.co/Normatividad/RESOLUCION%208430%20DE%201993.pdf
7. Van Der Zanden AG, Te Koppele-Vije EM, Vijaya Bhanu N, Van Soolingen D, Schouls LM. Use of DNA extracts from Ziehl-Neelsen-stained slides for molecular detection of rifampicin resistance and spoligotyping of Mycobacterium tuberculosis. J Clin Microbiol. 2003;41:1101-8.
8. Duraisingh M, Curtis J, Warhurst, D. Plasmodium falciparum: Detection of polymorphisms in the dihydrofolate reductase and dihydropteroate synthetase genes by PCR and restriction digestion. Exp Parasitol. 1998;89:1-8.
9. Kimura M, Kaneko O, Inoue A, Ishii A, Tanabe K. Amplification by polymerase chain reaction of Plasmodium falciparum DNA from Giemsa-stained thin blood smears. Mol Biochem Parasitol. 1995;70:193-7.
10. Edoh D, Steiger S, Genton B, Beck HP. PCR amplification of DNA from malaria parasites on fixed and stained thick and thin blood films. Trans R Soc Trop Med Hyg. 1997;91:361-3.
Recibido: 27 de agosto de 2012.
Aprobado: 8 de febrero de 2013.
Dary Luz Mendoza Meza. Programa de Química. Universidad del Atlántico. Km 7 vía Puerto Colombia. Barranquilla, Colombia. Correo electrónico: darymendoza@mail.uniatlantico.edu.co