Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Medicina Tropical
versión impresa ISSN 0375-0760versión On-line ISSN 1561-3054
Rev Cubana Med Trop vol.68 no.1 Ciudad de la Habana abr. 2016
Rev Cubana Med Trop 2016;68(1)
ARTÍCULO ORIGINAL
Relación genética del virus dengue 1 aislado en Cuba durante la epidemia de 1977
Genetic relationship of dengue virus 1 isolated in Cuba, 1977
Gisell Díaz Gutierrez, Rosmari Rodríguez-Roche, María G. Guzmán Tirado
Instituto de Medicina Tropical "Pedro Kourí". La Habana, Cuba.
RESUMEN
Introducción: el dengue es una enfermedad aguda causada por cuatro serotipos del virus del dengue (DENV-1 al 4) y transmitida al hombre por el mosquito Aedes aegypti. En Cuba, la primera epidemia de dengue con confirmación de laboratorio ocurrió en 1977, identificándose al DENV-1 como agente causal. Se plantea que el virus se dispersó en varios países de Latinoamérica y el Caribe partir de su introducción en Jamaica en 1977.
Objetivos: realizar la caracterización molecular y determinar la relación genética del DENV-1 aislado en Cuba, 1977.
Métodos: a partir de un aislamiento de DENV-1 obtenido en Cuba en el año 1977 conservado en el Banco de Cepas del Laboratorio Nacional de Referencia de Arbovirus (Instituto de Medicina Tropical “Pedro Kourí”) se realizó la amplificación y secuenciación nucleotídica del gen de la envoltura con cebadores específicos. A partir de la secuencia obtenida se construyó un árbol filogenético de Máxima Verosimilitud, utilizando secuencias de cepas de DENV-1 circulantes a nivel mundial.
Resultados: el aislamiento de DENV-1 correspondiente a la epidemia cubana de 1977, se agrupó dentro del genotipo I, relacionándose genéticamente con la cepa de Referencia Hawaii/1943. Es significativo que, el resto de los aislamientos de Latinoamérica de ese mismo período pertenecen al genotipo V, reconocido como el genotipo Americano/Africano.
Conclusiones: los resultados del presente trabajo indican que durante la epidemia cubana de 1977 circuló una cepa de DENV-1 del genotipo I, lo que constituye la primera evidencia sobre la presencia de este genotipo en la región de Latinoamérica y el Caribe. No obstante, este hallazgo no descarta la posibilidad de una co-circulación de los genotipos I y V.
Palabras clave: dengue 1; Cuba; 1977; filogenia.
ABSTRACT
Introduction: dengue is an acute disease caused by four dengue virus serotypes (DENV-1 to -4) and transmitted to humans by Aedes aegypti mosquitoes. The first laboratory confirmed dengue epidemic in Cuba occurred in 1977, with DENV-1 as the causative agent. It has been stated that the virus spread to several Latin American and Caribbean countries after being introduced in Jamaica in 1977.
Objectives: perform a molecular characterization and determine the genetic relationship of DENV-1 isolated in Cuba in 1997.
Methods: Based on a DENV-1 isolate obtained in Cuba in the year 1997 and preserved at the Strain Bank of the National Arbovirus Reference Laboratory (Pedro Kouri Tropical Medicine Institute), nucleotide amplification and sequencing was performed of the envelope gene using specific primers. Starting from the sequence obtained, a maximum likelihood phylogenetic tree was developed using sequences of DENV-1 strains circulating worldwide.
Results: the DENV-1 isolate corresponding to the Cuban 1977 epidemic was classed as genotype I and genetically related to the reference strain Hawaii 1943. It is noteworthy that the remaining Latin American isolates from that period belong to genotype V, recognized as the American/African genotype.
Conclusions: results show that a DENV-1 strain of genotype I circulated during the Cuban 1977 epidemic. This is the first evidence of the presence of this genotype in the Latin American and Caribbean region. However, this finding does not rule out the possibility of co-circulation of genotypes I and V.
Key words: dengue 1; Cuba; 1977; phylogeny.
INTRODUCCIÓN
El dengue es una enfermedad viral aguda causada por los virus del dengue (DENVs, por sus siglas en inglés), de los que se conocen cuatro serotipos (DENV-1 al 4). Esta enfermedad es transmitida al hombre por el mosquito Aedes aegypti, su principal vector. Actualmente, el dengue y el dengue grave (según la última clasificación clínica de la OMS),1 constituyen la enfermedad viral transmitida por artrópodos de mayor importancia en humanos, ya que anualmente ocurren aproximadamente 390 millones de infecciones en más de 100 países.2
Los DENVs pertenecen al género Flavivirus, familia Flaviviridae y presentan como genoma una cadena de ARN positiva de aproximadamente 11 kb. Dicho ácido nucleico codifica para tres proteínas estructurales: cápside (C), membrana (M) y envoltura (E) y para siete proteínas no estructurales: NS1, NS2A, NS2B, NS3, NS4A, NS4B y NS5.3
Los análisis filogenéticos aplicados a los DENVs se basan fundamentalmente en la secuenciación del gen E. En los últimos años, es cada vez más frecuente la secuenciación del genoma viral completo. No obstante, la secuencia del gen E es aún una herramienta útil para determinar el origen y la dinámica de transmisión viral de los DENVs, ya que la proteína E es muy importante para la respuesta inmune del hospedero,4 y para la determinación de las interacciones evolutivas que se establecen entre el virus y el hospedero.5
En el caso específico de Cuba, la primera gran epidemia de dengue en el siglo XX se notificó en 1977, la cual fue causada por el DENV-1.Se plantea que dicha epidemia comenzó a finales de octubre de ese año en la región oriental del país, extendiéndose posteriormente de forma rápida por todo el país.6 Se estima que se infectó aproximadamente el 50 % de la población, observándose un cuadro benigno de dengue.7
Según la Organización Panamericana de la Salud (OPS), todas las epidemias de dengue ocurridas en el Caribe durante la década del 70 y principio de los años 80, estuvieron relacionadas epidemiológicamente con un brote de DENV-1 que tuvo su origen en Jamaica, 1977.8 Con el fin de estudiar la relación entre cepas de DENV-1 de diferentes orígenes geográficos, Chungue y sus colaboradores en 1995 realizaron un estudio de epidemiología molecular, donde se analizó la secuencia de un fragmento del gen E (180 pb) de 35 aislamientos de DENV-1 de diferentes regiones del mundo, donde se incluye un aislamiento cubano de 1977. El análisis realizado permitió la clasificación del DENV-1 en tres genotipos diferentes (1, 2, 3) con más de un 6 % de divergencia nucleotídica. El aislamiento cubano de 1977 se agrupó dentro del genotipo 2 junto a aislamientos de Senegal, Polinesia Francesa, Nueva Caledonia, Guyana Francesa, Guadalupe, Puerto Rico, Brasil, Perú, Nicaragua y Jamaica. De forma llamativa, dentro de dicho genotipo también se incluía la cepa Hawaii/1943.9
Los genotipos encontrados por Chungue y colaboradores no coinciden con los utilizados en la actualidad. Con el avance de la biología molecularse ha podido analizar la secuencia del gen E completo y las nuevas herramientas bioinformáticas han posibilitado la obtención de árboles filogenéticos de alta resolución que permiten clasificar las cepas de DENV-1 en cinco genotipos, nombrados del I al V.10 Según la nueva clasificación, las cepas aisladas en Latinoamérica desde finales de los años 70 hasta los 90 pertenecen al genotipo V, mientras que la cepa de referencia Hawaii/1943 se agrupa dentro del genotipo I. Asimismo, estudios recientes demuestran que el genotipo V se ha mantenido circulando en Latinoamérica y el Caribe hasta el presente.11
Tomando en consideración que existen pocos estudios con relación a la clasificación del DENV-1 que circuló en Cuba en 1977, en este trabajo se realizó la caracterización molecular a nivel del gen E completo de un aislamiento cubano y se realiza un análisis filogenético, utilizando métodos de máxima verosimilitud para determinar la relación genética del virus aislado en Cuba en ese período.
MÉTODOS
Aislamiento viral
Se utilizó un aislamiento viral correspondiente a la epidemia cubana de 1977, el cual estuvo almacenado a -80 °C por más de 35 años en el Banco de Cepas del Laboratorio Nacional de Referencia de Arbovirus (Instituto de Medicina Tropical “Pedro Kourí”). En el momento del estudio, el aislamiento utilizado contaba con un historial de pases en diferentes líneas celulares: cuatro pases en LLC-MK2 (riñón de mono Rhesus), dos pases en AP-61 (Aedes pseudoscutellaris) y dos pases en C6/36 HT (sublínea de Aedes albopictus de alta temperatura).
Extracción del ARN viral
La extracción del ARN viral se realizó en un extractor automático Qiacube (Qiagen, Alemania). Para ello se utilizaron 140 µL de muestra y los reactivos contenidos en el estuche comercial QI Aamp viral RNA (Qiagen, Alemania), siguiendo las indicaciones del fabricante.
Transcripción reversa-reacción en cadena de la polimerasa (RT-PCR, por sus siglas en inglés)
Para la amplificación del gen E se realizó una RT-PCR en un solo paso, a partir de 10 µL del ARN viral extraído previamente, para ello se utilizó el estuche comercial Transcriptor One-Step RT-PCR Kit (Roche, Alemania), siguiendo las instrucciones del fabricante. Los cebadores empleados en la amplificación fueron: D1F1+ (5’-AGTTGTTAGTCTACGTGGAC-3’) y D1F1- (5’-CCAATGGCYGCTGAYAGTCT-3’) a una concentración final en la mezcla de reacción de 0.5 µM.12
Reacción de secuencia
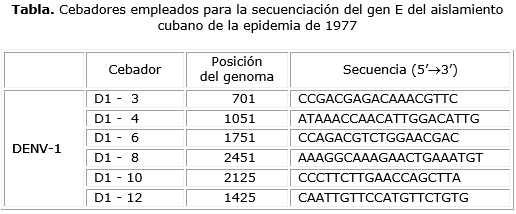
Para determinar la secuencia nucleotídica del gen E se purificó el producto de PCR obtenido, se trabajó con el estuche comercialQiaquick PCR purification (Qiagen, Alemania). Posteriormente, se efectuó la reacción de secuencia con el estuche comercial CEQ DTCS quickstart kit (Beckman Coulter, EE. UU.), siguiendo las instrucciones del fabricante. Los cebadores utilizados en dicha reacción, diseñados en este estudio, se muestran en la tabla 1. Los productos de la reacción de secuencia luego de purificados,se trasfirieron al secuenciador automático de electroforesis capilar CEQ 8800 (Beckman Coulter, EE. UU.).
Procesamiento y análisis de las secuencias
Las secuencias y los cromatogramas correspondientes al aislamiento cubano de 1977se procesaron utilizando el programa Sequencher 4.8 (Gene Codes Corporation, EE. UU.). Luego, la secuencia consenso de dicho aislamiento se comparó con una amplia selección de secuencias del gen E disponibles en la base de datos GenBank. Un total de 83 secuencias se alinearon y editaron con el programa Clustal X13 y BioEdit,14 respectivamente. Posteriormente, se construyó un árbol filogenético utilizando el método de Máxima Verosimilitud con el modelo de sustitución GTR, teniendo en cuenta la distribución gamma y los sitios invariantes (GTR + Γ + I), para lo cual se utilizó el paquete MEGA 5.15 Para evaluar cuan robustos eran los grupos filogenéticos obtenidos, se realizó un análisis de re-muestreo (del inglés, bootstraping), utilizando 1500 réplicas.
RESULTADOS
En este estudio se secuenció un aislamiento de DENV-1 correspondiente a la epidemia cubana de 1977, el cual se agrupó dentro del genotipo I (Fig.). Dicho aislamiento se relacionó genéticamente con la cepa de Referencia Hawaii/1943 y la cepa Mochizuki/1943 (aislada en Japón), con un porcentaje de divergencia nucleotídica de 99,1 % y 97,7 %, respectivamente. Es significativo que el resto de los aislamientos de Latinoamérica de ese mismo período se agrupan dentro del genotipo V, incluido unaislamiento obtenido en Jamaica en el año 1977, reconocido por la OPS como el posible origen de la epidemia de DENV-1 en las Américas. Este genotipo también se nombra Americano/Africano, por incluir cepas de África.10
DISCUSIÓN
El genotipo I de DENV-1, tiene su origen en Asia, específicamente en Japón (cepa Mochizuki/1943), el cual se introdujo en julio de 1943 en Honolulu, Hawaii, por pilotos de aviones comerciales que provenían del sur del Pacífico.16 Desde entonces, dicho genotipo solo ha sido reportado en el sudeste de Asia, China y el este de África.
La relación genética del aislamiento cubano de 1977 con aislamientos del genotipo I, es un resultado totalmente inesperado. Hasta el momento, se pensaba que al igual que otros aislamientos del Caribe, obtenidos entre 1977 y principios de los años 80,8 el aislamiento cubano estaría ubicado en el genotipo V, sobre todo porque no existen estudios previos en la literatura que confirmen la circulación del genotipo I en la región Latinoamericana. No obstante, la relación genética de otro aislamiento cubano de 1977 y la cepa de Referencia Hawaii/1943 ya había sido evidenciada.9 Si bien el estudio de Chungue y colaboradores mostraba un dendograma de baja resolución, ya que los agrupamientos obtenidos se basaban únicamente en un análisis simple de similitud entre las secuencias, dicho dendograma evidenciaba de forma clara la mayor similitud entre el aislamiento cubano de 1977 y la cepa de Referencia Hawaii/1943.
Los resultados del presente estudio, basado en construcciones filogenéticas de alta resolución, con altos valores de soporte estadístico en los nodos principales del árbol, refuerzan la estrecha relación genética existente entre el aislamiento cubano de 1977 y la cepa Hawaii/1943, agrupándose dentro del genotipo I. Estos resultados sugieren que en Cuba, específicamente en 1977, se introdujo un nuevo genotipo de DENV-1 (genotipo I). Sin embargo, este estudio tiene como limitante fundamental, que en el banco de cepas sólo se contaba con un aislamiento de este período epidémico. Por tanto, no podemos aseverar que dicho genotipo era el único agente causal de la gran epidemia de 1977, ya que pudo ocurrir una co-circulación del genotipo I y el genotipo V. Así mismo, en caso de que la co-circulación de ambos genotipos realmente hubiese ocurrido, al no contar con otras cepas del período no es posible determinar el genotipo predominante.
Si bien es llamativa la detección en Cuba en 1977 de un aislamiento similar a la cepa de referencia (circulante en Hawaii en 1943); dicha cepa no fue manipulada durante el estudio y tampoco se utiliza de forma rutinaria en ninguna de las técnicas que se desarrollan en el Laboratorio de Arbovirus, lo que descarta la hipótesis de contaminación de laboratorio. Por otra parte, esta no sería la primera evidencia sobre la circulación de cepas antiguas de dengue en nuestro país.17-19 Rodríguez-Roche y colaboradores demostraron que el agente causal de la epidemia de dengue hemorrágico ocurrida en Cuba en 1981, estaba estrechamente relacionado con la cepa de DENV-2 Nueva Guinea C/1944.20 Estos autores demuestran la ocurrencia de evolución viral a nivel de genoma completo en aislamientos obtenidos durante distintos momentos de la epidemia y descartan la hipótesis de contaminación viral, expuesta previamente por otros autores.21-23 Por su parte, Díaz y colaboradores también detectaron en México, en 1997, la presencia de cepas relacionadas genéticamente con la cepa Nueva Guinea C/1944. Estos investigadores sugieren la posibilidad de un escape de dicha cepa durante experimentos de laboratorio, lo que daría una explicación plausible a la circulación de una cepa similar luego de un período tan prolongado desde su aislamiento en 1944.24 Un hecho similar se evidenció durante la epidemia de influenza ocurrida en Rusia en 1977 causada por una cepa de influenza A H1N1, que había circulado en los años 50.25
Desde el punto de vista epidemiológico, también sería cuestionable que el genotipo I no haya sido detectado en la región Latinoamericana, teniendo en cuenta la probabilidad de dispersión del virus desde Cuba hacia otros países de la región. Una explicación a este fenómeno podría ser que en ese período existía muy poco intercambio entre los países latinoamericanos. Asimismo, un simple error de muestreo también explicaría este resultado, puesto que en esa época no se procesaba un gran volumen de muestras, se contaba con escasos aislamientos y dado el poco desarrollo de la biología molecular, la mayor parte de las cepas caracterizadas se limita a las conservadas por décadas en los bancos de Laboratorios de Referencia. Por otra parte, puede que el genotipo I circulara de forma minoritaria con una baja frecuencia y con restricciones debido a la inmunidad homotípica generada por la exposición previa a cepas del genotipo V. Esta misma explicación fue descrita por otros autores que detectaron a partir de 1979, el genotipo IV en nuestra región, aunque con muy baja representatividad.26,27
Consideramos que a pesar de las limitantes del presente estudio, los resultados muestran hallazgos de interés, reportándose por primera vez la circulación del genotipo I en la región de Latinoamérica y el Caribe. Por más de 30 años de estudio del dengue hemorrágico en Cuba, la secuencias de infección DENV-1/DENV-2 y DENV-1/DENV-3 se han asociado con las formas más graves de la enfermedad. De esta forma, la obtención de la secuencia nucleotídica de un aislamiento cubano de DENV-1, involucrado en una gran epidemia, que sensibilizó a la población cubana exponiéndola a un alto riesgo de dengue hemorrágico, sin dudas permitirá diseñar nuevos estudios de inmunopatogenia viral.
AGRADECIMIENTOS
Los autores dedican este trabajo a nuestro eterno Profesor, el Dr. Pedro Más Lago, quien aislara el virus dengue 1 utilizado en este estudio, durante la epidemia cubana de 1977.
REFERENCIAS BIBLIOGRÁFICAS
7. Cantelar de Francisco N, Fernandez A, Albert Molina L, Perez Balbis E. [Survey of dengue in Cuba. 1978-1979]. Revista Cubana de Medicina Tropical. 1981;33(1):72-8.
8. PAHO. Dengue in the Caribbean, 1977. Proceedings of a Workshop held in Montego Bay, Jamaica, 8-11 May, 1978 PAHO Scientific Publication 375. 1979.
10. Goncalvez AP, Escalante AA, Pujol FH, Ludert JE, Tovar D, Salas RA, et al. Diversity and evolution of the envelope gene of dengue virus type 1. Virology. 2002;303(1):110-9.
11. Rodriguez-Roche R, Villegas E, Cook S, Poh Kim PA, Hinojosa Y, Rosario D, et al. Population structure of the dengue viruses, Aragua, Venezuela, 2006-2007. Insights into dengue evolution under hyperendemic transmission. Infection, genetics and evolution: journal of molecular epidemiology and evolutionary genetics in infectious diseases. 2012;12(2):332-44.
16. Gibbons RV, Streitz M, Babina T, Fried JR. Dengue and US military operations from the Spanish-American War through today. Emerging infectious diseases. 2012;18(4):623-30.
17. Guzman MG, Deubel V, Pelegrino JL, Rosario D, Marrero M, Sariol C, et al. Partial nucleotide and amino acid sequences of the envelope and the envelope/nonstructural protein-1 gene junction of four dengue-2 virus strains isolated during the 1981 Cuban epidemic. The American journal of tropical medicine and hygiene. 1995;52(3):241-6.
18. Alvarez M, Guzman MG, Rosario D, Vazquez S, Pelegrino JL, Sariol CA, et al. [Direct sequencing of an amplified product from a serum sample]. Revista cubana de medicina tropical. 1996;48(1):53-5.
19. Sariol CA, Pelegrino JL, Martinez A, Arteaga E, Kouri G, Guzman MG. Detection and genetic relationship of dengue virus sequences in seventeen-year-old paraffin-embedded samples from Cuba. The American journal of tropical medicine and hygiene. 1999;61(6):994-1000.
27. Villabona-Arenas CJ, Zanotto PM. Worldwide spread of Dengue virus type 1. PloS one. 2013;8(5):e62649.
Recibido: 6 de julio de 2015.
Aprobado: 21 de diciembre de 2015.
Rosmari Rodríguez-Roche. Departamento de Virología, Instituto de Medicina Tropical "Pedro Kourí". La Habana, Cuba. Autopista Novia del Mediodía Km 6 1/2 La Lisa. Po Box 601 Marianao 13.
Correo electrónico: rosmari@ipk.sld.cu