Introducción
Los virus del papiloma humano (VPH) son causantes de la infección de transmisión sexual (ITS) más frecuente en todo el mundo.1 Estudios basados en evidencias epidemiológicas, clínicas y virológicas permiten demostrar que la infección por VPH es la causa del cáncer cervicouterino (CaCU).2
Se describen hasta la fecha aproximadamente 120 genotipos de VPH que, de acuerdo con su potencialidad para inducir carcinogénesis, se clasifican en genotipos de alto (VPH-AR) y de bajo riesgo oncogénico (VPH-BR). El grupo de VPH-AR comprende entre 15 y 19 genotipos que se relacionan, en mayor o menor proporción, con la etiología del CaCU.3 Prácticamente todos los casos de CaCU y sus lesiones precursoras inmediatas surgen de infecciones cervicales persistentes ocasionadas por aproximadamente 15 genotipos de VPH-AR.4 Los más frecuentes son VPH 16, 18, 31, 33, 35, 45, 52 y 58, aunque aproximadamente el 70 % de los casos de CaCU son producidos por los VPH 16 y 18.5
El CaCU y otros cánceres de origen epitelial asociados al VPH representan un importante problema de salud pública en el mundo,5 mientras que en Cuba el CaCU constituye una de las primeras causas de muerte por cáncer en la mujer. Aunque las tasas de mortalidad en Cuba por esta causa están entre las más bajas de América Latina, en los últimos años se evidencia un ligero incremento en este indicador. Tal comportamiento muestra la necesidad de un perfeccionamiento en el sistema de pesquisa, lo cual puede lograrse incluyendo la detección molecular de la infección por VPH.6,7
Los métodos de biología molecular basados en la detección del genoma de ADN del VPH son la principal vía para su diagnóstico.8 La PCR cuantitativa o en tiempo real (PCR-TR) facilita, además de la detección, la cuantificación del ADN viral con ventajas como la rapidez, la amplia variedad de plataformas de amplificación y el mínimo riesgo de contaminación.9 Estos métodos constituyen ensayos eficientes para poder definir programas de pesquisa para la detección precoz del CaCU y para evaluar la eficacia del empleo de las vacunas que se encuentran disponibles en el mercado internacional.10
Aunque en la actualidad la tendencia mundial es a incluir la PCR-TR para la detección del VPH en los programas de pesquisa del CaCU, en Cuba aún no se ha introducido esta prueba. Esto se debe a que los métodos comerciales aprobados internacionalmente son costosos y requieren una infraestructura de laboratorio y personal especializado. De ahí la importancia de contar con estuches comercializados en Cuba, que cumplan con los estándares de calidad, y que permitan garantizar la cobertura del diagnóstico molecular del VPH con una plataforma tecnológica nacional.
El presente trabajo tuvo como objetivo la evaluación de dos sistemas de PCR-TR para la detección del VPH-AR. Ambos estuches son comercializados por el Centro de Inmunoensayo (CIE), en La Habana, Cuba, y permiten la detección y genotipado de los tipos de VPH-AR de mayor relevancia clínica en la etiología del CaCU.
Métodos
Se realizó un estudio observacional con componente analítico, para evaluar los indicadores de desempeño de dos sistemas de PCR-TR comercializados por el CIE.
El estuche SUMASIGNAL VPH 16/18 es una PCR-TR que permite la detección, cuantificación y el genotipado combinado de VPH 16/18 en una sola reacción. Sin embargo, el fabricante no recomienda la cuantificación para el diagnóstico in vitro.
El estuche Human Papillomavirus DNA Diagnostic kit (PCR-fluorescence probing 13+2) (HPV 13 + 2) se basa en una PCR-TR para la detección cualitativa de 15 VPH-AR. En este caso, el sistema realiza la identificación individual del VPH 16 y 18, mientras que los 13 restantes (VPH 31, 33, 35, 39, 45, 51, 52, 53, 56, 58, 59, 66 y 68) se detectan como un grupo, independientemente de cuál sea el genotipo presente en la muestra clínica.
La evaluación de los dos estuches diagnósticos se realizó a partir de un universo de muestras de ADN. Dichas muestras resultaron positivas a diferentes genotipos del VPH, al ser caracterizadas previamente en el Laboratorio de ITS del Departamento de Virología, del Instituto de Medicina Tropical Pedro Kourí (IPK). Estas se recibieron de diferentes hospitales y regiones del país durante el periodo 2016-2019, para el diagnóstico del VPH.
Para la aplicación clínica del ensayo HPV 13 + 2, se estudiaron, además, muestras de cepillado endocervical que se colectaron entre mayo y septiembre de 2020. El universo estuvo constituido por mujeres aparentemente sanas que aceptaron voluntariamente participar en el estudio y que cumplieron los criterios de inclusión siguientes: ser sexualmente activas, no tener antecedentes de enfermedad de cuello uterino y edades entre 20 y 55 años. No se excluyeron mujeres con sintomatología de infecciones ginecológicas en el momento de la toma de muestras.
Todas las muestras cervicouterinas se obtuvieron mediante cepillados, se conservaron a -80 oC en una solución de transporte (Digene Inc., Gaithersburg, MD) y posteriormente se realizó la extracción del ADN y la detección del VPH.
Se estudiaron dos paneles de muestras de ADN que incluyeron, tanto muestras negativas a VPH, como positivas al VPH-AR y VPH-BR. El primer panel estuvo constituido por 150 muestras para la evaluación del estuche SUMASIGNAL HPV 16/18. Este panel fue evaluado en dos etapas, en una primera etapa se utilizaron 80 muestras (Tabla 1A). La segunda etapa se desarrolló con la finalidad de evaluar el proceso de extracción de ácidos nucleicos incluido en el ensayo y para ello se utilizaron 70 muestras de ADN. En la selección de este último grupo de muestras solo se incluyeron muestras positivas al VPH 16 y 18, y un mayor número de negativas con respecto a la primera etapa (Tabla 1B).
El segundo panel estuvo formado por 163 muestras de ADN previamente purificado por el estuche comercial QIAamp® DNA Mini Kit (QIAGEN, Hilden, Alemania) para la evaluación del ensayo HPV 13 + 2 (Tabla 1C).
Tabla 1 Panel de muestras de ADN para la evaluación del estuche: A. SUMASIGNAL HPV 16/18-primera etapa; B. SUMASIGNAL HPV 16/18-segunda etapa; C. HPV 13 + 2
| A) Clasificación de las muestras | Número de muestras | Genotipo de VPH detectado por el ensayo de referencia (número de muestras)* | |
|---|---|---|---|
| Negativas | No detección del VPH | 20 | |
| Positivas | VPH 16 y 18 | 40 |
Solo 16 (31) Solo 18 (3) 18, 11 (1) 18, 31 (1) 18, 33 (1) 18, 11, 58 (1) 18, 58 (1) 18, 31, 58 (1) |
| A otros genotipos diferentes de VPH 16 y 18 | 20 |
31 (4) 33 (3) 35 (2) 43 (1) 45 (1) 51 (1) 56 (1) 58 (1) 59 (1) 61 (1) 66 (1) 70 (1) 72 (1) 82 (1) |
|
| Subtotal de positivas | 60 | - | |
| Total | 80 | - | |
| Negativas | No detección del VPH | 30 | Ninguno |
| Positivas | VPH 16/18 | 40 |
Solo 16 (25) Solo 18 (3) 18, 45 (1) 18, 31, 33 (2) 18, 33, 66 (1) 18, 31, 58 (3) 18, 68 (4) 18, 58, 62 (1) |
| Total | 70 | - | |
| Negativas | No detección del VPH | 94 | |
| Positivas | Solo a VPH 16 | 16 | 16 |
| Solo a VPH 18 | 7 | 18 | |
| VPH 16 más otros genotipos incluidos en el ensayo HPV 13+2 | 13 |
16, 31, 53, 68 (1) 16, 51, 52, 53, 58 (1) 16, 51, 52, 53, 58, 83 (1) 16, 51, 52 (1) 16, 33 (1) 16, 35, 53, 58, 59 (1) 16, 31, 33, 85 (1) 16, 51, 59, 82 (1) 16, 66, 83 (1) 16, 58 (1) 16, 39, 51 (1) 16, 31, 51 (1) 16, 31 (1) |
|
| VPH 18 más otros genotipos incluidos en el ensayo HPV 13+2 | 4 |
6, 18, 33, 59, 61, 72 (1) 6, 18, 51, 58 (1) 18, 35, 70, 71 (1) 18, 33 (1) |
|
| Genotipos del VPH incluidos en el ensayo HPV 13+2, excepto VPH 16 y 18 | 13 |
58, 61 (1) 58, 83 (1) 31, 35 (1) 33, 58 (1) 58, 66 (1) 31, 68 (1) 35, 58 (1) 52, 66, 84 (1) 31,35,52,58,59,61,66,70,71,72,83,84 (1) 35, 51, 58, 59, 61, 66, 84 (1) 33, 51, 52, 58, 61, 66, 70, 83, 84 (1) 6, 52, 66 (1) 53 (1) |
|
| Genotipos de VPH no incluidos en el ensayo HPV 13+2 | 8 |
61, 72, 83 (1) 61 (3) 70 (3) 62, 84, 89 (1) |
|
| Otros agentes de ITS virales diferentes de VPH | 8 |
Virus del herpes simple (2) Citomegalovirus (2) Virus Epstein-Barr (2) |
|
| Subtotal de positivas a VPH | 61 | - | |
| Total | 163 | ||
*Los genotipos detectados se refieren a la prueba diagnóstica de referencia CLART® HPV2 (Genómica, España).
Para la aplicación clínica del estuche HPV 13+2 se emplearon 55 muestras de cepillados cervicovaginales autocolectadas por las mujeres participantes durante el periodo de estudio. Para estas muestras se realizaron todos los procedimientos de transporte, conservación, almacenamiento y extracción de ADN descritos anteriormente para la ejecución del ensayo de evaluación del estuche HPV 13 + 2.
Para la extracción del ADN, se empleó 200 µL de cada muestra y se obtuvo ADN purificado diluido en 100 µL de tampón de elusión, el cual se almacenó a -20º C hasta la realización del ensayo. Este procedimiento se realizó con el estuche comercial QIAamp® DNA Mini Kit (QIAGEN, Hilden, Alemania), según las indicaciones del fabricante.
Ensayos de referencia: Para la evaluación de los indicadores de desempeño de los estuches del CIE se empleó el sistema comercial CLART® HPV2 (Genómica, España), método cualitativo basado en microarreglos de baja densidad. El ensayo detecta simultáneamente 35 genotipos de VPH con mayor importancia clínica: 15 VPH-AR (VPH 6, 11, 40, 42, 43, 44, 54, 61, 62, 71, 72, 81, 83, 84 y 89) y 20 VPH-BR (VPH 16, 18, 26, 31, 33, 35, 39, 45, 51, 52, 53, 56, 58, 59, 66, 68, 70, 73, 82 y 85), según los criterios actualizados de la Agencia Internacional de Investigaciones del Cáncer (IARC).11 Para determinar la validez del estuche SUMASIGNAL VPH 16/18 desde el punto de vista cuantitativo, se utilizó el estuche comercial HPV 16/18 Real-TM Quant (Sacace Biotechnologies, Italia), que es una PCR-TR cuantitativa con genotipado para VPH 16 y 18. Ambos ensayos están certificados por agencias internacionales como la Comunidad Europea, la Food and Drugs Administration Agency (FDA, por sus siglas en inglés) y la Organización Mundial de la Salud (OMS), para su uso en el diagnostico in vitro del VPH.
Estudio de correlación cuantitativa entre el estuche SUMASIGNAL HPV 16/18 y el sistema de referencia: Dado que el sistema SUMASIGNAL HPV 16/18 contiene referencias cuantitativas (desde 4 x 104 hasta 4 x 107 copias/mL), que coinciden con la concentración de los estándares del sistema comercial HPV 16/18 Real-TM Quant de Sacace, se decidió evaluar su desempeño desde el punto de vista cuantitativo. Para ello se analizaron 40 de las muestras positivas a VPH 16 o 18, se graficaron las diferencias entre los logaritmos de las cargas virales obtenidas y se calculó la correlación entre ambos ensayos.
Se estimaron medidas de estadística descriptiva en Microsoft® Excel versión 15.0. Se calcularon los siguientes indicadores de desempeño de los estuches diagnósticos: sensibilidad y especificidad clínica, especificidad analítica o reactividad cruzada con otros genotipos del virus (de bajo o alto riesgo oncogénico) y con otros agentes virales o bacterianos de transmisión sexual, valores predictivos positivo y negativo, concordancia e índice kappa. Para estos análisis se empleó el paquete estadístico Epidat 3.1 y la aplicación LabCalc 1.0.1. Para comparar las cargas virales obtenidas por el método SUMASIGNAL HPV 16/18 con respecto al ensayo de referencia se utilizó el coeficiente de correlación de Pearson.
En todos los casos se obtuvo el consentimiento informado de las mujeres para participar en la investigación. Esta se desarrolló según las normas actualizadas de la Declaración de Helsinki y las Guías Éticas Internacionales para estudios biomédicos en sujetos humanos (CIOMS).12,13
Resultados
Indicadores de desempeño del estuche SUMASIGNAL HPV 16/18 en la primera etapa
Con el estuche SUMASIGNAL HPV 16/18 se obtuvo excelentes indicadores de desempeño (Tabla 2). Los valores de sensibilidad y especificidad clínica fueron satisfactorios (95 % y 98 %, respectivamente). En el cálculo de la sensibilidad clínica solo dos muestras resultaron discordantes y para la especificidad clínica, solamente una.
Tabla 2 Indicadores de la evaluación del desempeño clínico y analítico del estuche SUMASIGNAL HPV 16/18
| Parámetros | SUMASIGNAL HPV 16/18 n = 80 (%) |
|---|---|
| Sensibilidad clínica (IC 95 %) | 95 (89-102) |
| Especificidad clínica (IC 95 %) | 98 (93-102) |
| Especificidad analítica RC (IC 95 %) | 95 (89-102) |
| Valor predictivo positivo (IC 95 %) | 98 (93-102) |
| Valor predictivo negativo (IC 95 %) | 95 (89-102) |
| Concordancia (IC 95 %) | 96 (92-100) |
| Índice kappa (IC 95 %) | 0,93 (0,85-1,01) |
Estudio de correlación cuantitativa entre el estuche SUMASIGNAL HPV 16/18 y el sistema de referencia HPV 16/18 Real-TM Quant de Sacace
Las diferencias entre los logaritmos de las cargas virales obtenidas en ambos ensayos se muestran en la figura 1.
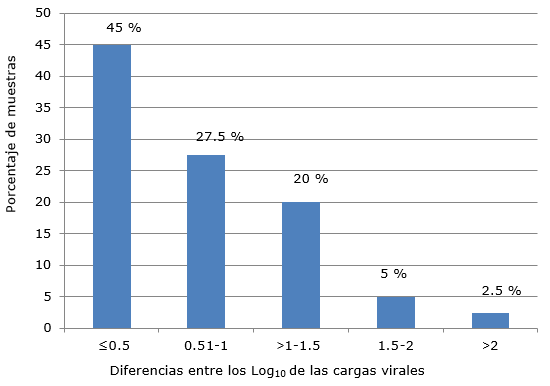
Fig. 1 Comportamiento de las diferencias entre los logaritmos de las cargas virales obtenidas en el ensayo SUMASIGNAL HPV 16/18 y la prueba de referencia HPV 16/18 Real-TM Quant, de Sacace.
Además, se realizó un estudio de correlación entre los logaritmos de las cargas virales obtenidas por ambos métodos mediante el cálculo del coeficiente de correlación de Pearson, para el que se obtuvo un valor de r = +0,688 (Fig. 2).
Indicadores de desempeño del estuche SUMASIGNAL HPV 16/18 en la segunda etapa. Evaluación de dos métodos de extracción de ADN
El estuche SUMASIGNAL HPV 16/18 incluye un paso de extracción de ADN que consiste en la incubación de la muestra clínica durante 10 min a temperatura ambiente en presencia de 5 μL de una solución de lisis. Este paso, recomendado por el CIE, se comparó con el sistema comercial QIAamp® DNA Mini Kit (QIAGEN, Hilden, Alemania) (Tabla 3).
Los valores de especificidad clínica, especificidad analítica y valor predictivo positivo fueron de un 100 %, cuando se emplearon ambos métodos de extracción de ADN. No así la sensibilidad clínica que fue superior cuando se empleó el método de extracción de QIAGEN. Con el uso del método de lisis se obtuvieron dos muestras discordantes y solo una con el sistema comercial. Sin embargo, los valores de sensibilidad clínica fueron muy buenos para ambos protocolos de extracción y no se observaron diferencias significativas (p > 0,05).
Tabla 3 Evaluación del desempeño analítico del estuche SUMASIGNAL 16/18 al comparar dos métodos de extracción de ADN
| Parámetros* | Extracción (%) SUMASIGNAL HPV 16/18 n = 70 | Extracción (%) QIAGEN n = 70 |
|---|---|---|
| Sensibilidad clínica (IC 95 %) | 95 (89-102) | 98 (93-102) |
| Especificidad clínica (IC 95 %) | 100 (100-100) | 100 (100-100) |
| Especificidad analítica RC (IC 95 %) | 100 (100-100) | 100 (100-100) |
| Valor predictivo positivo (IC 95 %) | 100 (100-100) | 100 (100-100) |
| Valor predictivo negativo (IC 95 %) | 94 (85-102) | 97 (91-103) |
| Concordancia (IC 95 %) | 97 (93-101) | 99 (96-101) |
| Índice kappa (IC 95 %) | 0,94 (0,87-1,02) | 0,97 (0,92-1,03) |
*Los indicadores de desempeño se determinaron contra la prueba diagnóstica de referencia CLART® HPV 2 (Genómica, España).
Indicadores de desempeño del estuche Human Papillomavirus DNA Diagnostic kit (PCR-fluorescence probing 13 + 2) (HPV 13+2)
Los indicadores de desempeño analítico del estuche HPV 13 + 2 (Tabla 4) mostraron una sensibilidad y especificidad clínica, y un valor predictivo negativo del 100 %. El valor predictivo positivo y la concordancia con la prueba de referencia fueron excelentes (> 95 %).
Tabla 4 Evaluación del desempeño analítico del estuche HPV 13+2 del CIE
| Parámetros* |
HPV 13+2 N= 163 |
|---|---|
| Sensibilidad clínica (IC 95 %) | 100 (100-100) |
| Especificidad clínica (IC 95 %) | 100 (100-100) |
| Especificidad analítica RC (IC 95 %) | 84 (68-101) |
| Valor predictivo positivo (IC 95 %) | 96 (91-101) |
| Valor predictivo negativo (IC 95 %) | 100 (100-100) |
| Concordancia (IC 95 %) | 99 (97-101) |
| Índice kappa (IC 95 %) | 0,97 (0,93-1,01) |
*Los indicadores de desempeño se determinaron contra la prueba diagnóstica de referencia CLART® HPV 2 (Genómica, España).
La especificidad analítica que incluyó la reactividad cruzada fue el más crítico de los indicadores obtenidos (84 %). Este resultado correspondió a cinco muestras discordantes, que no debieron ser detectadas por el sistema, ya que eran positivas a genotipos de VPH no incluidos en el diseño de la PCR. La reactividad cruzada solo se observó con otros genotipos de VPH-AR, ya que no se obtuvo amplificación para otros virus con genoma de ADN como virus del herpes simple, citomegalovirus o virus Epstein-Barr. Tampoco amplificaron muestras positivas a otros agentes causantes de ITS como Chlamydia trachomatis.
Aplicación a muestras clínicas del estuche Human Papillomavirus DNA Diagnostic kit (PCR-fluorescence probing 13 + 2) (HPV 13 + 2)
Frecuencia de infección por VPH y distribución por grupos etarios
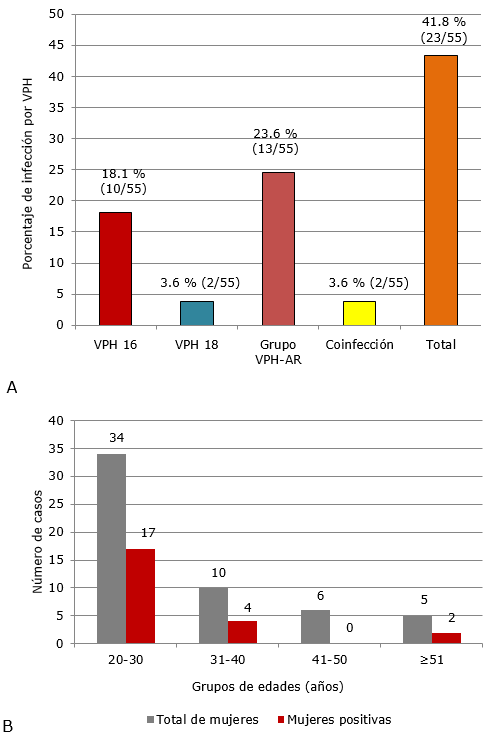
En la figura 3 se muestran los resultados obtenidos cuando se realizó la PCR-TR con el estuche HPV 13 + 2.
Como se observa en la figura 3A, se obtuvo una frecuencia total de infección por VPH del 41,8 %, el genotipo individual más frecuente fue el VPH 16 con 18,1 %, y casi el 24 % de las muestras resultaron positivas a uno o más genotipos del grupo de los 13 VPH-AR. Las coinfecciones se detectaron en un bajo porcentaje de las muestras estudiadas.
En la figura 3B se muestran los resultados del diagnóstico de VPH aplicando el estuche HPV 13 + 2, según los grupos etarios de las mujeres analizadas. Como se evidencia, la mayor parte de las mujeres incluidas en el estudio estaban entre los 20 y 30 años de edad, también fueron las que tuvieron la mayor frecuencia de infección.
Discusión
En la presente investigación se evaluaron dos estuches diagnósticos comercializados por el CIE para la detección in vitro de VPH y su posible aplicación en el programa de pesquisa del CaCU en Cuba.
El estuche SUMASIGNAL HPV 16/18 tuvo excelentes indicadores de desempeño, ya que los valores de sensibilidad y especificidad analítica, los valores predictivos negativos y positivos, y la concordancia fueron superiores al 95 %. Asimismo, el índice de kappa resultó muy bueno, pues tuvo un valor entre 0,81 y 1,00.14
En cuanto a la especificidad analítica o reactividad cruzada, se detectó solo una muestra como falso positivo. Particularmente esta muestra, que era negativa para VPH 16 y 18, era positiva a VPH 45, genotipo que presenta una marcada relación filogenética con VPH 18.15
El sistema de referencia CLART® HPV2 es una prueba de referencia universal para el diagnóstico in vitro de VPH, con excelentes estándares de desempeño. Se validó en numerosos estudios de cohorte, aleatorios y controlados, que incluyeron a cientos de miles de mujeres en todo el mundo, los cuales demostraron su valor clínico. Por lo tanto, las nuevas pruebas de VPH deben mostrar que poseen características clínicas equivalentes (no inferiores) a la prueba de referencia, antes de ser usadas para tamizaje.16
Los resultados del presente estudio coinciden con datos publicados por otros autores que demuestran la utilidad de los sistemas de PCR-TR para el diagnóstico in vitro de VPH y su aplicación para la detección precoz del CaCU.17,18
El estuche SUMASIGNAL HPV 16/18 está diseñado por sus fabricantes como un sistema cualitativo; no obstante, al compararlo contra el sistema comercial HPV 16/18 Real-TM Quant de Sacace, se observó que un gran número de muestras presentaron diferencias mínimas entre los logaritmos de las cargas virales, según ambos métodos.
El coeficiente de Pearson (r = +0,688) fue considerado como bueno, ya que se obtuvo una asociación positiva entre las cargas virales cuantificadas por ambos métodos. Sin embargo, para establecer el valor de la carga viral de VPH 16 y 18 como marcador con significación clínica se necesitaría una correlación casi perfecta, con valores de r entre 0,9 y 1,0. Por tal motivo los resultados sugieren el uso del valor de la carga viral obtenida por el ensayo SUMASIGNAL HPV 16/18, como criterio para seguimiento, pero no como un biomarcador de evolución clínica.19
El estuche SUMASIGNAL HPV 16/18 incluye un paso de extracción de ADN muy rápido y sencillo recomendado por el fabricante. El uso del procedimiento mostró un elevado valor de concordancia con la prueba de referencia diagnóstica e indicadores de desempeño muy buenos. La mayoría de los sistemas de PCR-TR comerciales requieren de laboriosos procesos de purificación de ADN, tal es el caso del estuche comercial HPV Genotypes 14 Real-TM Quant (Sacace Biotechnologies Srl, Italia) que recomienda un protocolo de extracción mediante perlas magnéticas y del sistema Abbott Real Time High Risk HPV (ART; Abbott Molecular, EE. UU.), que incluye un procedimiento de purificación de ADN con precalentamiento y largas incubaciones de las muestras clínicas.20 Los resultados evidencian que el protocolo de extracción incluido en el estuche SUMASIGNAL 16/18 es comparable con un sistema comercial de extracción por columnas, donde se obtiene ADN a partir de muestras clínicas cervicovaginales con elevada calidad y pureza. Este hallazgo permite utilizar un procedimiento de extracción de ADN simple, rápido, que no requiere el empleo de equipamiento adicional, con el consiguiente ahorro de tiempo y recursos.
El estuche SUMASIGNAL 16/18 mostró un buen desempeño para el diagnóstico in vitro de VPH 16 o 18; pero tiene limitaciones para su inclusión como prueba molecular dentro de un programa de pesquisa para la detección precoz del CaCU, pues solo incluye la detección simultánea de dos genotipos de VPH, que, aunque son de gran relevancia en la etiología del CaCU, no son los únicos. Según los criterios actualizados de la IARC, la infección con otros 13 genotipos de VPH-AR (VPH 31, 33, 35, 39, 45, 51, 52, 53, 56, 58, 59, 66 y 68) constituye un factor de riesgo para el desarrollo de lesiones precursoras y CaCU, porque al igual que los VPH 16 y 18, tienen la capacidad para desarrollar persistencia y transformar las células infectadas.11 Por esta razón, la evaluación de un estuche comercializado en Cuba, con capacidad para detectar un número mayor de genotipos ofrece grandes ventajas. El estuche HPV 13 + 2 es una PCR-TR para la detección cualitativa de 15 genotipos de VPH-AR.
Las investigaciones encaminadas a la validación de sistemas de PCR-TR con genotipado amplio enfocan sus estudios de reactividad cruzada en que no existan reacciones cruzadas con VPH-BR,21 un logro también del presente trabajo, pues la identificación equívoca de estos agentes generaría interpretaciones clínicas erróneas que conducirían a protocolos de seguimiento y tratamiento inadecuados.21
El análisis de las muestras discordantes obtenidas en el presente estudio reveló que todos los casos infectados con el genotipo 70 fueron detectadas por el sistema HPV 13 + 2, aun cuando no está incluido entre los 15 genotipos que amplifica el ensayo. De acuerdo con el árbol filogenético de la familia Papillomaviridae,15 el genotipo 70 tiene una elevada homología con otros genotipos que están incluidos en el grupo que detecta la PCR-TR HPV 13 + 2. Tal es el caso de VPH 39, 59 y 68, que se encuentran muy cercanos al VPH 70, en la misma rama del árbol filogenético, pues todos pertenecen a la misma especie a7.15 Esta puede ser la causa de la reactividad cruzada o baja sensibilidad analítica observada, sin embargo este resultado pudiera ser un valor añadido del estuche HPV 13 + 2.
Dicho sistema se escogió para su aplicación a muestras clínicas, lo que permitió evaluar su utilidad para el diagnóstico in vitro en muestras autocolectadas por las mujeres participantes en la investigación. Teniendo en cuenta que las mujeres eran aparentemente sanas, sin antecedentes de enfermedad de cuello uterino, el porcentaje de positividad fue elevado (41,8 %).
Según datos publicados, la prevalencia de infección cervical por VPH-AR en la población abierta oscila desde 1,6 a 25 %, es mayor en las mujeres jóvenes que inician las relaciones sexuales y va disminuyendo con el aumento de la edad.22 Se estima una prevalencia global del 11,7 %, y particularmente en América Latina es del 16,1 %.22 Sin embargo, en estudios previos realizados en mujeres cubanas con citologías negativas, la frecuencia de infección fue superior a lo encontrado en América Latina. Los resultados de la presente investigación coinciden con los hallazgos de un estudio realizado por Soto y otros en 2014 con mujeres de La Habana. Los autores analizaron, mediante PCR-TR, un total de 322 mujeres entre 30 y 59 años, 211 con lesiones cervicales y 111 con citología normal. En las mujeres con citología normal se detectó un 41,6 % de infección, cifra muy superior a lo que se informa para mujeres latinoamericanas.23
En otro trabajo realizado por estos mismos autores en 2016, estudiaron 519 mujeres entre 15 y 59 años, con una citología cervical negativa en los dos años anteriores al estudio. En las mujeres con citología negativa confirmada, la prevalencia fue aún mayor (58,4 %; 147/303). La alta frecuencia de infección por VPH en mujeres de La Habana es un fenómeno que se asoció a predictores de riesgo particulares para la población femenina de la capital.24
Una investigación más reciente demostró que la prevalencia de infección por VPH puede variar, incluso dentro del país. Guilarte y Soto, en el año 2020, investigaron la circulación de VPH en tres provincias de Cuba: La Habana, Villa Clara y Holguín. Analizaron 500 mujeres con antecedentes de citología negativa con edades entre 16 y 67 años. Los autores encontraron diferencias entre provincias para los genotipos circulantes y las variables relacionadas con la infección. No obstante, el genotipo más frecuente fue el VPH 16.25
Al analizar la distribución de los genotipos en la presente investigación, aunque el sistema HPV 13 + 2 no discrimina entre los 13 VPH-AR, sino que los detecta como grupo, se pudo observar un predominio de los virus incluidos en dicho grupo. Aunque el VPH 16 fue el más prevalente, casi el 24 % de los casos estaba infectado con otro VPH-AR. Esto demuestra la importancia del genotipado amplio, pues no solo es fundamental contar con métodos con alta sensibilidad analítica, sino también su genotipificación.
Otro elemento que se evaluó fue la autocolecta de las muestras de cepillado cervicovaginal. El principal indicador de la calidad de este procedimiento fue la amplificación del gen de la beta globina humana, como control interno del proceso de amplificación y como criterio de utilidad de la muestra clínica. En todas se obtuvo amplificación adecuada del control interno, avalando la utilidad de esta práctica, como ya se ha confirmado en estudios previos.26 Esto facilitaría extender el uso del sistema de PCR-TR evaluado hasta la atención primaria de salud y las comunidades rurales en Cuba.
Por todo lo antes expuesto, la presente investigación apoya la posibilidad de contar con dos estuches de producción nacional validados para la detección molecular y el diagnóstico in vitro de VPH-AR, así como la oportunidad de incluir estos ensayos en algoritmos para la detección precoz del CaCU en Cuba. Se confirmaron los estándares altos de calidad de ambas pruebas, lo que permitiría su uso con una cobertura nacional en una plataforma tecnológica disponible para todo el país.