Introducción
Mycoplasma pneumoniae es causa frecuente de infecciones del tracto respiratorio alto y bajo en humanos, especialmente entre niños en edad escolar y adultos jóvenes. Debido a la falta de sensibilidad y al tiempo prolongado necesario para la detección de M. pneumoniae mediante el cultivo bacteriológico, actualmente se utilizan técnicas moleculares para el diagnóstico de estas infecciones.1
Las tetraciclinas, fluoroquinolonas y macrólidos son los antimicrobianos eficaces para su tratamiento, y estos últimos son los más empleados, principalmente en niños debido a los efectos adversos que producen las fluoroquinolonas y tetraciclinas.2 Sin embargo, su uso extensivo condujo a la rápida emergencia a nivel mundial de aislados clínicos de M. pneumoniae resistente a macrólidos.3 El principal mecanismo molecular asociado a esta resistencia es la presencia de mutaciones puntuales dadas por la sustitución nucleotídica en las posiciones 2063/2064 (2058/2059, numeración de Escherichia coli) en la región de la peptidiltransferasa (dominio V) del gen 23S del ARNr.4
El método fenotípico comúnmente utilizado para determinar la susceptibilidad a los macrólidos es la determinación de la concentración inhibitoria mínima (CIM).5 Los métodos moleculares, de amplio uso en la actualidad para la detección de estas mutaciones, se basan fundamentalmente en la secuenciación del dominio V; ambos procedimientos son fiables, pero laboriosos. Además, varios estudios reportan ensayos basados en la técnica de la reacción en cadena de la polimerasa en tiempo real (RT-PCR, por sus siglas en inglés) para la detección de resistencia a macrólidos directamente a partir de muestras clínicas positivas a M. pneumoniae, que han mostrado una excelente correlación con el método de secuenciación.6
En el año 2019 se reporta por primera vez la detección y caracterización de aislados y muestras positivas a M. pneumoniae resistente a macrólidos en Cuba, empleando la técnica de CIM y la secuenciación.7 Sin embargo, la implementación de un algoritmo basado en un método de RT-PCR para determinar la frecuencia de infecciones causadas por M. pneumoniae resistente a macrólidos directamente a partir de muestras clínicas es una tarea de suma relevancia en el contexto epidemiológico actual para el correcto manejo de las infecciones respiratorias en Cuba. Este es un método mucho más rápido que proporciona ventajas sobre los procedimientos de detección de resistencia existentes.
El presente trabajo tiene como objetivo implementar un método de RT-PCR para la detección de mutaciones puntuales en muestras clínicas positivas a M. pneumoniae, diagnosticadas en el Laboratorio Nacional de Referencia de Micoplasmas del Instituto de Medicina Tropical Pedro Kourí (LNR-M/IPK).
Métodos
Cepas de referencias
Se utilizaron cepas de referencia de micoplasmas de origen humano de la Colección Americana de Cultivos Tipo (ATCC, por sus siglas en inglés), pertenecientes a la colección de cepas del LNR-M/IPK: Mycoplasma amphoriforme A39T (ATCC BAA-992), Mycoplasma buccale CH 20247T (ATCC 23636), Mycoplasma faucium DC 333T (ATCC 25293), Mycoplasma fermentans PG 18T (ATCC 19989), Mycoplasma hominis PG 21T (ATCC 23114), Mycoplasma orale Patt (ATCC 15544), M. pneumoniae FH (ATCC 15531) y M6696. Además, se emplearon los aislados autóctonos de M. pneumoniae Mp1 y Mp5. Estas cepas y aislados estaban conservados a -80 °C hasta su utilización.
Muestras clínicas
Se estudiaron 24 muestras clínicas previamente demostradas como positivas a M. pneumoniae en el LNR-M/IPK, que estaban conservadas a -80 °C. Las muestras consistían en exudado faríngeo, esputo, líquido pleural y secreción bronquial, que provenían de pacientes con síntomas respiratorios.
Preparación de los patrones de ADN de Mycoplasma pneumoniae
La cepa referencia de M. pneumoniae FH (ATCC 15531) se cultivó en 50 mL de medio de cultivo SP4 durante 21 días a 37 °C. El cultivo se centrifugó a 15 000 g, durante 30 min a 4 °C y a partir del sedimento celular obtenido se procedió a la extracción de ácido desoxirribonucleico (ADN) mediante el estuche comercial QIAamp DNA Mini Kit (Qiagen, Alemania) siguiendo las instrucciones del fabricante. Se cuantificó la concentración de ADN de los extractos obtenidos mediante espectrofotometría y se prepararon diluciones decimales seriadas desde 105 hasta 10-1 copias de genoma por microlitros (copias/µL) de ADN.
RT-PCR para la detección de las mutaciones A2058G y A2059G en Mycoplasma pneumoniae
Se realizó una RT-PCR para la detección de las mutaciones A2058G y A2059G en el gen que codifica para el ARNr 23S de M. pneumoniae, a partir de muestras clínicas positivas, según la metodología descrita por Kristiansen y otros desarrollada para Mycoplasma genitalium en 2016.8) Se realizaron dos modificaciones a la metodología original. La primera consistió en el uso de los cebadores MH 23S-11: 5’-TAA CTA TAA CGG TCC TAA GG-’3 y MP 23S-22: 5’-ACA CTT AGA TGC TTT CAG CG-’3 descritos por Pereyre y otros en el 2004 para la amplificación de un fragmento de 352 pb del gen 23S del ARNr de M. pneumoniae. La segunda modificación se basó en cambios en el programa de amplificación, específicamente en la cantidad de ciclos empleados, descrito más adelante.9
Se preparó una mezcla de reacción para un volumen final de 25 µL. Por cada reacción de PCR se adicionó 1X de Tampón de PCR (Qiagen, Alemania); 2,5 mM de MgCl2 (Qiagen, Alemania); 200 nM de cada desoxirribonucleótido (Qiagen, Alemania); 500 nM de los cebadores; 200 nM de la sonda correspondiente al genotipo salvaje: Cy5-GGACGG AAAGACCCCGTG AAG CTTT-BHQ2; 100 nM de las sondas MRM-A2063G (FAM-GAC GGG AAG ACC CCG TGA AGC TTT-BHQ1) y MRM-A2064G (FAM-GAC GGA GAG ACC CCG TGA AGC TTT-BHQ1), y 1,5 U de Taq-polimerasa (Qiagen, Alemania).
Como controles positivos se emplearon 5 µL de ADN de la cepa de referencia M6696 y el aislado autóctono Mp5 que contienen las mutaciones A2059G y A2058G, respectivamente, así como la cepa de referencia M. pneumoniae FH y el aislado autóctono Mp1 con genotipo salvaje. Se emplearon también como controles positivos ADN de muestras clínicas positivas a M. pneumoniae genotipificadas previamente que contenían las mutaciones A2058G y A2059G.7 Como control negativo se empleó agua ultrapura.
La amplificación se realizó en un equipo Rotor-Gene Q 5-plex Versión 2.1.0.9 (Qiagen, Alemania), utilizando un programa de un ciclo inicial de 95 °C durante 5 min, 10 ciclos de 95 °C durante 5 s, 60 °C durante 30 s y 72 °C durante 5 s y 40 ciclos de 95 °C durante 1 s, 61 °C durante 30 s y 72 °C durante 5 s.
Para la determinación de la presencia de las mutaciones se utilizó la prueba Scatter en combinación de lectura de los canales de emisión de fluorescencia verde y rojo. Las muestras positivas a M. pneumoniae con alguna de las mutaciones asociadas a la resistencia a macrólidos presentan una mayor fluorescencia en el canal de emisión verde; mientras que las muestras positivas a M. pneumoniae que no poseen dichas mutaciones lo hacen en el canal de emisión rojo.
Evaluación de la sensibilidad, especificidad, repetibilidad y reproducibilidad de la RT-PCR
El límite de detección se determinó como la menor dilución de ADN que mostraba una amplificación en el 95 % de las réplicas. Para ello, se empleó como estándares 5 μL de cada una de las diluciones realizadas a los extractos de ADN cuantificado de la cepa de referencia M. pneumoniae FH (ATCC 15531). La sensibilidad diagnóstica se determinó como la fracción del número total de muestras positivas que son correctamente asignadas con el método de RT-PCR utilizado: verdaderos positivos (VP) / VP + falsos negativos (FN). La especificidad se determinó como la fracción del número total de muestras negativas que son asignadas correctamente con el método de RT-PCR utilizado: verdaderos negativos (VN) / VN + falsos positivos (FP). Para ello se empleó como control positivo 5 μL del extracto de ADN de la cepa tipo de M. pneumoniae (dilución 103), y como muestra 5 μL del resto de las cepas de referencia de las especies de micoplasmas de origen humano. Estos resultados se expresaron en porciento. Se evaluaron como valores de desempeño el cálculo de r2 y la eficiencia del ensayo. Los ensayos se realizaron por triplicado en días alternos, en diferentes laboratorios y con diferentes equipos Rotor-Gene para evaluar la reproducibilidad de los resultados. Se utilizaron tres réplicas por cada dilución de ADN para evaluar la repetibilidad.
Resultados
La RT-PCR mostró un 100 % de especificidad para M. pneumoniae. Se obtuvo una correcta identificación de los genotipos correspondientes a cada control. El genotipo salvaje, identificado por la RT-PCR como “Wild Type” amplificó en el canal de emisión de fluorescencia rojo (Fig., cuadrante superior derecho), y los genotipos mutantes, identificados como “Mutant”, amplificaron en el canal de emisión de fluorescencia verde (Fig., cuadrante superior izquierdo). No se produjo amplificación en las muestras correspondiente a las demás especies de micoplasmas analizadas, al igual que para el control negativo, las que fueron identificadas como “None” y ubicadas en el cuadrante inferior izquierdo (Fig.). La sensibilidad fue de un 92 %, ya que dos de las muestras positivas a M. pneumoniae analizadas por el método resultaron falsas negativas. El límite de detección de la RT-PCR fue 2 copias/µL, lo que se traduce en 10 copias/reacción. Esta es la menor dilución de ADN en la que se alcanzó una amplificación en el 95 % de las réplicas. Este resultado se obtuvo por cada una de las réplicas, y las siguientes dos veces que se repitió el ensayo en diferentes laboratorios y bajo las condiciones de cada uno (Tabla).
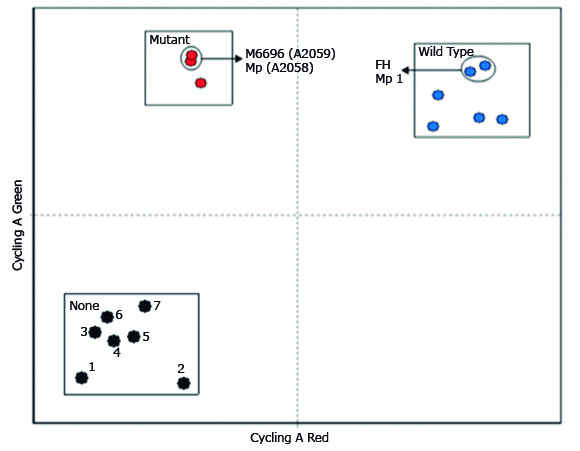
Fig Resultados de la RT-PCR para la detección de resistencia a macrólidos mediante el análisis Scatter en los canales de emisión de fluorescencia rojo (eje horizontal) y verde (eje vertical). 1: Control negativo; 2: M. amphoriforme A39T; 3: M. faucium DC 333T; 4:M. fermentans PG; 5: M. hominis PG 21T; 6: M. orale Patt; 7: M. buccale CH 20247T.
Tabla Resultado de la evaluación por triplicado de los patrones de ADN de M. pneumoniae mediante la RT-PCR implementada
| Ensayo por triplicado | ||||||
|---|---|---|---|---|---|---|
| 106 | 105 | 104 | 103 | 102 | 10 | |
| 1 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 |
| 2 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 |
| 3 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 | 3/3 |
De las 24 muestras clínicas positivas a M. pneumoniae, se logró genotipificar el 91,7 % (22/24) de ellas mediante la RT-PCR para la detección de resistencia a macrólidos. El 77,3 % (17/22) de las muestras se identificó como sensibles a macrólidos (genotipo salvaje), y el 22,7 % (5/22) como resistente a macrólidos (genotipo mutante), al detectarse las mutaciones A2058G/A2059G. Esta técnica no discrimina entre una mutación u otra.
Discusión
Los macrólidos se consideran la primera línea de tratamiento para las infecciones causadas por M. pneumoniae. Sin embargo, los altos niveles de resistencia a estos antimicrobianos desarrollados por este patógeno representan una problemática a nivel mundial. Teniendo en cuenta el lento crecimiento de esta especie, lo que complejiza la determinación de la susceptibilidad antimicrobiana de los aislados clínicos, se ha impulsado el desarrollo y puesta en práctica de métodos moleculares rápidos y certeros para la detección de resistencia a macrólidos directamente a partir de muestras clínicas positivas a M. pneumoniae.
En 2016, Kristians y otros describen un método de RT-PCR basado en el uso de sondas de hidrólisis para la detección de polimorfismos de simple nucleótido en el gen 23S del ARNr de M. genitalium, mediante la detección de las mutaciones A2058G y A2059G asociados con la resistencia a macrólidos a partir de muestras clínicas.8 Teniendo en cuenta la cercanía filogenética que existe entre el gen 23S del ARNr de esta especie y el de M. pneumoniae, con una similitud del 96,7 %,10 en la presente investigación se implementó esta técnica de RT-PCR con algunas modificaciones, para su empleo en la detección de resistencia a macrólidos, a partir de muestras clínicas positivas a M. pneumoniae.
Con la metodología implementada para el estudio de resistencia a macrólidos a partir de muestras positivas a M. pneumoniae se obtuvo una correcta identificación de los genotipos salvajes y mutantes en las cepas de referencia y muestras/aislados autóctonos empleados como controles positivos en el ensayo. Este método de RT-PCR no discrimina entre estas dos mutaciones, sino que las agrupa espacialmente separándolas de las muestras con genotipos salvaje que también se agrupan entre sí.8
En la literatura internacional se describe una variedad de ensayos moleculares basados en la técnica de RT-PCR para la detección, a partir de muestras clínicas, de mutaciones en el gen del ARNr 23S de M. pneumoniae que confieren resistencia a macrólidos. Estos métodos permiten discriminar los genotipos mutantes entre sí y con el genotipo salvaje.
Los resultados de la presente investigación fueron similares a los reportados por Liu y otros en 2014, quienes diseñaron un método de PCR de ciclaje basado en la combinación de una sonda quimérica marcada y una ARNasa H, así como lo informado por Guo y otros en 2019, quienes desarrollan una técnica de PCR alelo específico. Esta última se acopla al análisis de curvas de disociación para detectar las mutaciones asociadas a la resistencia a macrólidos localizadas en la posición A2058G y A2059G del gen 23S del ARNr de M. pneumoniae. Los resultados de ambas PCR aplicadas en aislados clínicos revelan una concordancia del 100 % con los resultados de la secuenciación. El límite de detección que se obtiene en ambos estudios coincide y en ellos se informa una alta especificidad para M. pneumoniae, ya que las PCR resultan negativas para todas las demás especies de micoplasmas analizadas.11,6
Además, el método implementado mostró una mayor sensibilidad y especificidad que las reportadas por Li y otros en 2009, al emplear una PCR múltiple que utiliza la tecnología de sonda de hibridación y la transferencia de energía fluorescente de resonancia (FRET) diseñada para la identificación, directamente en muestras clínicas positivas, de tres mutaciones (A2058G, A2059G y C2617A/G) en la región 23S del ARNr de M. pneumoniae asociadas con la resistencia a macrólidos. En ese estudio la PCR desarrollada resulta positiva para M. genitalium y Mycoplasma pirum, y muestra un límite de detección de 14 copias/reacción.12 Por su parte, Peuchant y otros en 2009, al emplear dos métodos de PCR-FRET para la detección de resistencia a macrólidos en muestras clínicas positivas a M. pneumoniae, demostraron un 100 % de especificidad de estos. Sin embargo, informaron un límite de detección de 20 copias/reacción en ambos casos, aunque los ensayos fueron validados con aislados clínicos caracterizados genéticamente y mostraron una alta concordancia con el método de secuenciación.6
El método de RT-PCR implementado en la presente investigación identifica, directamente a partir de muestras clínicas, los genotipos mutantes A2058G o A2059G y los diferencia de los genotipos salvajes de forma correcta, con una elevada especificidad y sensibilidad, además. Estos resultados permiten recomendar su uso de manera rutinaria en el LNR-M/IPK para el diagnóstico de infecciones causadas por M. pneumoniae resistentes a macrólidos, lo que contribuiría al uso de opciones terapéuticas más eficaces.














