Introducción
La leucemia-linfoma de células T (ATLL) es causada por el virus linfotrópico humano de células T tipo 1 (HTLV-1). Consiste en una neoplasia de células T CD4+ en ganglios linfáticos y sangre periférica, que afecta a varios órganos diana.1,2
El proceso de oncogénesis se inicia en la primera fase de infección viral, lo que genera resultados clínicos diversos y afecta tanto a mujeres como hombres en edad adulta, aunque estos últimos son más susceptibles a este tipo de infección.3,4 La cifra de mortalidad por ATLL es de un 51 % en pacientes con más de 55 años de edad. Este tipo de leucemia es de curso clínico progresivo y además de la manifestación leucémica, se evidencia hepatoesplenomegalia, linfadenopatía sistémica, lesión cutánea, hipercalcemia, entre otros.5)
La ATLL es endémica en América del Sur y Central. Se manifiesta frecuentemente de manera asintomática, lo que sugiere un periodo de latencia bastante extenso. En Latinoamérica el 1 % de la población adulta se consideran portadores asintomáticos; lo que genera un impacto en la salud pública ya que dificulta un diagnóstico oportuno y su pronóstico no es favorable.6,7
El HTLV-1 es un virus ARN monocatenario retrotranscrito perteneciente a la familia Retroviridae conocido como el agente causal de la ATLL. Se caracteriza por presentar diversas proteínas reguladoras virales, entre ellas, basic zipper protein (HBZ). Esta proteína está codificada en la cadena negativa del genoma del virus y es importante en la determinación de la infectividad del virus HTLV-1 y el proceso leucemogénico a través de la regulación del crecimiento y supervivencia de las células tumorales.8)
Para la correcta determinación de casos de ATLL se han evaluado diversos candidatos a marcadores, la mayoría presenta desventajas debido a la falta de preparación de los centros y la falta de acceso de las poblaciones endémicas a estos centros especializados de alto nivel. Entre los marcadores inmunológicos se ha evaluado la supresión en la inmunidad humoral y celular en casos asintomáticos sin resultados concluyentes por lo que es necesario realizar más estudios para comprobar su estabilidad.2,9
Debido al alto impacto que ha generado ATLL en la salud pública, como consecuencia al curso de la enfermedad en pacientes incluso asintomáticos, además de que gran parte de la población afectada se encuentra en zonas vulnerables y de difícil acceso a centros médicos, no es posible el diagnóstico o evaluación adecuada del desarrollo y progresión de esta enfermedad. De de ahí la importancia de desarrollar un algoritmo de diagnóstico preciso para ATLL y diferencial para este tipo de leucemias, que a su vez pueda determinar la progresión y la viabilidad de un tratamiento; necesidades que la proteína HBZ puede cubrir como biomarcador.10
Este trabajo tuvo como objetivo presentar a la proteína HBZ como una alternativa como marcador diagnóstico y de seguimiento al tratamiento de ATLL.
Métodos
Se consultó material académico en diferentes bases de datos científicas como PubMed, Springerlink, Proquest y ScienceDirect. Se consideró en la selección de la información: datos y signos clínicos, efectividad en la identificación y amplificación de la proteína HBZ, reconocimiento de las fases del virus y etapas de la leucemia, epidemiología, mecanismos moleculares en la patogenia del virus. Se acotó que el período de búsqueda no fuese mayor al año 2015 (90 %) y solo el 5 % de los años anteriores se consideró como referente teórico. Los idiomas utilizados fueron inglés y español.
Análisis y síntesis de la información
Leucemia-linfoma de células T en adultos
La ATLL consiste en una expansión clonal neoplásica de las células infectadas con el virus HTLV-1. Es necesario destacar que este virus infecta varias células, sin embargo tiene tropismo para las células CD4. El riesgo de desarrollar ATLL en personas infectadas es cerca del 4 % y generalmente se presenta un retraso desde la infección inicial hasta el período de transformación maligna.4,11,12
A menudo la ATLL genera diversas alteraciones genéticas asociadas con la quimiorresistencia; además el factor edad en los pacientes es de gran importancia clínica debido a que dificulta el tratamiento; por ejemplo, fármacos como vincristina y ASP (asparaginasa) resultan tóxicos en individuos de edad avanzada; que se evidencia en qué la tasa de mortalidad es del 51 % en pacientes con más de 55 años de edad.5,13
Los síntomas de la infección no aparecen en la mayoría de las personas; sin embargo cuando estos se manifiestan el paciente presenta linfadenopatía generalizada, lesiones cutáneas, hepatoesplenomegalia e inmunodeprersión anunciada por infecciones oportunistas establecidas. La oncogénesis se inicia en la primera fase de infección viral y en su segunda fase se mantendrán las propiedades oncogénicas características de las células ya transformadas.3,12,14,15,16
La ATLL cuenta con cuatro subtipos clínicos como: agudo, linfomatoso, crónico y latente (tabla 1).2,3
Tabla 1 Subtipos clínicos de la leucemia-linfoma de células T en el adulto
| Agudo | Fase de peor pronóstico. Se evidencian lesiones cutáneas, síntomas pulmonares con infiltrados y dolor óseo. |
Tiempo de progresión acelerado, en unos cuantos meses será fatal Mediana de supervivencia desde el diagnóstico: 6 meses |
55 |
| Linfomatoso | Se evidencian linfadenopatía masiva y esplenomegalia con ausencia de células tumorales circulantes, además de lesiones cutáneas, hipercalcemia, lesión renal y trastornos neuropsiquiátricos. |
Lento crecimiento. Tiempo de supervivencia: 9- 10 meses. |
20 |
| Crónico | El curso de la enfermedad es más indolente. | Mediana de supervivencia: 2 años. | 20 |
| Latente | A menudo aparecen lesiones cutáneas; sin embargo, estas son más leves. Los síntomas respiratorios y gastrointestinales están ausentes. | Mediana de supervivencia sin tratamiento: 3 años. | 5 |
Epidemiología de la leucemia-linfoma de células T en adultos
En 1980 se describió un virus perteneciente a la familia Retroviridae, identificado como virus linfotrópico T humano que se clasificó en dos tipos: HTLV-1 y HTLV-2.17) Este virus se ha convertido en un problema de salud pública en comunidades nativas de Latinoamérica.18,19) Se calcula que aproximadamente 1,5 billones de personas a nivel mundial están infectadas; el 95 % permanece como portadores asintomáticos y de ellos, el 5 % puede llegar a presentar leucemia asociada a HTLV-1.7,20,21)
Una de las maneras de transmisión del virus son las transfusiones sanguíneas. En Bogotá, Colombia se ha observado un porcentaje de seroprevalencia del 0,3 % de casos en donantes de sangre, a diferencia de los Estados Unidos de América (0,01 %) y Europa (0,001 %) lo que refiere un mayor riesgo de transmisión del virus en la capital de Colombia.7,20
En investigación realizada en el año 2021 por Malpica L y otros del Grupo Latinoamericano de Linfoproliferativos se identificaron pacientes con ATLL en 11 países de América Latina. Gran parte de los casos provenían de Perú (63 %), seguido de Chile (17 %), Argentina (8 %) y Colombia (7 %).18,22
Estudios demuestran que en algunos países latinoamericanos se ha visto el incremento en el número de casos de pacientes con HTLV-1, incluso en provincias donde no se presentaba el virus. Además se ha logrado identificar que una de las vías de propagación viral ha estado directamente relacionada con las transfusiones sanguíneas considerado como uno de los factores más importantes de riesgo en la transmisión de HTLV-1.21,23)
HTLV-1
El virus linfotrópico humano de células T tipo 1 es reconocido como el primer retrovirus oncogénico y como el causante etiológico de la leucemia-linfoma de células T. Además está asociado a otras enfermedades como mielopatía/paraparesia espástica tropical (HAM/TSP), uveítis, dermatitis entre otras.18,24,25,26) El HTLV-1 afecta a millones de personas en todo el mundo, aunque no todas las personas infectadas por el retrovirus desarrollan procesos patológicos, entre el 1 % y 5 % desarrollan ATLL y entre el 3 % y 5 % HAM/TSP; el resto permanece como portadores asintomáticos de por vida.27
Este retrovirus se transmite mediante tres vías, una de ellas es la transmisión a través de transfusiones sanguíneas o por intercambio de agujas. Este tipo de transmisión se ha asociado también a HAM/TSP en aproximadamente el 5 % de personas infectadas. También se puede transmitir mediante contacto sexual y transmisión de madre a hijo por lactancia materna, la cual generalmente desencadena ATLL.28,29
Estructura de HTLV-1
HTLV-1 cuenta con un genoma conformado por diversos genes retrovirales fundamentales como lo son: gag, pro, pol, Env y Px, este último es de gran importancia debido a que allí se encuentran las proteínas accesorias virales TAX y HBZ (fig.).30,31,32)
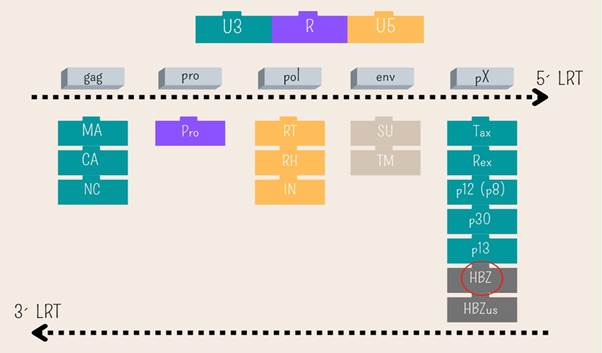
Fig. Representación del genoma de HTLV-1. La repetición terminal larga (LTR) consta la región única de 3 ́(U3), región repetida (R) y región única de 5; además el ARN del HTLV-1 contiene dos sentidos de lectura, en la cadena en sentido positivo, el gen gag codifica proteínas como: matriz (MA), cápsida (CA) y nucleocápsida (NC); Pro codifica la proteasa viral (Pro); Pol codifica la transcriptasa inversa (RT), RNase H (RH) y la integrasa (IN); env codifica la unidad de superficie gp46 (SU) y la unidad transmembrana gp21 (TM) y por último la región Px contiene los genes de diversas proteínas accesorias virales qué son: Tax, Rex, p12, p8, p30, p13 y de la hebra antisentido las proteínas basic zipper protein (HBZ), que se encuentra señalada con un círculo rojo y unspliced (HBZus).
El HTLV-1 cuenta con dos hebras genómicas provisionales con sentido positivo y negativo. La primera codifica gran parte de las proteínas estructurales y la segunda codifica HBZ, específicamente esta es sintetizada a partir del extremo 3´ como el único gen viral expresado en ATLL con implicación importante en la leucemogénesis ya que suprime la proliferación de células ATLL.30,33
Basic zipper protein y leucemogénesis
La HBZ cuenta con un dominio de activación (AD), un dominio central (CD) y un dominio básico de cremallera de leucina (bZIP), importantes para la patogenia de la enfermedad.33,68 Esta proteína es fundamental para preservar la latencia en las células infectadas. Se ha definido su función en la respuesta inmune es mantener la proliferación de células leucémicas; además está presente y constante en todas las células ATLL, o sea, la, HBZ permite la proliferación y patogenia constante del virus.33,34)
Generalmente la proteína HBZ aparece en subtipos clínicos tardíos de la infección como la latente y la crónica. Esto se explica por la capacidad de mantener en el virus no solo su proliferación, sino la infección y leucemogénesis, debido a que su participación en estos procesos reprime otras proteínas del virus capaces de generar una alta respuesta inmune como la molécula Tax frente a linfocitos T citotóxicos.35,36
El desarrollo de cánceres implica una tasa elevada de proliferación celular, es decir, el aumento del número de células como resultado del crecimiento y la multiplicación celular descontrolada. La HBZ, a través de acciones pleiotrópicas consecuencia de la interacción con otras biomoléculas, como junD, bTERT, E2F1, Wnt5a, entre otras; logra la proliferación celular. Por ello se considera un aspecto importante en la leucemogénesis ya que mejora la proliferación celular de células T.35
En cuanto a las propiedades oncogénicas de la HBZ se encuentra: la supresión de la transcripción viral de otras proteínas inmunogénicas, la promoción de la proliferación de células T y la supresión de la apoptosis celular, lo que favorece la leucemogénesis.37 La linfoproliferación celular se logra a través de la modulación de señales celulares que están implicadas en procesos inflamatorios como INF-y, TGF-b Foxp3 y Wnt5a.37,38)
Diferentes estudios exponen que pacientes que no están expuestos al ARNm de HBZ, ya sea por la deleción del segmento que codifica la proteína o por silenciamiento del gen, muestran menores índices de proliferación de células ATLL.39,40 A través de la HBZ el virus también se une a otras moléculas de señalización celular tales como SMAD 2,3, NFAT, FoxO3a y otras. Estas interacciones permiten la promoción del linfoma e incluso impedir la apoptosis celular.38) Además le permite al virus modificar a conveniencia el funcionamiento de NF-kB, mecanismo importante en la supresión de la apoptosis y la proliferación celular.35,40,41)
Se identificó un socio en la vinculación para la proteína de unión al elemento de respuesta cAMP-2 (CREB-2). Esta relación se conoció mediante el cribado de levadura bihíbrida (Y2H), técnica molecular que se usa para descubrir la interacción entre proteína y proteína. La relación de HBZ y CREB es fundamental en la oncogénesis de la leucemia ya que la unión de ambas moléculas hace que Tax (otra proteína del virus relacionada con la patogenia) reduzca su expresión y como consecuencia los linfocitos T citotóxicos no encuentran su diana principal, lo que provoca que el virus genere resistencia y evasión de la respuesta inmune del huésped.37,38,40
Localización citoplasmática y nuclear
Se han realizado diversos procesos de localización subcelular de esta proteína mediante metodologías como microscopía confocal e inmunofluorescencia, en los que se identificó la localización de HBZ tanto en el citoplasma como en el núcleo de la célula huésped, lo que se correlaciona con la patogenia y la presencia de neoplasia.42
La expresión y localización de la HBZ depende de la progresión hacia diferentes enfermedades asociadas con HTLV-1. Así, en los casos de pacientes portadores asintomáticos o HAM/ TSP, la proteína HBZ permanecerá en el citoplasma celular. No obstante, en el caso de ATLL en la infección primaria en los linfocitos T se ha establecido su presencia también en el citoplasma pero su progresión neoplásica producida en la ATLL crónica o aguda está relacionada con una translocación progresiva de HBZ desde el citoplasma hacia el núcleo. Es decir, la localización de HBZ es un factor importante para determinar los cambios leucemogénicos; dicha translocación del citoplasma al núcleo podría estar influenciada de factores y ciertos mecanismos de retención a nivel del citoplasma de los linfocitos T.42,43
HBZ: caracterización molecular
La caracterización molecular del genoma viral permite identificar los resultados clínicos asociados al HTLV-1. Un elemento fundamental para la persistencia del retrovirus es la proteína HBZ, proteína reguladora codificada por el dominio bzip de HTLV-1.
Las variaciones genómicas involucradas en la patogénesis pueden presentarse en genes como GBP 2 y CD40LG, los cuales son marcadores de la infección del virus en ATLL.38
Después de un análisis de 568 secuencias de la región HBZ se estableció que la mutación R119Q es determinante para la actividad del virus y la diferenciación clínica. Diversos estudios soportan que la aparición de esta mutación se presenta con más frecuencia en pacientes asintomáticos.35,38,44,45)
HBZ y Tax diferenciación
Para comprender el desarrollo de la transformación oncogénica en un individuo con HTLV-1 es necesario reconocer la importancia de Tax y HBZ.
La Tax es una proteína reguladora codificada por la región pX del genoma del virus. La expresión de esta proteína es una proliferación celular persistente caracterizada por una expansión anormal de las células infectadas, que genera daño de ADN características del ATLL.8,44,45)
La Tax contribuye al mejoramiento de la maduración de células dendríticas ejerciendo cambios en su función, lo que genera una presentación constante de antígenos a las células T. Además es un activador de factores de transcripción, entre los cuales se encuentra: NF-kB y AP-1 implicados en el proceso de transactivación de IL-8.5 Las células T son cruciales en el mantenimiento de la homeostasis del sistema inmunológico, puesto que actúan suprimiendo la proliferación y la producción de citocinas por las células T patógenas.17
La Tax induce la producción de las citocinas proinflamatorias que favorecen significativamente el desarrollo de inmunidad citotóxica, además de la producción de inmunoglobulinas. La Tax induce la producción de CXCL10, CCL1, CCL3 y CCL4, las dos últimas quimiocinas atraen a las células dendríticas hacia los ganglios linfáticos.5
Por su parte, la HBZ induce la producción de CCL21 y CCR4 en las células infectadas con el virus y apoya la proliferación y migración de células T infectadas; cabe destacar que la expresión de HBZ se ha correlacionado con la carga proviral HTLV-1 resaltando como un factor fundamental para preservar la latencia en las células infectadas.33,35) La HBZ interactúa con CREB, suprimiendo la transactivación mediada por la proteína Tax, además inhibe la vía canónica NF-kB.5
Es indispensable diferenciar la función de HBZ y de Tax dentro del proceso de transformación celular y mantenimiento del fenotipo leucémico. Las propiedades inmunogénicas de Tax conducen a una eliminación pronta de las células que expresan esta proteína; por su parte, la HBZ se expresa en todos los pacientes con ATLL.46
Si bien tanto Tax como HBZ mantienen la persistencia viral e influyen en la patogénesis; la HBZ induce la replicación viral y suprime la respuesta inmunológica y, aunque el proceso oncogénico se atribuye a Tax por ser el encargado de secuestrar los mecanismos básicos de control de homeostasis celular, solo el 40 % del total de pacientes con ATLL expresan esta proteína, a diferencia de la HBZ que es expresada en la totalidad de los pacientes con ATLL. Ello correlaciona directamente con que esta proteína se encuentra implicada en las primeras fases del proceso oncogénico del ATLL y por otro lado se ve implicada en el mantenimiento del estado leucémico.32,43)
HBZ como marcador diagnóstico del ATLL
La proteína HBZ es estable y puede ser identificada desde los inicios de la enfermedad, en fases agudas y crónicas; para lo cual se usan diferentes pruebas como por ejemplo qRT- PCR, Southern o Western blot, ELISA, ISH (tabla 2).
Tabla 2 Pruebas para la determinación de la proteína HBZ
| qRT-PCR.8,47,48 |
Extracción ADNc procedente del ARNm del HBZ. Se cuenta con un |
|
Carga de ARNm de HBZ directamente proporcional a la carga proviral en pacientes ATLL |
| Hibridación in situ (ISH).51,52 |
Secuencia para codificar HBZ en el virus. ( Marcaje con sonda |
Secciones de tumores en parafina | Visualización de la proteína en microscopio de fluorescencia o de estándar de campo brillante si la reacción es enzimática |
| Ensayo de inmunoabsorción ligado a enzimas (ELISA).30,52,53,54 |
ELISA en marcaje IgG. |
Sangre periférica. Plasma Nódulos linfáticos |
Calcular la absorbancia mediante Densidad óptica (OD) |
|
|
Las enzimas de restricción de Pst1 y EcoR1 digieren el ADN del virus en los respectivos sitios de clivaje. Se realiza electroforesis |
Sangre periférica Aspirados de médula ósea Tumores Líquidos bronquiales |
Los productos son separados por tamaño en geles de agarosa al 0,7 %. Se lee el peso molecular en membranas de nylon. Es detectable según la expresión de la región pX del virus |
Estudios asociados
Algunos estudios evidencian la utilidad de la proteína HBZ como marcador diagnóstico para ATLL (tabla 3).
Tabla 3 Utilidad diagnóstica de la HBZ
| Baratella M y otros43 (2017) | Estudio de PBMC en pacientes con ATLL mediante inmunofluorescencia y microscopía confocal. | Entre el 80 y 83 % de las células fueron positivas para HBZ. Además se localizó como estructuras moteadas en el núcleo. |
| Saito M y otros57 (2009) | Cuantificación de niveles de ARNm de HBZ en células PBMC de 10 pacientes con ATLL y PA. | Mayor nivel ARNm ATLL/ PA ( |
| Tarokhian H y otros40) (2018) | Se utilizó ADN y ARNm de PBMC de 18 pacientes con ATLL, 10 OP y 18 PA con el fin de evaluar la expresión génica por medio de qRT-PCR y ensayo TaqMan. |
Expresión de HBZ mayor en ATLL (101,76 ± 61,3) que en PA (0,12 ± 0,05) y OE (0,01 ± 0,1) ( HBZ se expresa en casi todas las células malignas del paciente con ATLL y su expresión se correlaciona positivamente con CPV (R = 0,50; |
| Takatori M y otros51 2021 | 62 biopsias ATLL, 57 no ATLL y 41 PA de cinco hospitales de un área endémica de HTLV-1. Nuevo algoritmo diagnóstico técnica |
Diagnóstico 94 % (112/119) Distinguió ATLL de casos no ATLL. Sensibilidad y especificidad 100 %. |
CPV: Carga proviral; PBMC: Células mononucleares de sangre periférica; PA: portadores asintomáticos; OE: Otras enfermedades; Carga de ARNm de HTLV-1 HBZ = valor de HBZ/valor de HPRT(hipoxantina ribosil transferasa).
Los estudios citados muestran el gran uso del ARNm HBZ y su correlación con la carga proviral lo que lo involucra una vez más la oncogénesis y que debe tomarse en consideración como aspecto fundamental en el diagnóstico.
En los niveles de ARNm de HBZ de ATLL en comparación con otras enfermedades como la mielopatía/paraparesia espástica tropical (HAM/TSP) se observa un mayor nivel lo que permite diferenciar de una forma específica los casos de ATLL de los no ATLL.
HBZ posible evaluador del tratamiento
Encontrar valores altos de carga proviral a partir de PCR es equivalente a la expresión del ARNm de HBZ y viceversa. Se ha demostrado que pacientes que muestran recuperación al ATLL tienen una reducción notable en la expresión de HBZ. Además, esta molécula se ha propuesto por varios autores como diana terapéutica y debe ser considerada como parte de la prevención y evitar la progresión a una fase crónica.58,59,60,61
En la actualidad aún no existe un tratamiento curativo para el ATLL, pues aunque los subtipos indolentes son de mejor pronóstico, los subtipos agresivos aún son objeto de estudio para diversos tratamientos. (40,61,62 Varios estudios han logrado determinar que la expresión de ARNm de HBZ en relación con la carga proviral (PVL) de HTLV-1 son una propuesta tentadora a la hora de realizar el seguimiento al tratamiento aplicado en un paciente con ATLL.37,63)
Akbarin MM y otros37) realizaron un estudio qué incluyó pacientes diagnosticados con ATLL (12 masculinos,13 femeninos y 18 entre hombres y mujeres asintomáticos con HTLV-1) y determinaron que una PVL alta se puede correlacionar directamente con un tiempo de supervivencia más reducido en los pacientes con ATLL, lo que permite a su vez controlar y predecir los resultados de la infección por el virus en el que generalmente el tratamiento de primera línea consiste en una quimioterapia combinada.37,38)
En otros estudios luego de una cuantificación de los niveles de expresión de ARNm de HBZ en pacientes con ATLL, la expresión de ARNm de HBZ fue bastante alta; en pacientes postratamiento con INF-α se evidenció una disminución en el nivel de expresión del ARNm de HBZ, la que estuvo relacionada con la mejoría; no así en el caso de los pacientes sin mejoría. Ello sugiere la utilidad de la HBZ como biomarcador de respuesta al tratamiento.8,57,64
La ATLL tiene un mal pronóstico ya que su reacción a la quimioterapia no es favorable; además el tratamiento de primera línea genera efectos secundarios qué afectan significativamente la calidad de vida del paciente.
Se ha estudiado si la hipericina (HY), un tipo de fotosensibilizante en la terapia fotodinámica (PDT) que por sus excelentes propiedades y actividades antitumorales actúa destruyendo las células malignas con efectos secundarios leves, tiene alguna influencia sobre HTLV-1.
Xu L y otros estudiaron la expresión del ARNm de HBZ después del tratamiento con HY mediante Western y RT-PCR semicuantitativa, y encontraron que la HY suprimió la expresión del HBZ de dosis dependiente (p < 0,01; en comparación con el grupo de control).65
Cabe resaltar que hay propuestas y serias consideraciones de inmunizar a partir de HBZ como se menciona por Zhang LL y otros, en su estudio que un péptido de HBZ puede ser usado como vacuna al inducir inmunidad a través de linfocitos T citotóxicos; incluso es posible obtener linfocitos T citotóxicos sensibilizados por la vacuna recombinante (rVV-HBZ).5,63,66 También se ha demostrado que entre otras moléculas candidatas para el desarrollo de una vacuna a HTLV-1, HBZ es la que cuenta con mayor antigenicidad en la identificación por parte del MHC clase II a partir de la identificación del epítope específico de HBZ.67,68
El HTLV-1 se ha convertido en un problema de salud pública en Latinoamérica; en que la demora en la detección genera incrementos significativos de los índices de mortalidad. El estado leucémico de la infección genera un índice de supervivencia escaso pues los tratamientos no generan la respuesta esperada y generalmente este se detecta en personas de edad avanzada en los que los tratamientos no son viables debido al factor edad.
La HBZ, a diferencia de otras moléculas de interés en una infección con HTLV-1 y específicamente en un estado leucemogenico, siempre se encuentra expresado en el paciente independientemente del estadio de la enfermedad; lo cual indica que está directamente relacionada con la oncogénesis; aparte de de reflejar la capacidad del virus de seguir replicando su material genético. Además, la HBZ es capaz de evitar las respuestas de células T citotóxicas; por otro lado, la ubicación de esta molécula permite identificar el tipo de manifestación generada por el retrovirus y el nivel de ARNm es mayor en ATLL que en otras manifestaciones clínicas.
A partir de la presente revisión, es posible plantear que la HBZ es una molécula estable y presente a lo largo del proceso infeccioso del virus, con la posibilidad de convertirse en un biomarcador diagnóstico y de seguimiento del tratamiento de pacientes infectados con HTLV-1 y con progresión leucémica o ATLL; que es tan versátil que, incluso se ha pensado que pueda ser usada en vacunas.














