Introducción
En el mundo en el 2020 se diagnosticaron 2 206 771 (11,4 %) y en Latinoamérica y el Caribe 97 601 (4,8 %) de nuevos casos de cáncer pulmonar (CP), con 1 796 144 y 86 627 pacientes fallecidos, respectivamente.1
El CP es la primera causa mundial de muerte por cáncer y la más evitable, ya que la mayoría de los casos de este cáncer son secundarios al tabaquismo.2,3 El cáncer de tráquea, bronquios y pulmón produjo 5626 muertes en 2019 en Cuba, con una tasa de 50,1 x 100 000 habitantes.4
El CP se divide en dos grupos: células pequeñas (SCLC, más agresivo, 15 % de casos y mediana de supervivencia < 2 años) y de células no pequeñas (NSCLC, aprox. 85 %).2,5 Los principales subtipos de NSCLC son adenocarcinoma (40-50 % de todos los CP), carcinoma de células escamosas (SQCC) y carcinoma de células grandes (LCLC).6 La clasificación de OMS de 2015 incorpora nuevos resultados.7
La resección quirúrgica en estadios iniciales del CP tiene pronóstico favorable con supervivencia a los 5 años de 70 %, pero, lamentablemente, la mayoría de los pacientes se diagnostican en estadios avanzados.8
Dentro de los factores de riesgo de CP se encuentran factores ambientales relacionados con estilos de vida y factores genéticos vinculados a oncogenes y genes supresores tumorales.9 Los factores de riesgo ambientales son contaminantes como arsénico, cromo, asbesto y humo de diésel.10,11,12,13 El tabaquismo es responsable de más del 80 % de los casos.7,14,15 La enfermedad pulmonar obstructiva crónica y la fibrosis pulmonar idiopática confieren también un alto riesgo.16,17)
Además, se han asociado factores metabólicos con el CP. Carreras-Torres y otros18 encontraron un papel causal de la insulina en ayunas y el colesterol de lipoproteína de baja densidad en la etiología del CP, así como con el índice de masa corporal en SQCC y SCLC. Otros autores han encontrado alteraciones del perfil lipídico asociado a estrés oxidativo.19
La comprensión de los factores genéticos que participan en el CP es básico para aclarar la etiología de la enfermedad y para el desarrollo de tratamientos específicos en grupos de pacientes.7,20 Se sugiere que alrededor del 20 % del CP es heredable, lo que confirma la asociación entre genética y riesgo de cáncer; la susceptibilidad genética es mayor en el adenocarcinoma.21
En el cáncer intervienen dos tipos de genes: oncogenes y genes supresores tumorales.22,23 Los oncogenes son versiones mutadas de genes involucrados en la regulación del ciclo celular. 24 Los proto-oncogenes se descubrieron en virus tumorales que derivaban de genes de las células hospederas animales, los cuales codificaban proteínas reguladoras del crecimiento celular.
Los genes supresores tumorales codifican proteínas que normalmente restringen la división celular. El crecimiento celular incontrolado por defectos de los genes supresores tumorales, a diferencia de los oncogenes, es genéticamente recesivo, pues se requieren que ambos genes de un par de cromosomas sean defectuosos. Esto se debe a que la función de estos genes es evitar la división celular y si una copia de un gen es normal se produce una proteína normal y una normal inhibición de la división.24
Las mutaciones germinales raras de genes como TP53, RB1 y EGFR confieren predisposición hereditaria al CP, aunque lo más frecuente es el efecto de los factores ambientales sobre determinados genes para desarrollar un fenotipo canceroso, que transcurre en múltiples etapas, con alteraciones genéticas y epigenéticas en genes susceptibles.20,25
El descubrimiento de genes involucrados en el CP tiene importancia práctica para la identificación de biomarcadores tumorales para el diagnóstico precoz más preciso del cáncer, lo que permite una mejor opción terapéutica y una mejor evaluación pronóstica de los pacientes.26 Con base en estos conocimientos se han diseñado tratamientos novedosos, como el empleo de anticuerpos monoclonales y la edición de genes. La desventaja principal de estos tratamientos es su costo y que requieren tecnología avanzada.
Métodos
Se revisaron preferentemente los artículos originales, las revisiones bibliográficas, las revisiones sistemáticas y los metaanálisis de los últimos 5 años. El periodo de búsqueda de información comprendió desde abril a julio del 2020. La autora principal elaboró un borrador inicial con un guion que envió sucesivamente a los otros autores, quienes le agregaron cambios y referencias, hasta que se concluyó el informe final enviado a la revista.
En US National Library Medicine National Institutes of Health con el descriptor “lung cáncer” se encontraron 279 620 trabajos a texto completo.
En Annual Reviews con “oncogenes lung cancer” se localizaron 452 artículos a texto completo sin límite de tiempo.
En SciELO se encontraron 591 artículos a texto completo al realizar una búsqueda con el descriptor “cáncer del pulmón”.
Análisis e integración de la información
ADN
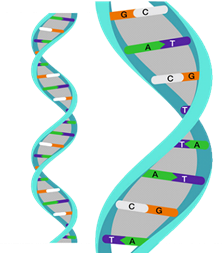
La molécula de ácido desoxirribonucleico (ADN) es un polímero de doble cadena de nucleótidos que se asemeja a una doble escalera de caracol (Fig. 1). Los travesaños de la escalera están compuestos por unidades de desoxirribosa unidas por fosfatos.27 Los peldaños de la escalera están formados por los puentes de hidrógeno entre bases nitrogenadas complementarias.
Así como el código binario es para el lenguaje de la computadora donde cada instrucción se escribe como una serie de 0 y 1, es la secuencia de las cuatro bases nitrogenadas (adenina [A], guanina [G], citosina [C] y timina [T]) lo que explica las direcciones para hacer cada proteína.28
La A siempre se aparea con T mediante dos puentes de hidrógeno y la G con la C por tres puentes de hidrógeno. Por tanto, solo se necesita determinar la secuencia de bases en una cadena para conocer la secuencia de bases de su cadena complementaria.
Cromosomas
Si se estira el ADN nuclear (nDNA) mide más de 6 pies de largo con un código de 6 billones de letras. Por tanto, el ADN se empaqueta en el núcleo como cromosomas. El tamaño de los cromosomas humanos oscila entre 50 000 000 y 300 000 000 de pares de bases y puede contener de cientos a miles de genes.
Por ejemplo, el cromosoma uno el más grande, contiene 2100 genes codificantes de proteínas, mientras el cromosoma y contiene el menor número con 60 genes.27 Dentro del núcleo, excepto los espermatozoides y óvulos, existen 46 cromosomas como 23 pares, 22 pares autosómicos y un par sexual.
Genes
Los genes son sectores de ADN que determinan la secuencia de cada proteína de un organismo.27 Un gen codifica una o cientos de proteínas diferentes, como promedio tres proteínas. Se estima que los seres humanos sintetizan de 250 000 a un millón de proteínas diferentes.
El tamaño de un gen depende del número de pares de bases que contiene, desde unos cientos hasta más de dos millones.
La secuencia de ADN en el gen se divide en dos regiones: exones e intrones. Durante la transcripción, el ARN mensajero se copia a partir del gen y en los ribosomas se traduce a proteína.27 Los exones son regiones codificantes de gen que determinan los aminoácidos que componen una proteína; los intrones realizan funciones reguladoras y se eliminan.
Muchos genes se denominan con un símbolo por las proteínas que codifican.27 El Human Genome Organization (HUGO) Gene Nomenclature Committee establece un nombre y un símbolo oficial para cada gen, pero los genes reciben múltiples nombres en la literatura.
Genotipo y fenotipo
El fenotipo es un rasgo visible o característica debida a la expresión genética, como el color de los ojos o la tasa del metabolismo del citocromo p450.
Los seres humanos son diploides, es decir, tienen dos versiones de cada gen, heredados de cada progenitor. El proceso de meiosis determina que un gen de los dos sea heredado por los descendientes, lo que es la base de la ley de segregación mendeliana.27
Cuando se produce la fecundación, el huevo fertilizado contiene el genoma de los descendientes, el cual pasa a las futuras células en cada división celular. El genotipo se refiere a la combinación específica de dos alelos en la herencia individual.
Los alelos son variantes del mismo gen con pequeñas diferencias en su secuencia de bases, lo que puede variar la proteína codificada.27 Algunos genes tienen más probabilidad de presentar variación y el término polimorfismo (múltiples formas) se emplea generalmente para genes con múltiples alelos. Un polimorfismo genético es una variación en una secuencia de ADN presente en más del 1 % de una población.
La mayoría de los genes en los cromosomas autosómicos tienen expresión bialélica, lo que significa que ambas copias expresan la proteína codificada.
Genes en el cáncer pulmonar
El cáncer se debe una proliferación incontrolada de células debida a un desbalance entre la multiplicación celular y la muerte celular programada.29
El estudio de los genes implicados en el CP se dificulta por la gran heterogeneidad genética de estos tumores30,31,32 y por el gran tiempo transcurrido entre la aparición de los primeros cambios histológicos y las manifestaciones clínicas.
La heterogeneidad interpaciente del CP se relaciona con variaciones genéticas y fenotípicas en personas con el mismo tipo de cáncer, lo que podría explicar la diferente respuesta al tratamiento.33 La heterogeneidad intratumoral se refiere a diversidades subclonales de células tumorales en un tumor específico, mientras la intertumoral es la diversidad entre el tumor primario y las metástasis.
La heterogeneidad, uno de los principales desafíos del CP por sus implicaciones terapéuticas, se atribuye a mecanismos no genéticos, genéticos y epigenéticos.33,34 Los tumores presentan aberraciones cromosómicas como aneuploidia, ganancia y pérdida de regiones cromosómicas, reordenamientos genéticos, ganancia en número de copias y amplificaciones.33 Un ejemplo, el SCLC con frecuencia presenta una deleción 3p2.35
La inestabilidad genómica, uno de los sellos distintivos del cáncer, produce aberraciones genéticas que van desde mutaciones simples o de pocos nucleótidos, a cambios en partes del cromosoma. La instabilidad cromosómica es una inestabilidad genómica asociada a variaciones numéricas o estructurales de los cromosomas, como pérdida o ganancia de fragmentos de cromosomas, translocaciones, deleciones y amplificaciones.33) Su importancia clínica en el CP se debe a su peor pronóstico, independiente de factores de riesgo como estadio tumoral, edad y sexo. La heterogeneidad genética contribuye a la resistencia a los medicamentos en pacientes con CP.36
En NSCLC los genes con más frecuencia mutados con potencial función de genes diana son los siguientes: EGFR, FGFR1, KRAS, PIK3CA, ERBB2 (HER2), BRAF, ALK, ROS1, MAP2K1/MEK1, RET, NRAS y AKT1.7,37
Un estudio confirmó en adenocarcinomas una alta tasa de mutaciones de TP53 (50 %), KRAS (27 %), EGFR (17 %), STK11 (15 %), KEAP1 (12 %), NF1 (11 %), BRAF (8 %), SMAD (4 %).7 Otros genes frecuentemente mutados son U2AF1, RBM10 y ARID1A. Por otro lado, alteraciones frecuentes en el número de copias se observan en: ganancia de TERT, MYC, MCL1, EGFR, ERBB2, NKX2-1; la pérdida de TP53 y CDKN2A.7 Las mutaciones de TP53 y U2AF1 tienen pronóstico negativo y se asocian a menor supervivencia.
Según datos de Clinical Lung Cancer Genome Project (CLCGP) y Network Genomic Medicine (NGM), los genes más frecuentemente mutados en CP son TP53, KRAS, STK11, EGFR, KEAP1 y NFE2L2 (4,5 %).38
Las mutaciones de EGFR, KRAS y ALK también juegan un papel en la reprogramación metabólica de las células cancerosas pulmonares para apoyar la alta tasa proliferativa y las grandes demandas energéticas de estas células.39
Oncogenes
Las mutaciones que producen oncogenes son dominantes, lo que significa que se necesitan defectuosos los dos alelos de un gen del par de cromosomas homólogos.22 Los oncogenes codifican proteínas de secreción que actúan como moléculas señales, factores de crecimiento, receptores, proteínas citoplasmáticas (proteínas G y proteína quinasas) y factores de transcripción nucleares que controlan la expresión de genes esenciales para la división celular.24,40
Los proto-oncogenes se convierten en oncogenes por mutaciones en el DNA que causan ganancia de función, lo que produce una proteína que funciona sin los eventos activadores normales o en cantidades anormales.
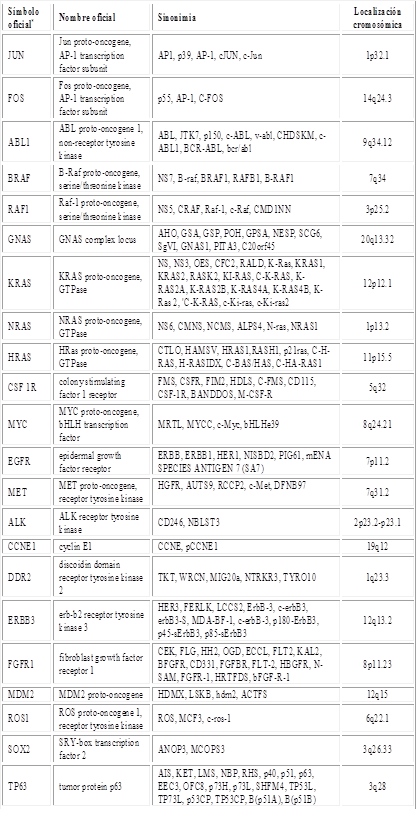
Los proto-oncogenes se activan a oncogenes por diversos mecanismos, como mutaciones puntuales, translocaciones cromosómicas, integración del genoma viral y amplificación.22 Los oncogenes que se describen en la tabla 1 no solo participan en la carcinogénesis pulmonar, también en otros cánceres como mamarios, prostáticos y digestivos; se pusieron ejemplos ilustrativos de los numerosos genes involucrados.26,38
Para ampliar sobre oncogenes se recomienda a Harold Varmus,41 premio Nobel de Fisiología o Medicina 1989 por su descubrimiento del origen celular de los oncogenes y el papel de los virus tumorales.
Genes supresores tumorales
Los genes supresores tumorales son frecuentemente inactivados por alteraciones genéticas en el ADN como mutaciones puntuales, deleciones (pérdidas) y reordenamientos de ambas copias del gen. Dentro de los eventos epigenéticos de la carcinogénesis se encuentra la metilación del ADN y a la hipoacetilación e hipermetilación de histonas.42
La metilación del promotor de los genes supresores tumorales es un sello distintivo del CP y constituye un evento precoz en el proceso de carcinogénesis43 Este proceso puede acoplarse con mutaciones o deleciones que inactivan a los genes supresores tumorales.
Los genes estabilizadores, vigilantes de genoma o cuidadores codifican proteínas que reparan los defectos genéticos de la replicación del ADN aberrante, por radiación ionizante o carcinógenos.24 Estas mutaciones originan daño irreparable en ADN o mutaciones en otros genes como proto-oncogenes y genes supresores tumorales. Entre estos genes están ATM y BRCA1 y se consideran genes supresores tumorales.
El secuenciado del genoma en 110 pacientes con SCLC reveló la casi universal pérdida de los genes supresores TP53 y RB1.5 Algunos supresores tumorales metilados en los promotores son p16INK4a, RASSF1A, APC, RARβ, CDH1, CDH13, DAPK, FHIT y MGMT.43 Los principales genes supresores tumorales implicados en el CP se describen en la tabla 2.6,29,38,40
Aplicación práctica
La detección de genes de susceptibilidad al CP tiene importancia para la identificación de biomarcadores diagnósticos y pronósticos, y para el diseño de terapias más eficaces para su tratamiento, enmarcadas en la medicina de precisión o personalizada (Fig. 2).21,32
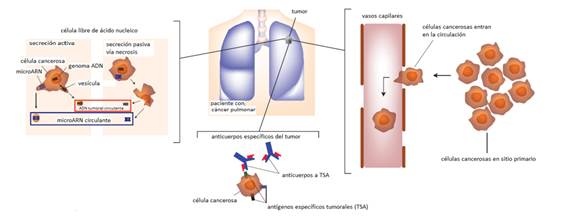
Fuente: Blandin Knight S, Crosbie PA, Balata H, Chudziak J, Hussell T, Dive C. Progress and prospects of early detection in lung cancer. Open Biol. 2017;7:170070. DOI: 10.1098/rsob.170070Licencia: https://creativecommons.org/licenses/by/4.0/
Fig. 2 Potenciales diana para biomarcadores sanguíneos de detección temprana del cáncer pulmonar.
Biomarcadores
El diagnóstico del CP se basa en los síntomas y ocurre frecuentemente cuando las posibilidades de curación son remotas. Numerosos biomarcadores de detección precoz se investigan, aunque todavía su uso clínico es limitado debido a sus bajas sensibilidad y especificidad o relevancia funcional.
En estadios avanzados las mutaciones de EGFR confieren una mayor sensibilidad a los inhibidores de tirosina quinasa (TKIs) como erlotinib, gefitinib, y afatinib. Sin embargo, las mutaciones de KRAS aparecen más en fumadores y confieren peor pronóstico.11 La presencia de una mutación de HER2 puede ser un biomarcador predictivo de la respuesta a trastuzumab en NSCLC.44
Las mutaciones de BRAF, BRCA1, EGFR, DLC1, KRAS, RET y MET se emplean para determinar el pronóstico del CP.45,46 En pacientes con NSCLC y mutaciones de BRAF ocurren metástasis a ganglios linfáticos axilares. Los ganglios linfáticos axilares no se involucran habitualmente en el CP; en consecuencia, la metástasis en estos ganglios es solo 0,6-0,7 %, aunque en pacientes con mutaciones de BRAF la incidencia es de 15 %.
Los micro-ARNs (miARN), una familia de pequeños ARN no codificantes, inhiben la traducción de ARNm y promueven su degradación.47,48 La alteración en la expresión de miRNA se implica en la patogenia de la mayoría de los cánceres.2
Los miRNAs dirigidos directamente a EGFR son miR-7, miR-27a-3p, miR-30, miR-34, miR-128, miR-133, miR-134, miR-145, miR-146, miR-149, miR-218, y miR-542-5p.47 El proto-oncogen ROS1 es otro gen diana porque miR-760 suprime su expresión y reduce la proliferación celular en células de NSCLC.47
Los cambios epigenéticos son cambios heredables en el fenotipo o en la expresión genética no atribuibles a cambios en la secuencia de ADN. Los dos principales tipos de regulación epigenética son metilación del ADN y modificación de histonas. Estos cambios también son importantes en la carcinogénesis pulmonar, ya que activan oncogenes e inhiben genes supresores tumorales. Se evalúan biomarcadores epigenéticos de CP.
La hipermetilación de promotores parece un evento inicial en la carcinogénesis pulmonar, lo que es útil en la detección precoz.43 Por ejemplo, la hipermetilación de p16INK4a se observa en lesiones precursoras de NSCLC y la metilación del promotor de PTPRN2 es un evento inicial en el adenocarcinoma y en la hiperplasia adenomatosa atípica premaligna.
Es importante mencionar que algunos de estos cambios epigenéticos precoces se detectan con técnicas de recolección de muestras no invasivas o mínimamente invasivas, una característica importante para la pesquisa del cáncer.43 Por ejemplo, la metilación aberrante del ADN se detecta en esputo, lavado broncoalveolar y saliva de pacientes con CP.
Tratamiento
Como parte de la medicina de precisión se encuentra la edición de genes que permite manipular y modificar la secuencia de genes específicos, tecnología en desarrollo con resultados prometedores; aunque, se están debatiendo mucho los principios éticos de su aplicación en humanos.
La tecnología CRISPR-Cas9 abrió una nueva era de la biotecnología con la edición genética precisa de cualquier célula.49 Investigadores chinos inyectaron linfocitos genéticamente modificados como una estrategia terapéutica para eliminar células malignas del pulmón mediante el sistema inmune. Para ese propósito, el gen PDCD1 se inactivó con CRISPR-Cas9, lo que podría ser más efectivo que el bloqueo con inmunoterapia del producto de este gen.49 Para ampliar sobre esta prometedora tecnología se recomienda a Ventura y Dow.50
Como más de 60 % de los carcinomas NSCLC expresan EGFR, este receptor se ha vuelto una importante diana terapéutica para estos tumores. Los TKIs son efectivos en mutaciones activantes de tumores en dominio tirosín quinasa del gen EGFR, por lo que algunos ensayos clínicos sugieren que los pacientes con NSCLC avanzada con estas mutaciones, en vez de quimioterapia, la mejor opción de tratamiento sería la terapia inicial con TKI.
Otros agentes que inhiben la actividad de EGFR son los anticuerpos monoclonales que se unen al receptor y funcionan como antagonistas competitivos de sus ligandos (ejemplo cetuximab). Estos agentes se unen reversiblemente a EGFR e inhiben su actividad; se unen con mayor afinidad a receptores mutados. Otros agentes se ensayan con resultados halagüeños, aunque se requiere continuar los estudios de seguridad y eficacia.
Además, los inhibidores de la angiogénesis como bevacizumab también están disponibles para el tratamiento del CP. Venetoclax se aprobó por FDA como inhibidor de la apoptosis.29
Estas terapias son prometedoras para el tratamiento personalizado del CP, aunque la resistencia medicamentosa y los efectos adversos pueden limitar su empleo clínico.
Conclusiones
El cáncer es una enfermedad genética de carácter multifactorial, en la cual los factores ambientales y genéticos interactúan en una compleja red que permite la aparición y desarrollo de este tipo de cáncer.
Dentro de los factores genéticos implicados en carcinogénesis pulmonar se destacan los oncogenes y los genes supresores tumorales.
En la carcinogénesis pulmonar se involucran los oncogenes JUN, FOS, ABL1, BRAF, RAF1, GNAS, KRAS, NRAS, HRAS, CSF 1R, MYC, EGFR, MET, ALK, CCNE1, DDR2, ERBB3, FGFR1, MDM2, ROS1, SOX2 y TP63 y los genes supresores tumorales TP53, CDKN2A, CDKN1A, RB1, CDK2AP1, ATM, ERCC2, BRCA1, CCND1, STK11, PDLIM2, PTEN, ARID1A, ASCL4, CUL3, EP300, KEAP1, KMT2D, NF1, NOTCH1, RASA1, ETD2 y SMARCA4.
Los oncogenes tienden a ser dominantes y derivan de versiones normales de genes que controlan la proliferación celular denominados proto-oncogenes, mientras los genes supresores son recesivos y requieren la inactivación de los dos alelos del gen para que se produzca el cáncer.
El conocimiento de la genética molecular del cáncer es un prerrequisito para el desarrollo y mejoría futura de las estrategias clínicas para el manejo del CP, aunque por la complejidad de las alteraciones genéticas y las interacciones con factores ambientales se requieren posteriores estudios que esclarezcan las preguntas que aún continúan sin respuesta.