Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Salud Pública
versión On-line ISSN 1561-3127
Rev Cubana Salud Pública v.32 n.2 Ciudad de La Habana abr.-jun. 2006
Universidad de Córdova, Montería, Colombia
Universidad del Sinú, Montería, Colombia
Presencia del gen de invasividad inv A en cepas de Salmonella spp. aisladas de alimentos del Caribe Colombiano
Paula Espinal Marin,1 Edgar Prieto Suárez,2 Vanessa Otero Jiménez3 y Salim Máttar Velilla4
Resumen
Objetivo: establecer la presencia del gen de invasividad invA en aislamientos de Salmonella spp. obtenidos de alimentos en la región Caribe Colombiana.
Métodos: se realizó un estudio microbiológico de control de alimentos en 4 ciudades de la región Caribe entre enero de 2002 y marzo de 2003. Se analizaron 1 300 muestras de alimentos provenientes de mercados y ventas callejeras.
Resultados: se recuperaron 74 aislamientos de Salmonella spp. , en carne de res 30 (40,5 %), embutidos 13 (17,6 %), pollo 12 (16,2 %), queso 9 (12,2 %), cerdo 6 (8,1 %) y otros 4 (5,5 %). Los serotipos más frecuentes fueron: S. anatum 14 (18,9 %), S. uganda 13 (17,6 %), S. newport 9 (12,2 %) y S. typhimurium 7 (9,5 %). El cebador invA amplificó un fragmento de 378 pb, el gen invA se detectó en 72 (97,3 %) aislamientos de Salmonella.
Conclusiones: se detectó la presencia del gen invA en los serotipos de Salmonella circulantes en alimentos en la región Caribe Colombiana. Las implicaciones epidemiológicas de estos resultados permiten sugerir a las autoridades sanitarias tomar medidas estrictas en el control, prevención y diagnóstico de la infección por Salmonella en esta región.
Palabras clave: Salmonella spp., gen inv A, alimentos, Colombia, Caribe.
Introducción
La salmonelosis es una de las principales causas de gastroenteritis en humanos y animales.1 Los reservorios de Salmonella de potencial transmisibilidad para el humano los constituyen principalmente personas y animales domésticos infectados, aguas y alimentos contaminados como carnes, huevos y productos lácteos no pasteurizados.2-4 Las infecciones por Salmonella enterica serovar Typhimurium y serovar Enteritidis son una causa importante de morbilidad y mortalidad especialmente en niños y personas inmunocomprometidas.5,6 En los Estados Unidos estos serotipos afectan aproximadamente 2 a 3 millones de personas y causan entre 500 a 2 000 muertes cada año.5,7 En Colombia existe un subregistro acerca del verdadero problema de Salmonella, tanto en identificación de reservorios, como en infecciones, morbilidad y mortalidad asociada.7 En Colombia el estudio más reciente sobre Salmonella aisladas de alimentos se realizó en la Costa Atlántica y demostró la presencia de diversos serotipos circulantes.8
El proceso de adaptación al huésped Salmonella spp. ha generado una variedad de mecanismos para colonizar, invadir, replicar y sobrevivir dentro del huésped.9 La capacidad de Salmonella spp. para adherirse y entrar a las células del epitelio intestinal es un paso esencial en el ciclo de vida de estos microorganismos, esta propiedad determina la virulencia en Salmonella, y está localizada en un grupo de genes adquiridos posiblemente por transferencia horizontal,10,11 y que pueden no estar presentes en las muestras ambientales.12 Estos genes se codifican en la denominada isla de patogenicidad 1 (SPI-1), que a su vez, codifica para un sistema de secreción tipo III requerido para la estimulación de la respuesta celular que conduce a la penetración de la bacteria en las células epiteliales, iniciación de apoptosis en macrófagos y producción de citoquinas pro-inflamatorias por células epiteliales.13-18 Estas islas con sus respectivos sistemas de secreción y sus genes pueden sufrir delecciones o perderse en las muestras ambientales.12
La importancia de esta región cromosomal en diferentes serotipos de Salmonella radica en la presencia y funcionalidad del gen invA, que codifica un componente esencial del aparato de secreción de proteínas asociadas con la invasión.2,10 El gen invA es un blanco ideal para la aplicación de métodos moleculares basados en la PCR, que permiten detectarlo en aguas y alimentos y distinguir entre aislamientos bacterianos patogénicos y no patogénicos.19,20 Este gen se encuentra en serotipos de Salmonella y puede estar ausente en muestras ambientales con una virulencia reducida que no está asociada con enfermedad.21
En Colombia no se conocen estudios que demuestren la presencia del gen inv A en los serotipos de Salmonella circulantes tanto en alimentos como en especímenes clínicos. Por ello la detección temprana del gen invA en las fuentes de infección por Salmonella spp. es de gran importancia en salud pública porque permite desarrollar acciones de control, saneamiento y prevención.
El objetivo del presente estudio fue determinar la presencia del gen de invasividad invA en aislamientos de Salmonella spp. obtenidos de alimentos en varias ciudades de la región Caribe Colombiana.
Métodos
Aislamientos bacterianos
Se realizó un estudio microbiológico de calidad de alimentos en cuatro ciudades de la región Caribe: Barranquilla (Departamento del Atlántico), Cartagena (Departamento de Bolívar), Montería (Departamento de Córdoba) y Sincelejo (Departamento de Sucre), entre enero de 2002 y marzo de 2003. Se analizaron 1 300 muestras de alimentos provenientes de mercados y ventas callejeras seleccionadas aleatoriamente.
Las muestras de alimentos tomadas de los sitios de expendio, se trasladaron refrigeradas al laboratorio del Instituto de Investigaciones Biológicas del Trópico (IIBT) de la universidad de Córdoba para ser procesadas antes de 24 h. El aislamiento de Salmonella se realizó por el método convencional de la Federal and Drug Administration ( Food and Drug Administrationa / AOAC BAM 2003. Disponible en: http:// www.cfsan.fda.gov/ebam/bam-toc.htm
Se pesaron 25 g de cada muestra de alimento que fueron inoculados en 225 mL de medios de preenriquecimiento, agua peptonada y caldo infusión cerebro corazón y se incubaron a 37 °C durante 18-24 h. Se inoculó 1 mL de cada muestra anterior en 9 mL de medio Rappaport y se incubaron a 43 °C entre 18-24 h. Se inoculó 1 mL de cada muestra en 9 mL de Tetrationato líquido y se incubaron a 37 °C durante 18-24 h. Cada muestra fue subcultivada tomando una asada de los tubos de enriquecimiento y su siembra por agotamiento en agar base XLT4 (Difco Detroit, Mi USA), XLD, SMID (Biomeriux, etoiac, France), SS, Hekctoen y Sulfito Bismuto; estas placas se incubaron a 37 °C por 18-24 h. A las colonias sospechosas de Salmonella se les realizaron pruebas bioquímicas convencionales (Urea, LIA y TSI) incubándolas a 37 °C durante 18-24 h para ser identificadas. Se confirmaron con antisueros polivalentes para Salmonella (Difco, Detroit, Mi, EE.UU.). Cada cepa se conservó por triplicado en tubos de Skin Milk a -70 ºC.
Los 74 aislamientos de Salmonella se clasificaron taxonómicamente hasta especie y serovariedad, en el Laboratorio de Microbiología del Instituto Nacional de Salud (INS) según esquemas serológicos convencionales,22 en una segunda fase, se analizaron mediante PCR para la detección del gen invA. Como control positivo para PCR se utilizó la cepa de Salmonella enterica serotipo Enteritidis portadora del gen invA y como control negativo la cepa ATCC 700603 de Klebsiella pneumoniae .
Amplificación del gen invA
Una colonia de Salmonella se suspendió en 5 mL de caldo LB (Louria Bertani) y se cultivó a 37 ºC en agitación por 24 h. A partir de este cultivo se tomaron 4 mL y se mezclaron con 196 mL de agua destilada. Se llevaron a ebullición por 5 min a 95 ºC y se centrifugaron a 12 000 r.p.m por 10 min a 4 ºC. La detección del gen invA por PCR se llevó a cabo con los iniciadores complementarios 5´ (5´GTGAAATTATCGCCACGTTCGGGCAA3´) y 3´(5´TCATCGCACCGTCAAA-GGAACC 3´), los cuales amplifican un fragmento de 378 pb con la secuencia conservada de los genes inv de Salmonella.10 Cada 100 mL de la mezcla de PCR contenía buffer PCR 10X, dNTP (2,5 mM de cada uno), 5 unidades de Taq ADN- polimerasa (Takara, Otsu, Shiga, Japón), 2 mL de los iniciadores y 50 mL de la muestra extraída. La amplificación se realizó en el termociclador (MJ Research, PTJ 100, Waltham, Massachussets, EE.UU) con 35 ciclos así: desnaturalización a 94 ºC por 1 min, asociación a 55 ºC por 1 min y extensión a 72 ºC por 1 min, seguidos por un paso de extensión final a 72 ºC por 10 min . Los productos de PCR se analizaron por electroforesis en un gel de agarosa al 1 % en buffer TBE 1X. El gel se tiñó con una solución de bromuro de etidio al 1 %. Se utilizó el marcador de peso molecular de 100 pb (Gibco BRL Rockville, Md) para la detección de los pesos moleculares de los productos de PCR.
Resultados
El análisis de los alimentos en las cuatro ciudades mostró la recuperación de Salmonella spp., con mayor frecuencia, en carne de res 30 (40,5 %), seguido por alimentos embutidos 13 (17,6 %) y en pollo 12 (16,2 %) (tabla 1).
Los serotipos de Salmonella más frecuentes correspondieron a S. anatum 14 (18,9 %), S. uganda 13 (17,6 %), S. newport 9 (12,2 %) y S. typhimurium 7 (9,5 %) (tabla 2).
Tabla 1. Aislamiento de Salmonella spp. según tipo de alimento y lugar de origen
| Ciudad de origen | Alimentos muestreados No. (%) | Tipo de alimento, No.y (%) de aislamiento de Salmonella, por ciudad | ||||||||
| Agua | Arepa huevo | Carne de res | Cerdo | Embutido | Pollo | Queso | Vísceras | Total | ||
| Barranquilla | 293 (22,54) | 1(3,8) | 1(3,8) | 13 (50) | 1(3,8) | 2 (7,7) | 3 (11,5) | 4 (15,4) | 1(3,8) | 26 (100) |
| Cartagena | 266 (20,46) | 0 (0) | 1(6,3) | 6 (37,5) | 1(6,3) | 2 (12,5) | 5 (31,3) | 1 (6,3) | 0 (0) | 16 (100) |
| Montería | 417 (32,08) | 0 (0) | 0 (0) | 8 (34,8) | 3 (13) | 9 (39,1) | 0 (0) | 3 (13) | 0 (0) | 23 (100) |
| Sincelejo | 324 (24,92) | 0 (0) | 0 (0) | 3 (33,3) | 1(11,1) | 0 (0) | 4 (44,4) | 1 (11,1) | 0 (0) | 9 (100) |
| Total | 1300 (100) | 1(1,4) | 2 (2,7) | 30 (40,5) | 6 (8,1) | 13 (17,6) | 12 (16,2) | 9 (12,2) | 1(1,4) | 74 (100) |
Tabla 2. Serotipos de Salmonella y lugar de origen
No. (%)
| Serotipo | Barranquilla | Cartagena | Montería | Sincelejo | Total |
| S. anatum | 7 (26,9) | 5 (31,3) | 2 (8,7) | 0 (0) | 14 (18,9) |
| S. brandenburg | 0 (0) | 0 (0) | 2 (8,7) | 0 (0) | 2 (2,7) |
| S. derby | 2 (7,7) | 0 (0) | 0 (0) | 0 (0) | 2 (2,7) |
| S. gaminara | 0 (0) | 0 (0) | 4 (17,4) | 0 (0) | 4 (5) |
| S. isangi | 3 (11,5) | 1 (6,3) | 0 (0) | 0 (0) | 4 (5,4) |
| S. muenchen | 0 (0) | 2 (12,5) | 0 (0) | 1 (11,1) | 3 (4,1) |
| S. newport | 0 (0) | 1 (6,3) | 7 (30,4) | 1 (11,1) | 9 (12,2) |
| S. rubislaw | 1 (3,8) | 1 (6,3) | 0 (0) | 0 (0) | 2 (2,7) |
| S. sandiego | 1 (3,8) | 0 (0) | 0 (0) | 2 (22,2) | 3 (4,1) |
| S. typhimurium | 2 (7,7) | 0 (0) | 5 (21,7) | 0 (0) | 7 (9,5) |
| S. uganda | 5 (19,2) | 4 (25) | 1 (4,3) | 3 (33,3) | 13 (17,6) |
| Otras* | 5 (19,2) | 2 (12,5) | 2 (8,7) | 2 (22,2) | 11 (14,9) |
| Total | 26 (100) | 16 (100) | 23 (100) | 9 (100) | 74 (100) |
* Salmonella spp, S. tennessee, S. sinstorf, S. senftenberg, S. saintpaul, S. kapemba, S. gine, S. galiema, S. duesseldorf, S. brenedy, S. agona.
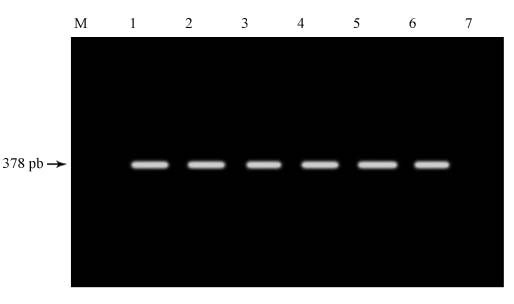
El cebador inv A amplificó un fragmento de 378 pb correspondiente a los productos de PCR de Salmonella portadoras del gen inv A (fig.).
Fig. PCR para detección del gen invA. Carriles: 1. S. anatum , 2. S. uganda, 3. S. derby, 4.
S. gaminara, 5. S. muenchen. 6. Control positivo Salmonella enterica serotipo Enteritidis portadora
del gen invA , 7. Control negativo cepa ATCC 700603 de Klebsiella pneumoniae. M. Marcador
de peso molecular de 100 pb.
De 74 aislamientos de Salmonella analizadas, 72 (97,3 %) fueron positivas para la detección del gen invA. Los dos aislamientos que no presentaron amplificación correspondieron a Salmonella enterica serotipo Typhimurium y Salmonella enterica serotipo Sandiego. Los controles positivo y negativo mostraron los resultados esperados. La distribución del gen en los serotipos de Salmonella se resume en la tabla 3.
Tabla 3. Distribución del gen invA por serotipos de Salmonella
| No. | Serotipo | Tipo de muestra | Lugar de origen | PCR gen invA | No. | Serotipo | Tipo de muestra | Lugar de origen | PCR gen invA |
| 10 | S. anatum | Carne molida | Montería | + | 4 | S. typhimurium | Chorizo | Montería | + |
| 17 | S. anatum | Cerdo | Montería | + | 23 | S. typhimurium | Cerdo | Montería | + |
| 24 | S. anatum | Carne molida | Barranquilla | + | 55 | S. typhimurium | Pollo | Barranquilla | + |
| 27 | S. anatun | Carne molida | Barranquilla | + | 56 | S. typhimurium | Queso | Barranquilla | + |
| 28 | S. anatun | Queso | Barranquilla | + | 93 | S. typhimurium | Carne molida | Montería | + |
| 31 | S. anatun | Carne molida | Barranquilla | + | 96 | S. typhimurium | Carne molida | Montería | + |
| 32 | S. anatun | Carne molida | Barranquilla | + | 9 | S. gaminara | Chorizo | Montería | + |
| 34 | S. anatun | Carne molida | Barranquilla | + | 11 | S. gaminara | Albóndiga | Montería | + |
| 60 | S. anatum | Salchicha | Cartagena | + | 12 | S. gaminara | Queso | Montería | + |
| 65 | S. anatum | Pollo | Cartagena | + | 13 | S. gaminara | Chorizo | Montería | + |
| 68 | S. anatum | Carne | Barranquilla | + | 45 | S. isangi | Pollo | Cartagena | + |
| 76 | S. anatum | Pollo | Cartagena | + | 69 | S. isangi | Mondongo | Barranquilla | + |
| 77 | S. anatum | Pollo | Cartagena | + | 73 | S. isangi | Pollo | Barranquilla | + |
| 78 | S. anatum | Carne molida | Cartagena | + | 74 | S. isangi | Queso | Barranquilla | + |
| 1 | S. uganda | Chorizo | Montería | + | 41 | S. muenchen | Carne molida | Cartagena | + |
| 26 | S. uganda | Carne molida | Barranquilla | + | 44 | S. muenchen | Carne | Cartagena | + |
| 35 | S. uganda | Carne molida | Barranquilla | + | 82 | S. muenchen | Carne molida | Sincelejo | + |
| 43 | S. uganda | Cerdo | Cartagena | + | 36 | S. sandiego | Carne molida | Barranquilla | + |
| 48 | S. uganda | Pollo | Sincelejo | + | 83 | S. sandiego | Pollo | Sincelejo | + |
| 54 | S. uganda | Carne molida | Cartagena | + | 84 | S. sandiego | Pollo | Sincelejo | - |
| 58 | S. uganda | Pollo | Barranquilla | + | 30 | S. derby | Arepa huevo | Barranquilla | + |
| 59 | S. uganda | Carne molida | Barranquilla | + | 61 | S. derby | Morcilla | Barranquilla | + |
| 66 | S. uganda | Chorizo | Cartagena | + | 20 | S. brandenburg | Carne | Montería | + |
| 67 | S. uganda | Pollo | Cartagena | + | 79 | S. brandenburg | Carne | Montería | + |
| 72 | S. uganda | Carne molida | Barranquilla | + | 40 | S. rubislaw | Queso | Cartagena | + |
| 85 | S. uganda | Carne molida | Sincelejo | + | 81 | S. rubislaw | Agua | Barranquilla | + |
| 88 | S. uganda | Pollo | Sincelejo | + | 70 | S. gine | Queso | Barranquilla | + |
| 5 | S. newport | Chorizo | Montería | + | 71 | S. brenedy | Chinchurria | Barranquilla | + |
| 6 | S. newport | Queso | Montería | + | 38 | S. duesseldorf | Carne | Cartagena | + |
| 7 | S. newport | Carne | Montería | + | 25 | S. senftenberg | Chorizo | Barranquilla | + |
Aislamientos positivos gen invA 72 (97,3 %), negativos 2 (2,7 %).
Discusión
El presente trabajo es un estudio pionero en Colombia en la detección del gen invA en Salmonella aislada de alimentos.
El número de personas con salmonelosis se ha incrementado en las últimas décadas, es una de las causas principales de enfermedad entérica bacteriana, en humanos y en animales, con una tasa elevada de morbilidad y mortalidad, tiene, además, una alta repercusión económica en el sentido negativo. Lo anterior ha obligado a la formulación de nuevas medidas preventivas, de control y de diagnóstico adecuadas para detectar el microorganismo de manera rápida y oportuna tanto en alimentos contaminados, en reservorios, así como en las personas infectadas.
En relación con los reportes de pacientes en Colombia, para el año 2002 se notificaron 6 556 enfermos de intoxicación alimentaria en todo el país, de ellos el 22 % (1 442) correspondió al distrito capital de Bogotá, el 27,8 % a las entidades territoriales de Santa Marta y Cesar en la costa Atlántica (1 822 pacientes) y para la zona de influencia del presente estudio (Atlántico, Córdoba, Cartagena y Sucre) se notificaron 375 lo que representa el 5,7 %. También se registraron 256 enfermos de fiebre tifoidea y paratifoidea, de los cuales el Departamento de Sucre representa el 23 % y el del Atlántico el 5,9 % del total de los enfermos. Llama la atención que no se registraron datos de Sucre (Sistema Nacional de Vigilancia en Salud Pública SIVIGILA 2002, Ministerio de la Protección Social , Instituto Nacional de Salud). No se registraron enfermos en Cartagena para los años 2001 y 2002, tampoco en Córdoba para el año 2002 (tabla 4).
Tabla 4. Enfermos notificados en el Caribe Colombiano de intoxicación alimentaria, fiebre tifoidea y paratifoidea
| Departamento | Intoxicación alimentaria | Fiebre tifoidea y paratifoidea | ||
| Año 2001 | Año 2002 | Año 2001 | Año 2002 | |
| Atlántico | 43 | 24 | 11 | 15 |
| Cartagena | 41 | 5 | * | * |
| Córdoba | 11 | 346 | 1 | * |
| Sucre | * | * | 2 | 59 |
| Total | 95** | 375** | 14*** | 74*** |
*Sin dato
** Corresponden a los enfermos reportados en el área de influencia del estudio.
Total de enfermos 6 556 en el país año 2002.
*** Corresponden a los enfermos reportados en el área de influencia del estudio.
Total de enfermos 256 en el país año 2002.
De acuerdo con los resultados obtenidos en el presente estudio, y los reportes de enfermos de intoxicación alimentaria, fiebre tifoidea y paratifoidea, es claro que se deben realizar programas de control sanitario, de vigilancia epidemiológica, fortalecer los servicios de salud en la determinación de los diagnósticos etiológicos e implantar sistemas de registro de información que sirvan de base para los programas de vigilancia (Sistema Nacional de Vigilancia en Salud Pública SIVIGILA 2002, Ministerio de la Protección Social , Instituto Nacional de Salud ).
Recientemente el análisis genético de los factores de virulencia en bacterias patógenas ha mostrado gran utilidad ya que muchos de los genes asociados como los de invasividad están estrechamente relacionados y se encuentran codificados en regiones especiales como la isla de patogenicidad 1 (SPI1), que confiere a Salmonella la capacidad para invadir las células epiteliales.2,18 Es importante establecer si las cepas contienen sistemas de secreción tipo III específicos de SPI1, ya que los genes presentes en estos sistemas codifican componentes particulares que son altamente conservados.3,12 Con la PCR se puede detectar en Salmonella genes que median la invasividad invA, agfA, iagAB, invF, invH 2,3,23 a partir de heces y alimentos, además, permite diagnosticar la infección y la contaminación en menor tiempo comparado con los métodos de cultivo tradicionales y la confirmación serológica, que pueden tomar de 5 a 7 días.4
En el presente estudio se realizó la amplificación del gen invA, uno de los genes descritos en todas las cepas invasivas de Salmonella.6,10 Se encontró que 72 de 74 (97,3 %) cepas de Salmonella de diferentes serotipos obtenidas de alimentos, fueron positivas para el gen invA. Las cepas negativas para la detección del gen correspondieron a Salmonella enterica serotipo Sandiego y Salmonella enterica serotipo Typhimurium, en este último serotipo se ha descrito frecuentemente su presencia. Este resultado podría sugerir la pérdida o delección de la isla de patogenicidad SPI1 que puede afectar la expresión del gen debido a una alteración en el sitio de inserción en el cromosoma y/o por remoción de los genes contenidos en la isla, posiblemente por la transferencia horizontal de estos bloques de genes de virulencia.12,18
Por otra parte, de acuerdo con el sistema de vigilancia del Ministerio de Salud en Colombia se ha presentado en los últimos 5 años una tasa para la enfermedad diarreica aguda (EDA) mayor de 1 500 por 100 000 habitantes. El mismo sistema informa que el 50 % de los egresos hospitalarios por EDA se deben a Salmonella spp. y a Shigella,7 este panorama podría agravarse cada día con la presencia de brotes en los cuales el diagnóstico preciso y temprano es poco común. Por lo tanto, el uso de la PCR podría incluir sólo un incremento limitado en el costo del procesamiento de la muestra que al realizar el análisis costo-beneficio produciría una mejoría en la salud humana con un diagnóstico rápido y más sensible.3,4,6 Adicionalmente, debe sumarse la posibilidad de analizar directamente las fuentes de infección como alimentos, aguas y muestras fecales.6,24
De otro lado, se deben llevar a cabo estudios epidemiológicos moleculares que permitan comparar y correlacionar las cepas aisladas de los alimentos y las aisladas de los enfermos, lo que permitiría establecer con exactitud la circulación de los serotipos aislados de alimentos con capacidad de invasividad y demostrar su diseminación clonal hacia los humanos. Establecer esta relación puede contribuir al control de la salmonelosis por programas de salud pública.
En conclusión, el trabajo permitió detectar la presencia del gen invA en los serotipos de Salmonella circulantes en alimentos en la región Caribe Colombiana, los cuales son similares a los encontrados en otras partes del mundo. Las implicaciones epidemiológicas de estos resultados permiten sugerir a las autoridades sanitarias tomar medidas estrictas en el control, prevención y diagnóstico de la infección por Salmonella en esta región. Este estudio también propone la detección del gen invA directamente en muestras clínicas, reservorios y alimentos como un método posible para el control de las salmonelosis en el país.
Summary
Presence of the invasive gene invA in Salmonella spp. strains isolated from food in several cities of the Colombian Caribbean area
Objective: to establish the presence of invasive gene invA in Salmonella spp. strains obtained from food in several cities of the Colombian Caribbean area.
Methods: from January 2002 to March 2003, a microbiological study of quality control of food was carried out in four cities of the Colombian Caribbean area. One thousand and three hundred food samples were analyzed in fast food outlets located in city squares or markets.
Results: seventy four isolates of Salmonella were recovered: 30 (40.5) in meat; 13 (17.6 %) in sausage; 12 (16.2 %) in chicken; 9 (12.2 %) in cheese; 6 (8.1 %) in pork and 4 (5.5 %) in other types of food. The most frequently isolated serotypes were S.anatum in 14 (18.9 %), S.uganda in 13 (17.6 %), S. newport in 9 (12.2 %) y S. typhimurium in 7 (9.5 %). The invA primer amplified 378 pb fragment, invA gene was detected in 72 (97.3 %) Salmonella isolates.
Conclusions: it was possible to detect the invA gene in circulating serotypes of Salmonella isolates obtained from food in the Colombian Caribbean area, the epidemiological implications allow the health authorities to take measure for the prevention, control and diagnosis of Salmonella infection in the Colombian Caribbean area.
Key words: Salmonella spp., invA gene, food, Colombia, Caribbean.
Referencias bibliográficas
1. Brenner F, Villar R, Angulo F, Tauxe R, Swaminathan B. Salmonella nomenclature. J Clin Bacteriol. 2000;38:2465-7.
2. Guo X, Chen J, Beuchat L, Brackett R. PCR detection of Salmonella enterica serotype Montevideo in and on raw tomatoes using primers derived from hilA. Appl Environ Microbiol. 2000;66:5248-52.
3. Gooding C, Coudary P. Comparison of different primers for rapid detection of Salmonella using the polymerase chain reaction. Mol Cell Probes. 1999;13:341-7.
4. Ferretti R, Mannazzu I, Cocolin L, Comi G, Clementi F. Twelve–hour PCR-based method for detection of Salmonella in food. Appl Environ Microbiol. 2001;67:977-8.
5. Chiu C, Su L, Chu C. S almonella enterica serotype Cholerasuis: epidemiology, pathogenesis, clinical disease, and treatment. Clin Microbiol Rew. 2004;17:311-22.
6. Chiu C, Ou J. Rapid identification of Salmonella serovars in feces by specific detection of virulence genes, invA and spvC, by an enrichment broth culture-multiplex PCR combination assay. J Clin Microbiol. 1996;34:2619-22.
7. Instituto Nacional de Salud. Otros Eventos de notificación obligatoria: segundo semestre 2003: EDA, IRA, ETA, rabia y meningitis. Inf Quinc Epidemiol Nac. 2003;15:241-56.
8. Durango J, Arrieta G, Máttar S. Presencia de Salmonella en un área del caribe colombiano. Un riesgo para la salud pública. Biomédica. 2004;24:69-96.
9. Hardt W, Galàn J. A secreted Salmonella protein with homology to an avirulence determinant of plant pathogenic bacteria. Proc Natl Acad Sci USA. 1997;94:9887-92.
10. Galàn J, Curtiss R III. Distribution of the invA, -B, -C, and -D genes of Salmonella typhimurium among other Salmonella serovars: invA mutants Salmonella typhi are deficient for entry into mammalians. Infect Immun. 1991;59:2901-8.
11. Neidhardt F. Escherichia coli and Salmonella. In: Neidhardt F, Altman E, Roth J, Hessel A, Sandersaon K, editors. Transposons currently in use in genetic analysis of Salmonella species. 2sd ed. V2. Washington: ASM Press;1996.p.2613-37.
12. Mecsas J, Strauss J. Molecular mechanisms of bacterial virulence: type III secretion and pathogenicity islands. Emerg Infects Dis. 1996;2:271-88.
13. Pancetti A, Galan J. Characterization of the mut S -proximal region of the Salmonella typhimurium SPI-1 identifies a group of pathogenicity island-associated genes. FEMS Microbiol Lett. 2001;197:203-8.
14. Amavisit P, Lightfoot D, Browning G, Markham P. Variation between pathogenic serovars within Salmonella pathogenicity islands. J Bacteriol. 2003;185:3624-35.
15. Altier C, Suyemoto M, Lawhon S. Regulation of Salmonella enterica serovar Typhimurium invasion genes by csrA. Infect Immun. 2000;68:6790-7.
16. Galan J, Ginocchio C, Costeas P. Molecular and functional characterization of the Salmonella invasion gene invA : homology of invA to members of a new protein family. J Bacteriol. 1992;174:4338-49.
17. Sukhan A, Kubori T, Galan J. Synthesis and localization of the Salmonella SPI – 1 type III secretion needle complex proteins Prg I and Prg J. J Bacteriol. 2003; 185:3480-3.
18. Hensel M, Shea J, Bäumler A, Gleeson C, Blattner F, Holden D. Analysis of the boundaries of Salmonella pathogenicity island 2 and the corresponding chromosomal region of Escherichia coli K–12. J Bacteriol. 1997;179:1105-11.
19. Boyd E, Li J, Ochman H, Selander R. Comparative genetics of the inv-spa invasion gene complex of Salmonella enterica . J Bacteriol. 1997;179:1985-91.
20. Ziemer C, Steadham S. Evaluation of the specificity of Salmonella PCR primers using various intestinal bacterial species. Lett Appl Microbiol. 2003;37:463-9.
21. Ginocchio C, Rahn K, Clarke R, Galan J. Naturally occurring deletions in the centisome 63 pathogenicity island of environmental isolates of Salmonella spp. Infect Immun. 1997;65:1267-72.
22. Pezzlo M. Aerobic bacteriology. In: Isenberg H, editor. Clinical Microbiology Procedures Handbook. Washington: ASM Press; 2004.
23. Gentry-Weeks C. Identification of two phylogenetically related organisms from heces by PCR for detection of Salmonella. J Clin Microbiol. 2002;40:1487-92.
24. Baümler A, Heffron F, Reissbrodt R. Rapid detection of Salmonella enterica with primers specific for iroB. J Clin Microbiol. 1997;35:1124-30.
Recibido: 31 de mayo de 2005. Aprobado: 1 de octubre de 2005.
Salim Máttar Velilla. Universidad de Córdoba, Instituto de Investigaciones Biológicas del Trópico, Facultad de Medicina Veterinaria. Montería, Córdoba, Colombia. Tel/Fax: 57-4-560710, e-mail: mattarsalim@hotmail.com
1Bacterióloga MSc. Profesora-investigadora.
2Médico Infecciones y Salud en el Trópico MSc. Profesor-investigador.
3Bacterióloga. Profesora-investigador.
4Microbiólogo PhD. Profesor-investigador.