Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión On-line ISSN 2224-4697
Rev. Protección Veg. v.25 n.1 La Habana ene.-abr. 2010
CARACTERIZACIÓN BIOQUÍMICA DE AISLAMIENTOS DE Trichoderma spp. PROMISORIOS COMO AGENTES DE CONTROL BIOLÓGICO. I. EXPRESIÓN DE ACTIVIDAD QUITINASA
BIOCHEMICAL CHARACTERIZATION OF PROMISORY ISOLATES OF Trichoderma spp. AS BIOLOGICAL CONTROL AGENTS. I. EXPRESSION OF CHITINASE ACTIVITY
Ivonne González*, Danay Infante*, Belkis Peteira*, Benedicto Martínez*, Yailén Arias*, Noyma González*, Ileana Miranda**
*Grupo de Fitopatología y **Grupo de Plagas Agrícolas. Dirección Protección de Plantas. Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, La Habana, Cuba. Correo electrónico: marquetti@censa.edu.cu
RESUMEN
La mayoría de las especies del género Trichoderma son utilizadas como agentes de control biológico para el manejo de enfermedades causadas por hongos fitopatógenos de los géneros Phytophthora, Rhizoctonia, Sclerotium, Pythium y Fusarium. Esto es posible debido a la capacidad de estos de secretar enzimas hidrolíticas, tales como las quitinasas. Los aislamientos de Trichoderma spp. presentan diferente nivel de secreción de quitinasas, por lo que resulta de especial interés contar con indicadores que permitan seleccionar aquellos aislamientos más promisorios como agentes de control biológico. En este trabajo se evaluó la dinámica de inducción de las enzimas quitinasas de diez aislamientos de Trichoderma spp., en tres medios líquidos diferentes: medio basal, medio basal suplementado con quitina al 0,5% y medio basal suplementado con gelatina al 0,2%. La actividad enzimática se evaluó en el sobrenadante al primer, tercer, quinto y séptimo días de cultivo. Los aislamientos mostraron diferentes niveles de actividad quitinasa, los que dependieron del medio de cultivo y del aislamiento analizado. El medio con quitina resultó ser el más ventajoso para estimular la inducción de las quitinasas.
Palabras clave: Trichoderma; quitinasas; control biológico; caracterización bioquímica
ABSTRACT
Most of the Trichoderma species fungi are used as biological control agents for the management of the diseases caused by phytopathogen fungi of the genera Phytophthora, Rhizoctonia, Sclerotium, Pythium and Fusarium. It is possible due to the capacity of these fungi to secrete hydrolytic enzymes such as chitinases. Trichoderma isolates present different secretion levels of chitinase enzymes, thus it is of special interest to reckon on indicators that allow selecting the most promissory isolates as biological control agents. In this work, the dynamics of chitinase induction of ten Trichoderma spp. isolate was evaluated in three liquid media: basal medium, basal medium supplemented with chitin 0,5% and basal medium supplemented with gelatine 0,2%. The enzymatic activity was determined in the supernatants at the first, third, fifth and seventh days of the cultures. The isoaltes showed different levels of chitinase activity depending on the culture medium and isolate analyzed. The medium with chitin turned out to be the most advantageous in stimulating chitinase induction and, therefore, for the selection of the best candidates as biological control agents.
Key words: Trichoderma; chitinases; biological control; biochemical characterization
INTRODUCCIÓN
Las especies del género Trichoderma han sido ampliamente estudiadas por sus efectos como agente de control biológico de hongos fitopatógenos (1). Se han identificado muchos aislamientos con aplicaciones potenciales en el manejo de los hongos fitopatógenos en diversos cultivos de importancia económica y se emplean exitosamente como bioplaguicidas en casas de cultivo y en aplicaciones en campo (2).
Las especies de Trichoderma poseen diversos mecanismos de acción en el control de los hongos fitopatógenos, que incluyen la competencia por el espacio y los nutrientes, el micoparasitismo, la producción de compuestos inhibitorios (3), la inactivación de enzimas del patógeno (4) y la resistencia inducida (5). El micoparasitismo es un proceso que limita el crecimiento y la actividad del hongo fitopatógeno debido al ataque de Trichoderma a la hifa hospedante mediante enrollamientos, ganchos y cuerpos tipo apresorios, penetrando en la pared de la célula por la acción de las enzimas hidrolíticas (3). Estas enzimas son principalmente las quitinasas y las glucanasas que degradan parcialmente la pared celular de diversos hongos (3), nematodos (6) e insectos (7).
Las enzimas quitinasas y glucanasas pueden ser inducidas artificialmente, cuando estos hongos son cultivados en medios líquidos suplementados con monómeros de quitina N-acetilglucosamina o con polímeros tales como laminarina, quitina o paredes celulares de hongos (3). Los aislamientos de Trichoderma producen las enzimas hidrolíticas en diferentes niveles lo cual influye sobre la capacidad antagónica de estos. Por ello, el objetivo del trabajo fue determinar la dinámica de inducción de las enzimas quitinasas de un grupo de aislamientos de Trichoderma promisorios como agentes de control biológico.
MATERIALES Y MÉTODOS
Cepas y medios de inducción
Para este estudio se emplearon los aislamientos de Trichoderma 1, 13, 17, 25, 75, 78, 79, 85, 90 y TS3, procedentes del cepario del Laboratorio de Micología Vegetal del Centro Nacional de Sanidad Agropecuaria (CENSA) seleccionados por poseer alta capacidad antagónica (8). Todas los aislamientos se conservaron en medio Agar-Malta (Biocen) a 4ºC y para su uso se subcultivaron en medio PDA (Papa Dextrosa Agua, Biocen) en placas Petri de 9 cm contentivas PDA, las cuales se incubaron a 28ºC durante tres días, momento
en que se tomó el micelio para el desarrollo del experimento de dinámica de inducción.
Inducción de proteínas en medio líquido
Para la inducción de proteínas en medio líquido se ensayaron tres variantes diferentes que contenían compuestos con conocido efecto inductor de la actividad de las enzimas hidrolíticas: medio basal líquido que contenía extracto de levadura 1g.L-1 y peptona 4g.L-1, medio basal líquido suplementado con quitina 5g.L-1 y medio basal líquido suplementado con gelatina al 0,2% (p/v) (9). Los dos primeros medios se esterilizaron a 120ºC durante 20 min, mientras que el medio que contenía gelatina se esterilizó a 115ºC durante 15 min.
Para todas las variantes, se dispensó 20mL del medio en frascos estériles de 100mL y se inocularon con cuatro discos de micelio de 5mm de diámetro de la periferia de la colonia pura de cada aislamiento. Posteriormente, se incubaron estáticamente a 28ºC, en la oscuridad. Para cada tratamiento se realizaron tres repeticiones y tres réplicas. Los frascos se retiraron de la incubadora al primer, tercer, quinto y séptimo días, después de la inoculación y se conservaron a -20ºC, hasta su utilización en los análisis enzimáticos.
Cuantificación de proteínas
Se utilizó el sobrenadante para realizar la determinación de la concentración de proteínas totales por el método descrito por Bradford (10), realizando las lecturas de la absorbancia a 595nm del complejo proteína-Azul de Coomasie G-250 en un espectrofotómetro (Lasso Spec III, Lasso Biotech LTDA), a partir de una solución patrón de 1mg.mL-1 de albúmina de suero bovina (BSA) para la curva patrón.
Ensayos enzimáticos
Se determinó la actividad quitinasa con 0,2 mL de quitina coloidal 10 mg.mL-1 preparada acorde a Boller et al. (11), que se mezcló con 0,5 mL del sobrenadante de los frascos. La mezcla se incubó a 37 ºC durante una hora y posteriormente se añadió 0,1 mL de tetraborato de sodio 0,8 M pH 8,8. Posteriormente, se centrifugaron a 10000 rpm durante cinco minutos. De cada tubo se tomaron 500 µL del sobrenadante, los que se pasaron a tubos de cristal donde se calentaron a 100ºC por un período de tres minutos. Por último, se les adicionó 1 mL de p-dimetilaminoben-zaldehído y se incubó con esta solución a 38ºC durante 20 minutos. La curva patrón se determinó utilizando N-acetilglucosamina, a partir de una solución madre de 1mg.mL-1 a la cual se le realizó el mismo procedimiento que a las muestras. La lectura se realizó a 585 nm. El cálculo se realizó según la expresión:
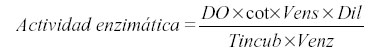
Donde DO: densidad óptica; cot: cotangente del ángulo de la curva patrón; Vens: volumen de ensayo; Dil: dilución; Tincub: tiempo de incubación y Venz: volumen de enzima. Las unidades de actividad fueron: mmoles de producto formado.min-1.mL-1 de enzima.
La actividad específica se determinó según la expresión:
Actividad específica = Actividad enzimática / concentración de proteínas (mg.mL-1).
Para determinar el medio y el día que permiten identificar cual de estos aislamientos presenta mayores niveles de concentración de proteínas y/o actividad quitinasa, se realizó el Análisis Multivariado de Componentes Principales por el paquete estadístico InfoStat (12).
RESULTADOS Y DISCUSIÓN
Los medios que más favorecen la expresión de las proteínas totales muestran el menor ángulo con respecto a cada aislamiento, por ejemplo: el aislamiento 75 mostró mayores niveles de concentración de proteínas totales en el medio basal al tercer día de cultivo, mientras que el aislamiento 13 se encuentra igualmente favorecido por los medios basal y el basal suplementado por quitina al séptimo y tercer día de cultivo, respectivamente (Fig. 1).
El componente 1, conformado por los medios basal suplementado con gelatina al tercer y quinto día de cultivo y basal al tercer día de cultivo, explicó el 31,8% de la varianza total, mientras que las dos variables del componente 2 (medio basal suplementado con quitina al tercer y séptimo días de cultivo) explicaron el 26,8% de la variación para un total acumulado en los componentes principales de un 58,6%.
Según el análisis de componentes principales para la concentración de proteínas totales de los aislamientos utilizados, se conformaron cuatro grupos de la siguiente manera: el grupo 1 formado por el aislamiento 75, grupo 2: aislamientos 1, 13, 17 y 25, grupo 3: aislamientos 78, 79, 85 y 90 y el grupo 4 integrado por el aislamiento TS3.
Los mayores niveles de concentración de proteínas totales se alcanzaron con el aislamiento 75 al tercer día de cultivo en el medio basal, seguido del medio suplementado con gelatina al tercer y quinto días de cultivo. Por otra parte, el aislamiento TS3 presentó los niveles más bajos de concentración de las proteínas totales, fundamentalmente en el medio basal al séptimo día de cultivo. Los aislamientos correspondientes a los grupos 2 y 3 mostraron comportamientos similares e intermedios entre los grupos 1 y 4.
El análisis de los componentes principales para la actividad enzimática específica quitinasa permitió agrupar los aislamientos de la siguiente manera: grupo 1 aislamiento 75, grupo 2 aislamiento 78, grupo 3 aislamientos 1, 79 y 85, grupo 4 aislamientos 13, 17, 25 y 90 y grupo 5 aislamiento TS3 (Fig. 2). Los medios basal, al quinto día, y basal suplementado con quitina, en el primer día de cultivo indujeron los mayores niveles de actividad quitinasa en el aislamiento 75, con respecto al resto de los aislamientos estudiados. El aislamiento 78 también mostró elevados niveles de actividad quitinasa en el medio suplementado con quitina al séptimo día. Estos resultados concuerdan con De la Cruz et al. (13), los cuales obtuvieron en experimentos realizados con diferentes fuentes de carbono, los mayores niveles de actividad quitinasa en medios que contenían quitina como suplemento.
El grupo 5 constituido por el aislamiento TS3, presentó los niveles más bajos de actividad quitinasa, mientras que los grupos 3 y 4 alcanzaron valores de actividad enzimática intermedios entre los grupos 2 y 5. El aislamiento TS3 ha sido utilizado como una alternativa al uso del bromuro de metilo en el control de nematodos fitoparásitos del género Meloidogyne (14).
Los medios basal al séptimo día de cultivo y basal suplementado con quitina al tercer y séptimo día de cultivo explicaron el 28,5 % de la varianza acumulada. Estos resultados demostraron la eficiencia de estos medios en la inducción de este sistema enzimático y podría explicar la eficacia de los aislados 75 y 78 contra Sclerotium y Rhizoctonia patógenos que contienen quitina en su pared (8). Estos medios podrían emplearse para realizar la selección de los aislamientos más promisorios como agentes de control biológico por inducir los mayores niveles de actividad quitinasa. A pesar de que los medios suplementados con gelatina al tercer y quinto días de cultivo indujeron mayores niveles de proteínas totales no sucedió de igual forma con la actividad quitinasa. Es probable que esta fuente de carbono por poseer estructura proteica favorezca de forma mayoritaria la inducción de otras proteínas degradantes de la pared celular como las glucanasas y las proteasas (15, 16). Por otra parte, el medio basal también indujo niveles significativos de concentración de proteínas totales, así como de actividad enzimática específica de las enzimas quitinasa, por lo que podría ser un medio eficaz para la selección de los aislamientos.
En los hongos, las quitinasas cumplen una función fisiológica en la división celular y la diferenciación (17), además de facilitar la penetración al hospedante (1) por degradación de la pared celular de estos. Lorito et al. (19) describen la posible función de las enzimas quitinolíticas en el control biológico al demostrar que la transferencia de genes que codifican para la endoquitinasa de T. harzianum Rifai (P1) en tabaco y papa producen un incremento de la resistencia a fitopatógenos.
La variabilidad encontrada entre las actividades enzimáticas quitinasa indica la utilidad de este parámetro en la selección de los aislamientos de Trichoderma como agentes de control biológico frente a diferentes dianas. No obstante, esto no es suficiente para establecer un sistema de selección de aislamientos de Trichoderma promisorios debido a que las quitinasas no son las únicas enzimas involucradas en el antagonismo (4) por lo que es necesario, además, realizar la dinámica de inducción de enzimas tales como glucanasas y proteasas.
REFERENCIAS
1. Jayalakshmi SK, Raju S, Usha Rani S, Benagi VI, Sreeramulu K. Trichoderma harzianum Rifai as a potential source for lytic enzymes and elicitor of defense responses in chickpea (Cicer arietinum L.) against wilt disease caused by Fusarium oxysporum f. sp. ciceri. Aust J Crop Sci. 2009,3(1):44-52.
2. Küçük Ç, Kývanç M. Mycoparasitism in the biological control of Gibberella zeae and Aspergillus ustus by Trichoderma harzianum strains. J Agric Technol. 2008;4(2):49-55.
3. Zeilinger S, Omann M. Trichoderma Biocontrol: Signal Transduction Pathways Involved in Host Sensing and Mycoparasitism. Gene Reg Syst Biol. 2007;1:227-234.
4. Verma M, Brar SK, Tyagi RD, Surampalli RY, Valéro JR. Antagonistic fungi, Trichoderma spp.: Panoply of biological control. Biochem Eng J. 2007;371-20.
5. Kapulnik Y, Chet I. Induction and accumulation of PR proteins activity during early stages of root colonization by the mycoparasite T. harzianum strain T-203. Plant Physiol Biochem. 2000;38:863-873.
6. Sharma P, Pandey R. Biological control of root-knot nematode; Meloidogyne incognita in the medicinal plant; Withania somnifera and the effect of biocontrol agents on plant growth. Afr J Agric Res. 2009;4(6):564-567.
7. Shakeri J, Foster HA. Proteolytic activity and antibiotic production by Trichoderma harzianum in relation to pathogenicity to insects. Enzyme Microb Tech. 2007;40(4):961-968.
8. Martínez B, Reyes Y, Infante D, González E, Baños H, Cruz A. Selección de aislamientos de Trichoderma spp. candidatos a biofungicidas para el control de Rhizoctonia sp. en arroz. Rev Protección Veg. 2008;23(2):118-125.
9. Peteira B, Esteves I, Montes de Oca N, Hidalgo-Díaz L. Estabilidad de la cepa IMI SD 187 de Pochonia chlamydosporia var. catenulata en medio sólido. Rev. Protección Veg. 2007;22(2): 124-127.
10.Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein dye binding. Anal Biochem. 1976;73:248-250.
11.Boller T, Ghri A, Mauch F, Vogeli U. Chitinase in bean leaves: induction by ethylene, purification, properties and possible function. Plant. 1983;157:22-31.
12.InfoStat versión 2009, Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina. 2009.
13.De la Cruz J, Rey M, Lora JM, Hidalgo-Gallego A, Domínguez F, Pintor-Toro JA, et al. Carbon source control on b-glucanases, chitobiase and chitinase from Trichoderma harzianum. Arch Microbiol. 1993;159:316-322.
14.Muiño BL, Botta E, Pérez E, Moreno D, Fernández E. Uso de Trichoderma como alternativa al bromuro de metilo en los cultivos protegidos, flores y ornamentales en Cuba. Fitosanidad. 2006;10(2):179-180.
15.Michel AC, Otero MA, Rebolledo O, Lezama R, Ochoa ME. Producción y efecto antagónico de quitinasas y glucanasas por Trichoderma spp., en la inhibición de Fusarium subglutinans y F. oxysporum in vitro. Revista Chapingo. Serie Horticultura. 2005;11(2):273-278.
16.Šimkoviè M, Kurucová A, Hunová M, Vareèka. Induction of secretion of extracellular proteases from Trichoderma viride. Acta Chim Slov. 2008;1(1):250-264.
17.De la Cruz J, Hidalgo-Gallego A, Lora JM, Benítez T, Pintor-Toro JA. Isolation and characterization of three chitinases from Trichoderma harzianum. Eur J Biochem.1992;206:859-867.
18.Herrera-Estrella A, Chet I. The biological control agent Trichoderma: from fundamentals to applications. In Handbook of Fungal Biotechnology. Arora D. New York: Marcel Dekker. 2003;147_156.
19.Lorito M, Woo SL, Fernandez IG, Collucci G, Harman GE, Pintor-Toro JA, et al. Genes from mycoparasitic fungi as a source for improving plant resistance to fungal pathogens. Proc Natl Acad Sci USA. 1998;95:7860-7865.
(Recibido 24-6-2009; Aceptado 24-12-2009)














