Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión impresa ISSN 1010-2752
Rev. Protección Veg. vol.28 no.3 La Habana sep.-dic. 2013
ARTÍCULO RESEÑA
Avances en el diagnóstico de la marchitez bacteriana (Ralstonia solanacearum): situación actual y perspectivas en Cuba
Advances in the diagnosis of bacteriol wilt (Ralstonia solanacearum): current situation in Cuba and perspectives
Eber Naranjo Feliciano, Yamila Martínez Zubiaur
Centro Nacional de Sanidad Agropecuaria (CENSA). Apartado 10, San José de Las Lajas, CP 32 700, Mayabeque, Cuba. Correo electrónico: yamila@censa.edu.cu.
RESUMEN
La marchitez bacteriana causada por Ralstonia solanacearum (Smith) Yabuuchi, es una devastadora enfermedad que afecta a varios cultivos de importancia económica a nivel mundial. La capacidad de supervivencia del patógeno y la ocurrencia de infecciones latentes hace que el diagnóstico tenga una importante función en la prevención de la enfermedad. El objetivo de este trabajo fue realizar una revisión crítica de las técnicas diseñadas para su diagnóstico y analizar los factores que influyen en el desempeño de las mismas. Los métodos como el cultivo in vitro y las pruebas de patogenicidad son laboriosos, dependen de la interacción planta-patógeno y tardan en arrojar los resultados. Las técnicas basadas en principios serológicos y en la detección de ácidos nucleicos permiten la detección sensible y automatizada del patógeno; sin embargo, la serología depende de la calidad de los anticuerpos y las técnicas moleculares son afectadas por sustancias inhibidoras presentes en las muestras y por la especificidad de los oligonucleótidos. El análisis de la probabilidad post-test en función de la prevalencia, permite decidir con mayor precisión las técnicas apropiadas para el diagnóstico. El uso combinado de técnicas basadas en diferentes principios biológicos aumenta la fiabilidad del diagnóstico, y se recomienda para los procedimientos de cuarentena y certificación de material de importación que se realizan en Cuba. El empleo de una nueva técnica requiere de la comprobación de su robustez mediante ensayos intra e interlaboratorios, antes de ser implementada en los sistemas de diagnóstico.
Palabras clave: detección, técnicas, sensibilidad, especificidad, prevalencia.
ABSTRACT
Bacterial wilt caused by Ralstonia solanacearum (Smith) Yabuuchi, is a devastating plant disease that affects several crops of worldwide economic importance. The pathogen ability for surviving in different environments and causing latent infections becomes an accurate diagnosis as a key point for the disease prevention. The aim of this work was the critical review of these techniques and the analysis of those factors affecting their performance parameters. Traditional methods based on in vitro cultures and pathogenicity tests are laborious, dependent of plant pathogen interactions and slow for obtaining the results. Advanced techniques based on serological principles and nucleic acid detection allow the automated and sensitive pathogen detection, but serology depends on antibody quality and molecular techniques are limited by oligonucleotide specificity and presence of inhibitory substances in the samples. Post-test probability analysis in function of the prevalence provides a higher accuracy in the decision making about the appropriate techniques for diagnosis. Combined use of several techniques based on different biological principles increases diagnosis reliability, and is therefore recommended for quarantine and certification of plant material imported by Cuba. The inclusion of a new technique into a diagnosis scheme requires a previous exhaustive testing of its performance with inter and intra laboratories evaluations.
Key words: detection, techniques, sensibility, specificity, prevalence.
INTRODUCCIÓN
La marchitez bacteriana causada por Ralstonia solanacearum (Smith) Yabuuchi, es una enfermedad que se encuentra distribuida a nivel mundial y afecta a cientos de especies de plantas en 54 familias (1). La bacteria es responsable de considerables pérdidas en muchos cultivos hortícolas y en solanáceas como la papa, donde deja inutilizadas grandes extensiones de tierras contaminadas con el agente (2).
Las afectaciones provocadas por este patógeno no solo radican en el hecho de que destruye los cultivos, también posee una excepcional habilidad para sobrevivir en el agua, el suelo y la rizosfera de plantas no hospedantes (3). Esto, sumado a la frecuente presencia de infecciones latentes, estableció la necesidad de conocer y contar con métodos sensibles y específicos para detectar la bacteria en dicho estado (1).
En la actualidad, existen una serie de pruebas descritas para el diagnóstico y la detección del patógeno (4). Sin embargo, el uso de estos métodos presenta ventajas y limitaciones en dependencia de las características de la técnica, la muestra problema o la prevalencia de la enfermedad; entre otros factores.
La marchitez bacteriana constituye una seria amenaza para la agricultura en Cuba por su extensa distribución en todas las islas del Caribe y el continente americano y la amplitud de hospedantes que posee. Adicionalmente, desde hace algunos años tiene lugar la importación de semillas de papa (Solanum tuberosum L.) procedentes de diferentes países. Esto conlleva un aumento del riesgo de introducción y establecimiento de la enfermedad, debido a que en el país existen las condiciones climáticas propicias para el desarrollo y supervivencia de R. solanacearum (5), resultando significativo que los servicios fitosanitarios conozcan y dispongan de las herramientas que permitan la detección de esta bacteria, con el fin de impedir la entrada de material infectado al país (5).
El objetivo de este trabajo fue realizar una revisión crítica de las principales técnicas disponibles para la detección de este importante patógeno, exponiendo los principales factores que influyen en el desempeño de las mismas y los aspectos a tener en cuenta para la correcta implementación de estos sistemas.
PARTE ESPECIAL
La marchitez bacteriana y su agente causal R. solanacearum
La marchitez bacteriana fue descrita por primera vez en los Estados Unidos en el año 1896 en cultivos de papa, tomate y berenjena (6). En la actualidad se encuentra distribuida en las regiones tropicales y subtropicales de los cinco continentes geográficos (7) y afecta a 54 familias de plantas (1).
Las pérdidas causadas por esta enfermedad son enormes a nivel mundial; no obstante, resulta difícil hacer una estimación exacta debido a la escasa información existente sobre los daños que ella produce en la agricultura de subsistencia y por el abandono de los cultivos altamente susceptibles al patógeno. En países donde R. solanacearum es un patógeno cuarentenado, la enfermedad produce pérdidas económicas directas por la completa destrucción de las cosechas infectadas y de manera colateral, por las restricciones para cultivar en las tierras infectadas y la prohibición del empleo de aguas contaminadas con la bacteria, entre otras medidas (1).
La especie heterogénea Ralstonia solanacearum se debe tratar como un complejo de especies. Tradicionalmente las cepas de este complejo fueron clasificadas de manera informal en 5 razas, de acuerdo a su gama de hospederos y en 6 biovares, relacionados con sus propiedades bioquímicas (8).
Por otra parte, el análisis de las secuencias nucleotídicas de la región intergénica (ITS) entre el ARNr 16S-23S y del gen de la endoglucanasa, permitieron dividir el complejo de cepas de la especie en cuatro grupos denominados filotipos. Estos grupos se corresponden con el origen geográfico primario de cada cepa y a su vez se encuentran subdivididos por variantes de secuencias nucleotídicas a las que se les denomina secuevares (Tabla 1) (8).
Ciclo de vida del patógeno y epifitiología de la enfermedad
Cuando esta bacteria se instala en una región, las posibilidades de diseminación son múltiples (a través de suelo infectado, aguas, labores culturales, restos vegetales, plantas reservorio, etc.) (9). En el suelo, R. solanacearum es capaz de soportar un período de cuatro años con la capacidad de producir marchitez bacteriana en hospedantes susceptibles (10) y sobrevivir el mismo tiempo bajo condiciones de oligotrofia (escasez de nutrientes) y en presencia de bajas temperaturas en microcosmos acuáticos (1).
Está descrito que la bacteria es atraída quimiotácticamente (movimiento estimulado por sustancias químicas) hacia sus hospedantes por diversos aminoácidos, ácidos orgánicos y especialmente por exudados de la raíz (1) y que la concentración de oxígeno dentro de las células del hospedante también cumple importante función en la localización de la planta por parte del patógeno (11). Posteriormente, la penetración a la planta ocurre, generalmente, por la raíz, a través de heridas o en puntos de emergencia de pelos radicales o raíces laterales (se ha comprobado que también puede penetrar por las hojas a través de los estomas) (2, 9).
Una vez en el interior de su hospedante, la bacteria pasa una primera fase en el córtex y posteriormente invade los haces conductores del xilema, donde se multiplica hasta causar el marchitamiento y/o la muerte de la planta. Posteriormente, la bacteria se libera de su hospedante hacia el suelo, el agua o restos vegetales donde puede sobrevivir hasta encontrar una nueva planta susceptible o reservorio (1).
Las infecciones latentes son de gran importancia para la diseminación del patógeno, por lo que también fueron muy bien documentadas. La capacidad de la bacteria de permanecer en material vegetal sin la manifestación de síntomas ha sido la principal vía de diseminación a largas distancias y la causa de brotes acontecidos en la Unión Europea a través de tubérculos de papa contaminados (12) y en plantas ornamentales en Estados Unidos(13). Otros estudios sugierieron que R. solanacearum puede prevalecer en la región del Caribe en forma latente en plantas del género Musa spp. (8, 14). De aquí la importancia de contar con métodos lo suficientemente sensibles para detectar la bacteria en dicho estado y limitar la diseminación de tan devastadora enfermedad.
Métodos de diagnóstico de la marchitez bacteriana
- Diagnóstico presuntivo basado en la sintomatología y pruebas morfológicas
Los síntomas externos más característicos de la enfermedad son marchitamiento, enanismo y amarillamiento del follaje. Éstos pueden aparecer en cualquier etapa del crecimiento del hospedante susceptible, aunque el marchitamiento total y colapso de la planta es más frecuente en plantas jóvenes (6). Los síntomas internos se basan en el cambio de color de los haces vasculares a color pardo o amarillo, en etapas tempranas de la enfermedad. A medida que la enfermedad avanza, la mayor parte de la planta se va marchitando y la coloración de los tejidos conductores va cambiando de marrón oscuro a negro. En estadios avanzados, la degradación de las vesículas ocluidas del xilema, combinada con la extensiva invasión y la destrucción de los tejidos circundantes (floema, córtex y epidermis) producen el colapso y muerte de la planta (6).
La realización de cortes transversales en la parte basal del tubérculo de papa permite observar los cambios en la coloración de la línea vascular y la presencia de exudado bacteriano, que puede fluir de forma espontánea o por la presión ligera con los pulgares. En el tomate, la realización de estos cortes en la base del tallo puede mostrar el oscurecimiento a color pardo de los haces vasculares de los que gotean exudados bacterianos. En este último cultivo, es característica la formación excesiva de raíces adventicias del tallo, especialmente en condiciones de bajas temperaturas (15).
Porciones de la parte basal del tallo de plantas sintomáticas se pueden tomar y colocarse en un recipiente translúcido con agua cristalina. Al cabo de varios minutos se observa la aparición de los exudados bacterianos en forma de hilos lechosos, como indicativo de la multiplicación del patógeno en los haces vasculares de la planta (prueba del flujo bacteriano) (16).
Durante el muestreo también es importante diferenciar los síntomas de pudrición parda en la papa causada por R. solanacearum de los de la pudrición anular causada por Clavibacter michiganensis subsp. sepedonicus. La reacción diferencial de los exudados bacterianos frente al hidróxido de potasio (3%), producto de diferencias en la composición de la pared celular de estos patógenos permite realizar esta discriminación (prueba del KOH) (17).
Otros métodos basados en la observación de los gránulos del polihidroxibutirato (PHB) presentes como material de reserva en las células de R. solanacerum, pueden ser empleados como técnica de diagnóstico presuntivo. Estos procedimientos requieren del uso de colorantes especiales y el empleo de un microscopio óptico de inmersión para su realización (18).
Cabe señalar que ninguno de estos métodos permiten la identificación específica de R. solanacearum, pero son de utilidad a la hora de realizar el muestreo y pueden ser empleados como técnicas auxiliares en estaciones fitosanitarias con pocos recursos.
- Métodos de aislamiento
El aislamiento de R. solanacearum a partir de muestras vegetales sintomáticas generalmente resulta fácil, debido a la alta concentración de la bacteria en los tejidos de la planta. Sin embargo, el cultivo puede fallar en infecciones latentes, en estadios avanzados de la infección o por la competencia o el sobrecrecimiento de bacterias saprófitas (9). Cuando se trata de muestras de suelo y agua, el asilamiento resulta más complejo, pues es mayor la presencia de bacterias antagonistas y otros microorganismos depredadores y se suma la posible entrada de la bacteria en estado viable no cultivable (1).
Para contrarrestar estos inconvenientes se diseñaron varios medios para el aislamiento selectivo del patógeno (18, 19, 20, 21), no obstante, ninguno de ellos fue completamente satisfactorio (9).
El Medio Sudáfrica Modificado (SMSA) fue el más utilizado por su selectividad, con valores de detección entre 10-102 UFC/ml a partir de suspensiones bacterianas puras. No obstante, se debe diferenciar R. solanacearum de otras bacterias que también pueden crecer en este medio (9). Otras desventajas radican en que presenta una mayor complejidad en su elaboración y resulta más costosa su implementación debido al uso de antibióticos específicos.
La sensibilidad del aislamiento selectivo en medio de cultivo sólido puede mejorarse si se realiza un paso previo de enriquecimiento en medio semiselectivo líquido. La aplicación de esta técnica aumenta la concentración del patógeno en la muestra (18, 19, 20) y permite la adsorción o dilución de compuestos inhibidores de técnicas como el ELISA (por sus siglas en inglés Enzime-Linked Immunosorbent Assay) y la Reacción en Cadena de la Polimerasa (PCR) (21, 22).
El inmunoaislamiento o inmunocaptura, es otra de las técnicas que fue introducida en los esquemas de detección para bacterias fitopatógenas. Se basa en la captura selectiva, mediante anticuerpos específicos, de células diana presentes en una muestra que contiene diversos microorganismos (23).
Esta metodología permite concentrar el patógeno de interés en la muestra problema, eliminar la presencia de bacterias antagonistas que puedan aparecer en el aislamiento, así como disminuir el efecto inhibidor así como disminuir el efecto inhibidor de algunos compuestos sobre técnicas como la PCR y el ELISA (19). La técnica es recomendada como una alternativa a la utilización de medios selectivos, debido a su alto porcentaje de recuperación de células diana, a partir de diferentes tipos de muestras y por el corto tiempo que se emplea en el aislamiento. No obstante, sus costos son elevados y no siempre se obtienen valores bajos de detección. Para R. solanacearum, esta prueba mostró valores de detección de 104 UFC/ml a partir de suspensiones bacterianas puras y permitió la detección del patógeno en malezas reservorio (24). Otros autores informaron que el método aumenta los niveles de detección del patógeno en muestras de agua cuando se usa previo a la realización de la PCR (25).
- Métodos biológicos
Las pruebas de patogenicidad resultan de fácil aplicación a R. solanacearum; sin embargo, debido a que dependen de la relación entre el hospedante específico y el patógeno, resulta necesario en primer lugar, determinar la raza y el biovar de cada cepa, para luego escoger el hospedante que se utilizará en el ensayo (9).
Los métodos de inoculación varían en dependencia de la planta, desde la punción o apertura de heridas en yemas axilares y la aplicación del patógeno en pequeños volúmenes, hasta métodos más naturales que requieren que las bacterias sean aplicadas a las raíces de las plantas. En éste último caso, se observó que la herida intencional de las raíces mejora la consistencia de la infección (18), ya que constituye una puerta de entrada adicional para la bacteria y posiblemente potencien el efecto quimiotáctico de los exudados de la raíz (1); no obstante, esta variante presenta la desventaja de que son necesarios grandes volúmenes de inóculo para su implementación (18).
Con óptimas condiciones de temperatura, humedad e iluminación, los valores de detección de los ensayos de patogenicidad varían entre 103-105 UFC/ml y los resultados son obtenidos en 7 días. Cuando la concentración del patógeno es inferior a estos valores, los síntomas pueden no manifestarse o demorarse en su aparición más de 20 días. Sin embargo, también es posible aislar el patógeno en plantas asintomáticas hasta valores de 10-102 UFC/ml (9).
Los métodos biológicos resultan de gran importancia para el diagnóstico, pues permiten confirmar la capacidad del patógeno para producir enfermedad en la planta hospedante. A pesar de esto, estas técnicas son laboriosas y presentan como desventajas su elevado costo, el tiempo requerido para la obtención de los resultados y las limitaciones para su aplicación en gran escala (26).
- Técnicas serológicas
Las pruebas serológicas son de gran utilidad debido a que, en ocasiones, el aislamiento del patógeno a partir del material vegetal puede fallar; además, algunas de estas técnicas permiten la detección del estado latente de la enfermedad, causa de la diseminación de la bacteria a nivel mundial (27).
El empleo de anticuerpos policlonales para la detección serológica de R. solanacearum puede dar lugar a reacciones cruzadas con bacterias cercanas como Ralstonia picketii (Ralston) Yabuuchi y Ralstonia syzygii (Roberts) Vaneechoutte et al. (9), por lo que los resultados positivos deben ser confirmados mediante pruebas secundarias (culturales, patogenicidad, PCR, métodos biológicos). Por otra parte, la mayoría de los anticuerpos monoclonales obtenidos resultan muy selectivos y no reaccionan con todas las cepas de la especie debido a la gran variabilidad que existe entre los diferentes serovares de R. solanacearum, lo que dificulta la obtención de un anticuerpo universal (9).
La Inmunofluorescencia Indirecta (IFI) fue ampliamente señalada para la detección de infecciones de R. solanacearum en tubérculos de papa (4, 27, 28, 29). Los límites de detección de la técnica están entre 103-104 UFC/ml, incluyendo células vivas, muertas y el estado viable no cultivable. Sus principales ventajas radican en que es posible observar la morfología de la bacteria, pudiendo descartar posibles falsos positivos y permitiendo la cuantificación de la población bacteriana (9). Como desventaja de la técnica se ha descrito, la frecuente ocurrencia de reacciones cruzadas producidas por bacterias que comparten antígenos de pared con morfología celular similar a R. solanacearum. Otra limitación radica en la interpretación subjetiva de la morfología de la tinción celular, la cual depende de la experiencia del observador (29).
El ELISA es otra de las técnicas implementadas para la detección de R. solanacearum a partir de diferentes tipos de muestras. Sus limitaciones están determinadas por factores como la calidad de los anticuerpos utilizados, la modalidad de la técnica empleada y la presencia en la muestra de compuestos inhibidores. Con la introducción de anticuerpos monoclonales y la utilización del enriquecimiento selectivo previo a la aplicación de la técnica, se pudo contrarrestar estos inconvenientes (9), con un aumento en la sensibilidad desde 104-105 UFC/ml, a partir de suspensiones bacterianas puras y hasta 1-10 UCF/ml cuando se trata muestras de agua o tejidos vegetales (9, 29).
En el Laboratorio de Bacteriología Vegetal del Centro Nacional de Sanidad Agropecuaria (CENSA) se pusieron a punto las modalidades de ELISA sándwich de doble anticuerpo (ELISA-DAS), ELISA en membrana de nitrocelulosa (ELISA-NCM) y ELISA-DAS empleando el sistema ultramicroanalítico (UM-ELISA-DAS). Con estos métodos se obtuvieron valores de detección entre 103 y 105 UFC/ml y el empleo del enriquecimiento previo también mostró un aumento en la sensibilidad, aunque resultaban necesarias unas 48 horas para obtener los resultados (29).
La técnica de ELISA es de fácil ejecución y automatización en cada una de sus modalidades (9), por esta razón, aunque en algunos países no figura como una técnica de selección principal en los esquemas actuales de detección de cuarentena, continúa siendo utilizada en el diagnóstico de rutina como técnica auxiliar (4).
Otras técnicas serológicas, como la aglutinación en portaobjetos y con partículas látex, también fueron implementadas para el diagnóstico de la enfermedad y los resultados se obtienen en minutos (29); sin embargo, presentan valores de detección por encima de las 106 UFC/ml y es frecuente la presencia de reacciones cruzadas con otros microorganismos, por lo que los resultados deben ser comprobados mediante otras pruebas.
- Técnicas basadas en la detección de ácidos nucleicos
En lo últimos años se produjo un crecimiento exponencial en el diseño de protocolos para la detección de ácidos nucleicos (26). Entre éstos, la técnica de PCR ha sido ampliamente utilizada en los esquemas de detección de esta bacteria a nivel mundial por sus elevados niveles de especificidad, sensibilidad y rapidez (9, 30).
Los límites de detección de la PCR dependen del tipo de protocolo y la naturaleza de la muestra utilizada. Se ha descrito que a partir de extractos de papa, el límite de detección de la técnica oscila entre 101-102 UFC/ml (31). Sin embargo, estos valores pueden afectarse por la presencia de sustancias inhibidoras presentes en las en partículas de suelo, en extractos bacterianos y en la savia de las plantas (32). En este sentido desempeña una importante función la calidad y correcta realización del protocolo de extracción de ácidos nucleicos. Otra limitación de esta técnica radica en la especificidad de los iniciadores. Se plantea que el cebador Y2, uno de los más utilizados, no es lo suficientemente específico, pues hibrida con R. syzygii, Xanthomonas arboricola (Vauterin et al.) pv. celebensis y se ha obtenido señal con bacterias saprofitas presentes en los tubérculos de papa (9).
El enriquecimiento selectivo también fue utilizado previo a la realización de la PCR, para la detección del patógeno en sustratos naturales como material vegetal, suelo y aguas corrientes. La combinación de estas técnicas permite disminuir el límite de detección hasta en dos órdenes en comparación con los resultados obtenidos con el empleo de la PCR convencional a partir de estos sustratos (9, 32). Otros autores utilizaron el innmunoaislamiento combinado con la PCR para la detección del patógeno en plantas reservorios (24) y en muestras de agua (25).
Con la aplicación de protocolos de PCR anidada se logró también un aumento en la sensibilidad y especificidad de la técnica (33). No obstante, esta metodología presenta el inconveniente del elevado riesgo de contaminación, pues se realiza en dos pasos de amplificación en tubos diferentes.
Con un novedoso procedimiento de PCR cooperativa, desarrollado por Caruso et al. (22) se logró eliminar este problema y se ganó en especificidad y sensibilidad, con la combinación de tres cebadores en la detección del patógeno en una única reacción. En este trabajo también se empleó el enriquecimiento previo y se logró un límite de detección de 1 UFC/ml a partir de muestras de agua sin extracción de ADN.
La PCR en tiempo real se ha convertido en una técnica estándar en la detección de bacterias fitopatógenas (30). Existen dos protocolos validados para la detección del patógeno en tubérculos de papa con valores de detección de 30 x 102 (34) y 30 UFC/ml (35), respectivamente. Con el empleo de esta técnica, se eliminan los pasos posteriores de la PCR como la tinción con bromuro de etidio y la electroforesis en gel de agarosa, lo que permite el análisis de un mayor número de muestras, mejor automatización del proceso y disminuye el riesgo de contaminaciones por manipulación. No obstante, los costos del equipamiento y los reactivos para su realización son altos y se ha descrito que puede mostrar reactividad cruzada con bacterias relacionadas como R. syzygii y Pseudomonas sp. (35).
Los métodos isotérmicos de amplificación de ADN también han sido implementados para la detección de R. solanancearum. Kubota et al. (36) diseñaron un protocolo de amplificación isotérmica LAMP (de sus siglas en inglés Loop-Mediated Isotermal Amplification) para la detección de esta bacteria, en el cual los resultados se obtienen en tan sólo 3 horas sin la necesidad de un termociclador y la lectura del resultado se puede realizar mediante electroforesis en gel de agarosa e incluso de forma visual mediante un aumento en la turbidez (36). Sin embargo, presenta como limitaciones que se obtiene como resultado un patrón de bandas tipo marcador de peso molecular y presenta una sensibilidad inferior a la PCR convencional (104-106 UFC/ml). A pesar de estos inconvenientes, esta metodología constituye una alternativa para los laboratorios que no poseen el equipamiento necesario para llevar a cabo una PCR convencional (30).
Otros protocolos fueron diseñados con el objetivo de la caracterización e identificación del patógeno, entre los que se encuentran la PCR múltiple para la detección de los filotipos de R. solanacearum (8) y los procedimientos de Rep-PCR (Regiones repetitivas del genoma) con los cebadores universales ERIC y BOX (37, 38).
La Hibridación Fluorescente in-situ (FISH) es otra de las técnicas que figura entre las pruebas moleculares para la detección de R. solanacearum. La misma se basa en la observación microscópica de la fluorescencia que emite una sonda marcada, cuando hibrida con una secuencia específica del patógeno de interés. Para R. solanacearum se ha utilizado el gen del ARNr 23S altamente conservado en la especie (39).
La FISH muestra un límite de detección inferior a la IFI, pero esta diferencia se debe a que esta prueba sólo detecta células vivas y depende de la viabilidad del patógeno. La FISH tiene como desventajas, el efecto indeseado de la autofluorescencia de los tejidos vegetales (39) y el elevado costo de la técnica. A pesar de estas limitaciones, este método es relativamente rápido (1 día) y permite discriminar posibles falsos positivos obtenidos mediante la IFI (9). Además, su capacidad de detección a partir de sustratos naturales como suelo, agua y muestras vegetales, constituye una alternativa a la aplicación de la PCR y por tanto, resulta de gran utilidad en el estudio de la epifitiología de la enfermedad en los nichos ecológicos de sobrevivencia del patógeno (39).
La capacidad de pequeñas moléculas de ADN para unirse de forma específica a diferentes tipos de ligandos también ha sido evaluada para la detección de células vivas de R. solanacearum (40). La técnica por la que se obtiene estas moléculas es denominada cell-SELEX (de sus siglas del inglés System Evolution of Ligands by Exponential Enrichment) y se basa en la unión y selección repetida de librerías de ADN o ARN a moléculas diana presentes en la célula del patógeno. Este proceso obtiene como resultado un conjunto de pequeñas secuencias de ácidos nucleicos (aptámeros) con una elevada especificidad por la célula diana (41).
Se plantea que esta técnica se muestra como una alternativa al empleo de anticuerpos monoclonales. Este método no necesita del empleo de animales ni de esquemas de inmunización, las pequeñas moléculas de ácido nucleico son capaces de unirse con una elevada afinidad a compuestos muy pequeños no inmunogénicos y tienen una mayor estabilidad ante cambios de salinidad y temperatura. Por otra parte, estos fragmentos son fácilmente acoplables con otros compuestos para su detección (fluoróforos, cromóforos, iones metálicos) y pueden ser obtenidas en grandes cantidades mediante síntesis química (41).
A pesar de todas estas ventajas, el empleo de esta metodología para la detección de R. solanacearum se vio limitada por la elevada variabilidad molecular presente en la superficie de la bacteria y la baja amplificación por PCR de los aptámeros potenciales (40). Otras desventajas del método están en el costo de su diseño, el cual requiere de varios pasos de extracción de ácidos nucleicos, amplificación por PCR y secuenciación de los aptámeros potenciales.
Parámetros de desempeño y validación de las técnicas de diagnóstico
Durante varios años los parámetros de sensibilidad y especificidad fueron empleados para evaluar las técnicas de diagnóstico, donde el desempeño aceptable de una técnica está avalado por valores superiores al 90%.
Las técnicas descritas en este trabajo demuestran adecuados valores de sensibilidad y especificidad (Tabla 2), lo que indica que todas son factibles de implementar para la detección del patógeno. Sin embargo, es importante considerar que tanto la sensibilidad como la especificidad, aún cuando son indicadores de la capacidad operacional de una técnica y dan información de los verdaderos positivos y negativos, tienen limitantes para demostrar el desempeño de una técnica pues ambos parámetros excluye los falsos positivos y falsos negativos en el análisis. Otros parámetros comprendidos en las validaciones son los valores predictivos negativo y positivo; sin embargo, estos fluctúan en función de la prevalencia de la enfermedad (30, 42).
Sin la estimación de la prevalencia de la enfermedad en una población o la probabilidad de infección de una planta, la interpretación de los resultados puede ser comprometedora. Para eliminar estas deficiencias, se han introducido las razones de verosimilitud (RV), las cuales se calculan en base a la especificidad y la sensibilidad y representan ambas, la probabilidad de la presencia del patógeno ante un resultado positivo (con el cálculo de la RV positiva) o negativo (con el cálculo de la RV negativa).
En la Tabla 2 se muestran las RV para cada una de estas técnicas implementadas y en todos los casos la RV positiva está por encima de 10 y la RV negativa está por debajo de 0.1, lo que significa que todas las técnicas tienen la habilidad de expresar si una planta está o no infectada, independiente de la probabilidad de la infección.
A partir de estos parámetros y la aplicación del teorema de Bayes, se pueden calcular los valores de probabilidad de que un resultado positivo o negativo, corresponda efectivamente con el estado sanitario de la planta, a lo que se denomina probabilidad post-test (PPT) (Tabla 3) (42).
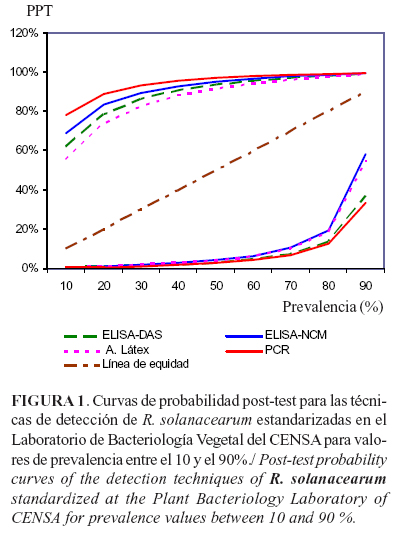
Para un mejor análisis, suelen construirse gráficos de PPT contra la prevalencia (o probabilidad pre-test), donde se pueden comparar simultáneamente el poder discriminatorio de las diferentes pruebas (Fig. 1) (43). El efecto de cada técnica se observa en dos curvas, una para los resultados positivos (curvas por encima de la línea de equidad) y otra para los negativos (curvas por debajo de la línea de equidad). La distancia entre los valores de PPT y la línea de equidad representa el valor de la probabilidad del diagnóstico y se emplea para la toma de decisiones (42).
Como la PPT representa la probabilidad de la presencia del patógeno, la técnica idónea para el diagnóstico a un valor determinado de prevalencia, es aquella que registra un mayor valor de PPT para los resultados positivos y un menor valor de PPT para los resultados negativos. Sin embargo, para decidir qué técnicas emplear, también se debe tener en cuenta los costos de los métodos y la disponibilidad de insumos, con el fin de alcanzar el diagnóstico más fiable al menor costo posible.
Si analizamos los valores de PPT de la Tabla 3, a bajos valores de prevalencia (10%), podemos observar que la técnica de PCR es la más apropiada para el diagnóstico de los positivos; mientras que, para los valores negativos registra valores de PPT idénticos al ELISA-DAS. Como a esta prevalencia de 10%, el número de muestras a procesar es menor, la técnica de PCR puede ser empleada para detectar la mayor cantidad de muestras infectadas en un primer tamizaje, mientras que como segundo método o método confirmatorio puede ser utilizada la técnica de ELISA-DAS.
Por otra parte, a altos valores de prevalencia (90%), las técnicas de PCR, ELISA-DAS y ELISA-NCM muestran valores similares de PPT para los valores positivos, mientras, para los valores negativos, las técnicas de PCR y ELISA-DAS son las que muestran menores valores de PPT. En este caso el número de muestras a procesar es elevado, por tanto es recomendable realizar un primer tamizaje con un método más económico como el ELISA en cualquiera de sus modalidades y posteriormente, realizar la confirmación de las muestras positivas con el método de PCR.
Este tipo de análisis presenta como ventaja adicional que permite evaluar el desempeño de la combinación de varias técnicas, mediante la multiplicación de las razones de verosimilitud de cada método por la prevalencia en el cálculo de la PPT (42). En la Tabla 3 se puede observar que cuando se combinan las técnicas de ELISA-DAS y PCR (ELISA-DAS+PCR*) se produce un aumento en la confiabilidad del diagnóstico, lo cual se puede observar gráficamente en la Figura 2, con curvas mucho más separadas de la línea de equidad que la de los métodos independientes.
Esta marcada diferencia en la fiabilidad del diagnóstico es la causa fundamental, por la cual las normas de los principales protocolos de diagnóstico, estipulan que los resultados deben ser emitidos a partir del uso combinado de técnicas confirmatorias basadas en principios biológicos diferentes (4) y es altamente recomendado su uso para los procesos relacionados con la certificación, importación y cuarentena (42).
En Cuba, R. solanacearum es un patógeno sometido a fuertes medidas de cuarentena y vigilancia fitosanitaria y representa un alto riesgo para la importación de material vegetal al país. Este estudio confirma la factibilidad del uso combinado del ELISA y la PCR para la detección del patógeno, teniendo en cuenta el orden de aplicación en función de la prevalencia de la enfermedad.
Otro aspecto importante en la implementación del diagnóstico radica en la evaluación de la robustez de las técnicas. La inclusión de un nuevo método en los esquemas de detección en un laboratorio, requiere de una validación previa a través de ensayos intra e inter-laboratorios, que garanticen la aptitud de estos requisitos. Estos parámetros pueden ser evaluados con el cálculo del coeficiente de Kappa de Cohen, el cual mide el grado de coincidencia entre las repeticiones de un sistema de clasificación (22).
Es importante señalar que, aunque los técnicas tradicionales como el aislamiento y los ensayos biológicos presentan limitaciones en su implementación, permiten la confirmación del diagnóstico mediante la comprobación de la capacidad del patógeno aislado para producir la enfermedad (postulados de Koch), por lo que la aplicación de estos métodos es decisiva para confirmar un resultado positivo según las directivas establecidas para la detección de R. solanacearum (4).
En concordancia con estas regulaciones, el futuro del diagnóstico parece ir encaminado a la detección de marcadores que evidencien la viabilidad celular y patogenicidad de la bacteria, como el ARNm de determinados genes (22) u otras moléculas presentes en la superficie celular del patógeno (40).
CONCLUSIONES
En la actualidad se encuentran disponibles tanto en Cuba como en el mundo diferentes técnicas para la detección y diagnóstico de R. solanancearum; no obstante, existe un grupo de factores que influyen en los resultados que se obtienen con cada metodología, por lo cual la evaluación de cada una se hace imprescindible antes de su implementación. Es importante conseguir toda la información posible sobre la robustez de las técnicas escogidas para los análisis y sus parámetros de desempeño frente a diversas condiciones. Posteriormente, el empleo de uno u otro ensayo, debe decidirse en función de la información disponible sobre la muestra problema (tipo de muestra, prevalencia y otros), el equipamiento y los recursos disponibles en el laboratorio, el costo de la técnica a realizar y las implicaciones de los resultados del diagnóstico.
REFERENCIAS
1. Álvarez MB. Biology of Ralstonia solanacearum Phylotype II in host and non-host environments. (PhD Thesis), Instituto Valenciano de Investigaciones Agrarias, Valencia, España. 2009.
2. González I, Arias Y, Peteira B. Plant-phytopathogen bacteria interaction: case study Ralstonia solanacearum-host plants. Rev Protección Veg. 2009;24(2):61-80.
3. Carmeille A, Prior P, Kodja H, Chiroleu F, Luisetti P, Besse P. Evaluation of Resistance to Race 3, Biovar 2 of Ralstonia solanacearum in Tomato Germplasm. Jour Phytopathol. 2003;154(3):398-402.
4. Anonymous. Commission Directive 2006/63/EC of 14 July 2006: amending Annexes II to VII to Council Directive 98/57/EC on the control of Ralstonia solanacearum (Smith). Off J Eur Communities. 2006;36-106.
5. Iglesia A, Álvarez E, Martínez Y, García A. Diagnóstico molecular de la Marchitez Bacteriana. Rev Protección Veg. 2008;23(2):75-79.
6. Kelman A. The bacterial wilt caused by Pseudomonas solanacearum. A literatura review and bibliography. U.S.A. North Carolina State College. 1953.
7. Cellier G, Prior P. Deciphering Phenotypic Diversity of Ralstonia solanacearum strains pathogenic to potato. The American Phytopathological Society. 2010;100(11):1250-1261.
8. Fegan M, Prior P. How complex is the R. solanacearum species complex. In: (Eds.) Allen C., Prior P. and Hayward A. The American Phytopathological Society, Bacterial Wilt: The Disease and the R. solanacearum Species Complex. 2005: 449-461.
9. Caruso P. Detección y caracterización serológica y molecular de Ralstonia solanacearum biovar 2, causante de la marchitez y podredumbre parda en patata. Tesis de Doctorado. Instituto Valenciano de Investigaciones Agrarias, IVIA, Valencia, España 2005.
10.Poussier S, Thoquet P, Trigalet-Demery D, Barthet S, Meyer D, Arlat M, et al. Host plant-dependent phenotypic reversion of Ralstonia solanacearum from non-pathogenic to pathogenic forms via alterations in the phcA gene. Mol Microbiol. 2003; 49: 91-1003.
11.Yao J, Allen C. The plant pathogen Ralstonia solanacearum needs aerotaxis for normal Biofilm formation and interactions with its tomato host. Jour Bacteriol. 2007;189: 6415-6424.
12.Hayward AC. The hosts of Pseudomonas solanacearum In: Edited by Hayward, AC & Hartman, GL. Bacterial wilt: the disease and its causative agent, Pseudomonas solanacearum. CAB International, 1994: 9-24.
13.Swanson JK, Yao J, Tans-Kersten JK, Allen C. Behavior of R. solanacearum race 3 biovar 2 during latent and active infection of geranium. Phytopathol. 2005;95(6):136-143.
14.Wicker E, Grassart L, Coranson-Beaudu R, Mian D, Guilbaud C, Fegan M, et al. Ralstonia solanacearum Strains from Martinique (French West Indies) exhibiting a new pathogenic potential. Appl Environ Microbiol. 2007;73(21):6790-6801.
15.Agrios GN. Plant Pathology (5ª ed). Editorial de San Diego, Academic Press, 2005.
16.Champoiseau PG, Jones JB, Momol TM, Pingsheng J, Allen C, Norman DJ, et al. Ralstonia solanacearum Race 3 biovar 2 causing brown rot of potato, bacterial wilt of tomato and southern wilt of geranium, 2010. en línea. Disponible en http://plantpath.ifas.ufl.edu/rsol/NRI_Project/Projectsummary.html (consulta: 28 de mayo de 2012).
17.Muthoni J, Shimelis H, Melis R. Management of Bacterial Wilt Ralstonia solanacearum Yabuuchi et al., 1995 of Potatoes: Opportunity for Host Resistance in Kenya. Journal of Agricultural Science. 2012; 4(9): 64-78.
18.Schaad NW, Jones JB, Chun W. Ralstonia solanacearum. In: Plant Pathogenic Bacteria. 3rd Edition. Editorial APS Press, 2001:151-162.
19.French ER, Gutarra L, Aley P, Elphinstone JG. Culture media for Ralstonia solanacearum isolation, identification and maintenance. Fitopatología. 1995;53(30):126-130.
20.Ito S, Ushijima Y, Fujii T, Tanaka S, Cameya-Iwaky M, Yoshiwara S, et al. Detection of viable cells of Ralstonia solanacearum in soil using a semiselective médium and PCR technique. Jour Phytopathol. 1998;5(146):379-384.
21.Caruso P, Gorris MT, Cambra M, Palomo JL, Collar J, López MM. Enrichment Double-Antibody Sandwich Indirect Enzyme-Linked Immunosorbent Assay that uses a specific monoclonal antibody for sensitive detection of R. solanacearum in asymptomatic potato tubers. Appl Envirol Microbiol. 2002;68(7):3634-3638.
22.Caruso P, Bertolini E, Cambra M, López MM. A new and sensitive Co-operacional polymerase chain reaction (Co-PCR) for a rapid detection of Ralstonia solanacearum in water. J Microbiol Methods. 2003;55:257-272.
23.De León GL. Diagnóstico, epidemiología y control de Clavibacter michiganensis subsp. michiganensis. Tesis de Doctor en Ciencias. Instituto Valenciano de Investigaciones Agrarias, Valencia, España. 2009.
24.Dittapongpitch V, Surat S. Detection of Ralstonia solanacearum in soil and weeds from commercial tomato fields using immunocapture and the polymerase chain reaction. Jour Phytopathol. 2003;151:239-246.
25.Peckham GD, Schell MA, Kim J, Berestecky JM, Alvarez AM. Immuno-capture of Ralstonia solanacearum by an EPS-specific monoclonal antibody enhances sensitivity of PCR. Phytopathology. 2009;99(6):S101-S101.
26.López MM, Llop P, Olmos A, Marco-Noales E, Cambra M, Bertolini E. Are Molecular Tools Solving the Challenges Posed by Detection of Plant Pathogenic Bacteria and Viruses? Curr. Issues Mol Biol. 2009;11:13-46.
27.Van der Wolf JM, Bekkum PJ, Elsas JD. Immunofluorescence colony staining and selective enrichment in liquid medium for studying the population dynamics of R. solanacearum (race 3) in soil. Bull OEPP/EPPO. 2003;28(2):71-79.
28.Stefanova M, García A. El servicio de diagnóstico de las bacterias fitopatógenas en Cuba: desarrollo y alcances. Fitosanidad. 2007;11(3):5-10.
29.Álvarez E, Iglesia A, García A, Blanco E. Optimización de métodos serológicos para la detección de Ralstonia solanacearum (Smith) Yabuuchi. Rev Protección Veg. 2008;23(1):26-31.
30.Palacio-Bielsa A, Cambra MA, López MM. PCR detection and identificationof plant-pathogenic bacteria: Updated Review of Protocols (1989-2007). J Plant Pathol. 2009;91(2):912-957.
31.Pastrik K-H, Maiss E. Detection of Ralstonia solanacearum in Potato Tubers by Polymerase Chain Reaction. Jour Phytopathol. 2000;148(12):619-626.
32.Poussier S, Cheron JJ, Couteau A, Luisetti J. Evaluation of procedures for reliable PCR detection of R. solanacearum in common natural substrates. J Microbiol Methods. 2002;12(2):16-27.
33.Poussier S, Luisetti J. Specific detection of biovars of Ralstonia solanacearum in plant tissues by nested-PCR-RFLP. European Journal of Plant Pathology. 2002;106(10):255-265.
34.Weller S, Elphinstone JG, Smith N, Boonham N, Stead D. Detection of R. solanacearum strains with a quantitative, multiplex, real-time, fluorogenic PCR (TaqMan) assay. Appl Environ Microbiol. 2000;66(1):2853-2858.
35.Ozakman M, Schaad NW. A real-time BIO-PCR assay for detection of Ralstonia solanacearum race 3, biovar 2, in asymptomatic potato tubers. Canadian Journal of Plant Pathol. 2003;25:232-239.
36.Kubota R, Vine BG, Álvarez AM, Jenkins DM. Detection of Ralstonia solanacearum by Loop-Mediated Isothermal Amplification. The American Phytopathological Society. 2008;98(9):1045-1051.
37.Norman DJ, Zapata M, Gabriel DW, Duan YP, Yuen JMF, Mangravita-Novo A, et al. Genetic diversity and host range variation of Ralstonia solanacearum strains entering North America. Phytopathology. 2009; 99:1070-1077.
38.Xue QY, Yin YN, Yang W, Heuer H, Prior P, Guo JH, et al. Genetic diversity of Ralstonia solanacearum strains from China assessed by PCR-based fingerprints to unravel host plant- and site-dependent distribution patterns. FEMS Microbiol Ecol. 2011;75(3):507-519.
39.Wullings BA, Van Beuningen AR, Janse JD, Akkermans ADI. Detection of Ralstonia solanacearum, Which Causes Brown Rot of Potato, by Fluorescent In Situ Hybridization with 23S rRNA-Targeted Probes. Appl Environ Microbiol. 1998; 64(11):4546-4554.
40.Champoiseau PG, Jones JB, Sefah K, Tan W. Selection of molecular aptamers for identification of live cells of Ralstonia solanacearum: A new method in plant pathology. Phytopathology. 2009; 99(6):S20-S20 Suppl.
41.Graham JC, Zarbl H. Use of Cell-SELEX to Generate DNA Aptamers as Molecular Probes of HPV-Associated Cervical Cancer Cells. PLoS ONE 2012; 7(4): e36103. doi:10.1371/journal.pone.0036103.
42.Olmos AC, Bertolini E, Cambra M. Validación de métodos de detección y diagnóstico de patógenos y costes de la especificidad y la sensibilidad. Boletín SEF. España, 2009;(69):7-11.
43.Lamb CR. Statistical briefing: estimating the probability of disease. Veterinary Radiology and Ultrasound. 2007; 48:297-298.
Recibido: 24-11-12.
Aceptado: 26-7-13.