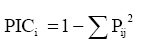Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión impresa ISSN 1010-2752
Rev. Protección Veg. vol.29 no.2 La Habana Mayo.-ago. 2014
ARTÍCULO ORIGINAL
Variabilidad molecular de aislamientos venezolanos de nematodos entomopatógenos y sus bacterias simbiontes1
Molecular varibility of Venezuelan isolates of entomopathogenic nematodes and their symbiont bacteria
Belkis PeteiraI*, Mayra G. RodríguezI, Carolina RosalesII, Anna MaselliII, Raúl CasadoII, Luis CastroII, Efraín SalazarII, Roberto EnriqueI, Ileana MirandaI
IDirección de Sanidad Vegetal, Centro Nacional de Sanidad Agropecuaria (CENSA). Autopista Nacional y Carretera de Tapaste, Apartado 10, San José de Las Lajas, Mayabeque, Cuba. *E-mail de contacto: bpeteira@censa.edu.cu.
IIInstituto Nacional de Investigaciones Agrícolas (INIA), Maracay. Estado de Aragua, Venezuela.
RESUMEN
El objetivo del trabajo fue conocer la variabilidad molecular en una colección de aislamientos venezolanos de nematodos entomopatógenos y sus bacterias simbiontes, a través de la aplicación de los marcadores RAPD. Los ADN de los nematodos y bacterias fueron extraídos siguiendo una modificación del método de Dellaporta descrita por Salazar et al. Los RAPD se desarrollaron con los cebadores de los juegos de reactivos OPA y OPB. Se determinaron las huellas genéticas de los aislados del nematodo y de la bacteria simbionte. El total de bandas amplificadas para nematodos fue de 493 y para bacterias 496, con 99,18 y 100% de polimorfismo, respectivamente. El agrupamiento obtenido para los nematodos estuvo relacionado con la altitud sobre el nivel del mar a la que fueron colectados, mientras que para la bacteria se encontró tendencia al agrupamiento según el tipo de suelo del que provenían.
Palabras clave: nematodos entomopatógenos, RAPD, Variabilidad molecular.
ABSTRACT
The aim of this study was to determine the molecular variability in a collection of Venezuelan isolates of entomopathogenic nematodes and their symbiont bacteria through the application of RAPD markers. The DNA of nematodes and bacteria were extracted by a modification of the method described by Salazar et al. RAPD assay was developed with the primers from the kits OPA and OPB. Genetic fingerprinting of isolates of the nematode and symbiotic bacteria were determined. The total amplified bands for nematode was 493 and 496 for bacteria, with 99,18 and 100% polymorphism, respectively. The clustering obtained for the nematodes was related to the altitude above the sea level at which they were collected, while for the bacteria, the clustering was in general related to the type of soil from which they came.
Key words: entomopathogenic nematodes, RAPD, Molecular variability.
INTRODUCCIÓN
En los últimos 20 años, el desarrollo científico y tecnológico en Cuba y Venezuela propició el hallazgo y desarrollo de diferentes cepas de organismos con potencialidades para constituirse en ingredientes activos de biopreparados/bioplaguicidas para uso agrícola. Diferentes organismos (hongos, bacterias y nematodos) fueron obtenidos en prospecciones y se evaluaron sus potencialidades como agentes de control biológico con resultados satisfactorios (1,2).
Numerosas especies y géneros de nematodos desempeñan funciones de organismos beneficiosos en los ecosistemas, así por ejemplo, representantes de las Familias: Mermithidae, Allantonematidae, Neotylenchidae, Sphaerularidae, Rhabditidae, Steirnenematidae y Heterorhabditidae fueron señaladas con diferentes niveles de impacto, actuando como agentes de control biológico. Las dos últimas familias, reciben en la actualidad la mayor atención en el área de control biológico de plagas insectiles (3, 4, 5). Por su gran potencial referente a este aspecto, se destacan los géneros Steinernema y Heterorhabditis (6).
El proceso de desarrollo de un biopreparado y/o bioplaguicida requiere de diferentes fases que abarcan desde el aislamiento del organismo, su identificación, caracterización, selección y el desarrollo de los bioensayos de eficacia, hasta la ejecución de los ensayos piloto bajo condiciones reales de aplicación (7). En dicho proceso, una de las primeras y más importantes etapas en los programas de biocontrol, es la selección certera de las cepas promisorias y el conocimiento de su variabilidad genética.
Este tipo de información básica e indispensable, eventualmente posee también impacto directo en el éxito del agente de control biológico (3, 5) y en muchas ocasiones, son necesarios métodos de análisis específicos, no solo para el control de la calidad sino también para hacer su trazabilidad, así como los análisis de residuos e impacto ambiental (7). En la actualidad constituye una práctica común la combinación de los análisis de datos morfométricos y el uso de técnicas moleculares (8).
El objetivo del presente trabajo fue conocer la variabilidad genética de nematodos entomopatógenos y sus bacterias simbiontes, aislados en Venezuela, a través de la aplicación de la PCR con cebadores arbitrarios, como base para el trabajo conjunto del Centro Nacional de Sanidad Agropecuaria (CENSA) de Cuba y el Instituto Nacional de Investigaciones Agrícolas (INIA -CENIAP) de Venezuela, en la prospección y desarrollo de nematodos entomopatógenos como una contribución al desarrollo de una agricultura sostenible en la región.
MATERIALES Y MÉTODOS
A partir de trabajos de caracterización previos, realizados con la colección de nematodos entomopatógenos del laboratorio de Nematología de la Unidad de Protección Vegetal del Instituto Nacional de Investigaciones Agrícolas de Venezuela (INIA) (1), se analizaron 12 aislamientos promisorios (Tabla), mediante los marcadores de tipo RAPD para conocer la variabilidad genética de las mismas. El estudio se realizó en el Laboratorio de Biotecnología de INIA Maracay, Estado Aragua y el Centro Nacional de Sanidad Agropecuaria (CENSA) en la Habana, Cuba. Se tomó como cepa de referencia la denominada HC1 procedente de la colección del CENSA, identificada y caracterizada por Sánchez (9).
Los nematodos se produjeron sobre el hospedante alternativo Galleria mellonella (L.), empleando posteriormente el método de trampas White para su colecta en agua destilada estéril. La solución con los nematodos, fue concentrada por centrifugación a 5000rpm durante 5 minutos, en tubos eppendorf. Para el aislamiento de las bacterias simbiontes, a partir de los nematodos se procedió siguiendo el protocolo establecido por Boemare (10). El procedimiento utilizado para la extracción de los ADN de los nematodos y las bacterias simbiontes fue una modificación del método de Dellaporta descrita por Salazar et al. (11), donde se eliminan los pasos de tratamiento con alcohol isoamílico, se hacen dos precipitaciones consecutivas con acetato de potasio 5M y se desarrolla en volúmenes de ensayo reducidos. El ADN obtenido fue resuspendido en 100 ul de buffer TE 1X (Tris-EDTA) y se conservó a -20ºC. La calidad del ADN se constató por electroforesis en geles de agarosa al 0,8% en solución amortiguadora de corrida TBE 0,5X, a 90 volts y teñidos con bromuro de etidio (5mg/ml) y observados en un transiluminador (LKB, Pharmacia). La concentración se estimó por la medición de la densidad óptica a 260nm, según Sambrook et al. (12).
Posteriormente se desarrolló el ensayo de la PCR con cebadores arbitrarios (RAPD). La reacción de amplificación se realizó en un volumen total de 15ul que contenía: 10mM Tris-HCl a pH 8,3; 50mM KCl, 1,5mM MgCl2, 0,001% de BSA, 0,2mM de cada dNTPs, 0,5mM del cebador, 10ng de ADN genómico y 1U de Taq ADN polimerasa. Se ensayaron cada uno de los 20 cebadores de los juegos OPA y OPB (40 cebadores en total de la Firma Comercial Operon Technologies), a una concentración de 0,3mM cada uno. El programa de amplificación fue de 1 ciclo de 5 minutos a 94ºC, seguido de 45 ciclos de: 1 minuto a 93ºC, 30 segundos a 36ºC y 2 minutos a 72ºC, y un ciclo de 7 minutos a 72ºC. Los productos de la PCR se visualizaron por electroforesis en geles de agarosa al 1,5%, en solución amortiguadora TBE 0,5X (45mM Tris-Borato, 1mM EDTA), a 90 volts y se tiñeron con bromuro de etidio, antes de ser observados al UV. Las amplificaciones se repitieron tres veces para cada muestra con cada uno de los cebadores.
Con los perfiles de amplificación se confeccionó una matriz binaria, donde las bandas se informaron como presentes (1) o ausentes (0), para cada aislamiento analizado de nematodo y su bacteria simbionte. Se tuvieron en cuenta las bandas más intensas y con los datos obtenidos se calculó el índice de similitud entre cada par de aislamiento, según Jaccard, los que fueron utilizados para la construcción de un Dendrograma empleando el método de Ward. Estos análisis se realizaron mediante el programa InfoStat versión 2009 (13).
El contenido de información polimórfica (PIC) se calculó según la fórmula:
Donde: Pij es la frecuencia del alelo j para el marcador i (14).
RESULTADOS Y DISCUSIÓN
El método de extracción desarrollado rindió cantidades adecuadas de ADN para los ensayos de la PCR, con concentraciones que oscilaron desde 50 a 150ng/ul, tanto en las muestras procedentes de los nematodos como de las bacterias. La calidad del ADN de la variante escogida quedó demostrada en la nitidez, resolución y repetibilidad de los resultados de las amplificaciones RADP. De igual forma, estos resultados avalan la correcta selección de las condiciones de amplificación, así como las concentraciones de los reactantes más críticos, elementos fundamentales para la adopción de esta técnica para el estudio de diversidad, especialmente cuando se comparan diferentes corridas de PCR y de electroforesis (Figs. 1 y 2).
Cada población analizada presentó patrones de bandas característicos con los cebadores utilizados, lo cual representa su huella genética. En las Figuras 3 y 4 se muestran los resultados de las amplificaciones para los nematodos y sus bacterias simbiontes en dos aislamientos como ejemplo (aislamientos 5 y 6) con todos los cebadores del juego de cebadores OPA. Tanto para nematodos como para bacterias se observó que hay polimorfismo entre aislamientos. Los patrones de amplificación también fueron diferentes cuando se compara el nematodo con su bacteria simbionte para un mismo cebador (Figs. 3 y 4).
La amplificación del ADN de los nematodos con los cebadores del juego OPA produjo un total de 278 bandas con un porcentaje de polimorfismo de 98,58; mientras que con el juego de cebadores OPB (excepto el cebador OPB09 que no se utilizó en el análisis por la baja resolución y calidad de los patrones) se amplificaron un total de 215 bandas, con un 100% de polimorfismo. En el caso de las bacterias, con el juego OPA se amplificaron 309 bandas y 187 bandas con el OPB, obteniéndose en ambos casos un 100% de polimorfismo. Por lo tanto, el total de bandas amplificadas para nematodos fue de 493 y para bacterias 496, con 99,18 y 100% de polimorfismo, respectivamente. Se deduce de estos resultados que los cebadores RAPD seleccionados fueron eficientes para establecer diferencias genéticas entre los aislamientos estudiados.
El conocimiento de la diversidad y distribución de los NEPs es extremadamente valioso no solo para estudios de ecología y biocontrol, sino también para futuros análisis bioprospectivos (15).
El polimorfismo detectado a través de los RAPDs, es el resultado de cambios en la composición de las bases de los sitios de adhesión de los cebadores (16). Los RAPD fueron efectivos para la identificación de poblaciones locales de especies animales y de nematodos entomopatógenos (17). En general, son considerados como herramientas útiles para el diagnóstico de NEPs, así como para establecer sus relaciones filogenéticas y análisis comparativos entre aislamientos promisorios por la expresión de genes involucrados en la patogenicidad en insectos (18, 19). También fueron considerados eficientes para la determinar la variabilidad genética entre aislados y especies de los géneros Heterorhabditis y Steinernema. Melo et al. (20), aseveraron que las técnicas tradicionales como la morfometría, morfología y cruces para identificación, son métodos que necesitan especialistas y que, además de ser complicados, toman mucho tiempo, razón por la cual emplearon las técnicas de PCR para la identificación de poblaciones nativas de nematodos entomopatógenos en cuatro departamentos de Colombia. Sin embargo, no fueron ampliamente utilizados debido a que la reproducibilidad de los resultados pueden variar por muchos factores, tales como la calidad y concentración de ADN, de MgCl2, Taq polimerasa, cebadores y las condiciones de amplificación, incluyendo el equipo utilizado (21, 22).
Es de notar los elevados porcentajes de polimorfismos obtenidos en las amplificaciones con los cebadores analizados. Kumar et al. (23) alcanzaron resultados similares, aunque realizaron sus ensayos con otros iniciadores. Estos autores usaron 13 cebadores para analizar 20 aislamientos de la bacteria simbionte, de los cuales solo 7 cebadores rindieron productos de amplificación adecuados por su nitidez, con un promedio de 6,28 bandas por cebador y un 95,45% de polimorfismo.
Por otra parte, los valores de PIC (Contenido de información polimórfica) para el análisis en las aislamientos de nematodos estuvieron entre 0,49 y 0,92 con un promedio de 0,71, mientras que para el análisis de los aislamientos bacterianas los valores fluctuaron entre 0,55 y 0,98 con un promedio de 0,76. Para ambos organismos los valores de PIC obtenidos se encuentran cercanos a 1, que es el valor máximo de contenido de información polimórfica para marcadores dominantes, como los RAPD. Esto sugiere que se cuenta con un sistema altamente efectivo para lograr el establecimiento de diferencias en el genotipo entre los aislamientos estudiados, por lo que a través de este indicador, también podemos aseverar que la selección de la técnica y de los iniciadores. En la literatura consultada se encontraron referencias a los valores de PIC para marcadores dominantes, pero en específico se hace alusión a los microsatélites. Estos marcadores fueron utilizados exitosamente en la caracterización de la variabilidad genética de diferentes cultivos como yuca, trigo con valores promedio entre 0,50 y 0,70, los cuales fueron calificados por los autores como valores buenos en los análisis realizados (24, 25, 26).
La generación de un elevado número de bandas (493 y 496 para nematodos y bacterias, respectivamente) es otro índice acerca de la capacidad discriminatoria de los cebadores seleccionados, en virtud de que, a medida que se genere un mayor número de bandas, el estudio adquiere una mayor robustez estadística, y por ende una mayor confiabilidad de los resultados. Al analizar el error estándar promedio se obtuvo un valor de 0,01; lo cual indicó que los datos generados fueron estadísticamente confiables y pueden ser considerados como representativos de la variabilidad real presente entre los aislamientos estudiados.
El Dendrograma resultante del análisis de las distancias genéticas para los nematodos mostró la formación de 2 grupos (Fig. 5) al parecer relacionados con la altitud sobre el nivel del mar a la cual fue colectado cada uno de los aislamientos analizados. En el grupo I se localizaron los aislamientos colectados entre los 20-950 msnm. En el grupo II se ubican los colectados a 1105-3000 msnm. En este último grupo se ubicaron los aislamientos provienen de los Estados de Táchira y Mérida, los cuales son próximos geográficamente y un aislamiento de Aragua, de la zona de la Colonia Tovar, que se caracteriza por su altitud y microclima templado diferente al resto del Estado donde está enclavada esta localidad.
Por su parte, el agrupamiento resultante del análisis de las distancias genéticas para las bacterias mostró también la formación de 2 grupos (Fig. 6). En este caso se observa una tendencia a la ubicación según el tipo de suelo. De esta forma, en el Grupo I se ubican, de forma general, los aislamientos que fueron colectados en zonas con suelos franco limosos o arcillosos, mientras que en el Grupo II la mayoría de los aislamientos bacterianos analizados provenías de suelos tipo arenoso. En este último pueden destacarse además dos brazos. Un brazo donde se agrupan aislamientos provenientes de suelos arenosos y alturas menores (20-110msnm) junto a la cepa de referencia (cepa 13) que también proviene de bajas altitudes. En el otro brazo, tres de los cuatro aislamientos provienen de suelos franco arenosos (altitudes entre 950 y 1620 msnm), junto al aislamiento número dos, que aunque proviene de un suelo franco arcillo-limoso, fue colectado a 3000msnm.
Se conoce que algunos factores abióticos son críticos en la aplicación de nematodos entomopatógenos en el suelo como agentes de control biológico. Entre ellos se incluyen la radiación ultravioleta, la humedad del suelo y la temperatura. Los nematodos necesitan una adecuada humedad en el suelo para su sobrevivencia, movimiento y niveles de oxígeno óptimos. Estas características varían de acuerdo al tipo de suelo, donde también debe tenerse en cuenta la temperatura, parámetro que tiene gran efecto sobre la actividad del nematodo (27). También se informó que los nematodos provenientes de elevadas altitudes pierden su variabilidad más rápidamente que aquellos de altitudes medias o bajas y que los que fueron seleccionados de bajas altitudes se adaptan mejor (28). La temperatura también se encuentra relacionada con la altitud y aunque en este estudio no se tuvieron todos los datos para este parámetro, sí se pudiera inferir que la distribución de los aislados en relación con la altitud debe estar mediada por la temperatura, al igual que con el tipo de suelo. Tales aspectos deben ser objeto de en otros estudios futuros conducentes a la selección de las cepas más promisorias para su producción masiva.
La información referente a la caracterización por vías moleculares de los agentes de control biológico es esencial por razones comerciales, cuando es necesaria la protección de una cepa en particular (8). Los resultados de los patrones RAPD permitieron establecer la huella genética de los aislados, aspecto importante para el registro y protección de las cepas que sean seleccionadas por sus potencialidades para el desarrollo de futuros productos a base de nematodos entomopatógenos en Venezuela.
Los patrones RAPD poseen diversos usos en el estudio de los nematodos entomopatógenos. Así por ejemplo, Shapiro et al. (29), los utilizaron con éxito en un análisis de diversidad genética entre poblaciones salvajes y de laboratorio de H. bacteriophora. Estos autores informaron que la heterogeneidad genética de una población aislada recientemente era similar a la de la población HP88, cepa que había sido reproducida en condiciones de laboratorio durante mucho tiempo. Hashmi y Gaugler (30), determinaron la variabilidad genética intra e interespecifica para siete especies de Heterorhabditis a través de los RAPD, utilizando 10 iniciadores previamente definidos como útiles para la cuantificación de la variabilidad genética entre esas especies. En ese caso encontraron que entre individuos de especies conespecíficas el valor de similitud fue de 96,2%; entre distintos aislamientos pertenecientes a tres especies de Heterorhabditis alcanzaron un porcentaje de similitud de 83,8; mientras que, para las diferentes especies el valor fue de 31,3%. Las bandas obtenidas por los RAPDs fueron correlacionadas de forma positiva con la clasificación morfológica previamente descrita, aunque no fueron capaces de separar algunas de las especies estudiadas.
Kaya et al. (31), pudieron diferenciar, a partir de los patrones RAPDs obtenidos con 11 cebadores, las relaciones genéticas entre cepas nativas de Steinernema y a su vez diferenciarlas de cepas utilizadas como referencia en sus estudios.
En nuestro trabajo, el análisis de la variabilidad exhibida por los aislamientos, sugiere la relación de los agrupamientos (Dendrograma) en base a la procedencia de los mismos en cuanto a altitud sobre el nivel del mar para los nematodos, mientras que para las bacterias parece haber una tendencia hacia el agrupamiento según el tipo de suelo. Ambos aspectos deben ser objeto de investigaciones futuras que permitan correlacionar, con alta certeza, las características de los aislamientos y las condiciones edafoclimáticas del sitio donde se colectaron. Estos aspectos se deben tener en cuenta para la regionalización de las cepas que se seleccionen en cuanto a su uso, utilizándolas en ambientes que le son propicios para su acción efectiva como agentes de control biológico. La asimilación de los enfoques moleculares en la sistemática de los nematodos entomopatógenos ha tenido una escalada significativa en los últimos años. Los caracteres moleculares fueron rápidamente adoptados y se convirtieron en herramientas útiles en la identificación de especies y la sistemática de los nematodos. Especialmente, los enfoques moleculares son esenciales cuando se trata de ambigüedades taxonómicas y también para resolver problemas en la identificación de especies morfológicamente similares (32, 33, 34). Varios métodos y marcadores moleculares fueron utilizados no solo con el propósito de diagnóstico de especies crípticas, poblaciones y cepas, sino también para determinar las relaciones evolutivas entre diferentes nematodos (3). Los métodos de diagnóstico fueron desarrollados basados en la reacción en cadena de la polimerasa, incluyendo PCR-RFLP y la amplificación de ADN polimórfico al azar (RAPD) que facilita la identificación por personas no expertas.
REFERENCIAS
1. Rosales LC. Evaluación de nematodos entomopatógenos como biorreguladores de algunas plagas agrícolas. Tesis de doctorado en Zoología Agrícola. Universidad Central de Venezuela. Facultad de Agronomía, Maracay, Venezuela. 2013. 154 pp.
2. García R, Rosales LC, Fernández-Larrea O, Pérez MC, Rodríguez MG. Impacto del programa de transferencia tecnológica de control biológico en el INIA, mediante el convenio Cuba Venezuela. INIA Hoy. 2009;6:217-223.
3. Lacey LA, Frutos R, Kaya HK, Vails P. Insects pathogens as biocontrol agents: do they have a future?. Biological Control. 2001;21:230-248.
4. Grewal PS, Ehlers R, Shapiro-Ilan D. Nematodes as biocontrol agents. CABI Publishing, CAB International, Oxon, U.K., 2005. 505 pp.
5. Stock SP, Hunt DJ. Morphology and systematics of nematodes used in biocontrol. In Grewal PS, Ehlers RU, Shapiro-Ilan D. (eds.). Nematodes As Biocontrol Agents. CAB International, Oxon, U.K. 2005. 1-68.
6. Pamjav H, et al. Noval application of PhastSystem polyacrylamide gel electrophoresis using restriction fragment length polymorphism - internal transcribed spacer patterns of individuals for molecular identification of entomopathogenic nematodes. Electrophoresis. 1999;20:1266-1273.
7. Montesinos E. Development, registration and commercialization of microbial pesticides for plant protection. Int Microbiol. 2003;6:245-252.
8. Ehlers RU. Biocontrol Nematodes. In H.M.T. Hokkanen & Ann E. Hajek (Eds). Environmental Impacts of Microbial Insecticides. Progress in Biological Control. Kluwer Academic Publishers. 2003. London. 177-220p.
9. Sánchez L. Heterorhabditis bacteriophora HC1. Estrategia de desarrollo como agente de control biológico de plagas insectiles. Tesis en opción al Grado de Doctor en Ciencias Agrícolas. Universidad Agraria de La Habana, La Habana. Cuba. (2002), 100 pp.
10.Boemare N. Biology, taxonomy and systematic of Photorhabdus and Xenorhabdus. In: Gaugler R. (Ed.). Entomopathogenic Nematology. CABI Publishing, New York, 2002, pp 35-56.
11.Salazar E, Marin E, Rosales C, Rodríguez MG, Enrique R, Suárez Zoraida. Identificación de 6 poblaciones de Heterorhabditis sp. mediante el uso de marcadores moleculares RAPD. VII Congreso Internacional Biotecnología y Agricultura, mayo del 2007. Villa Clara, Cuba.
12.Sambrook J, Fritsch E, Maniatis T. Molecular Cloning. A laboratory Manual, 2nd Edition. Cold Spring Harbor Laboratory Press 1989.
13.InfoStat version 2009. Grupo InfoStat FCA, Universidad Nacional de Córdoba, Argentina.
14.Suresh Handi, et al. Genetic diversity among maize varieties revealed by phenotypic descriptors and RAPD profiles. The Journal of Agricultural Sciences. 2013;8(2):91-106.
15.Uribe-Lorío L, Mora M, Stock P. First record of entomopathogenic nematodes (Steinernematidae and Heterorhabditidae) in Costa Rica. J of Invertebrate Pathol. 2005;88:226-231.
16.Hashmi G, Glazer I, Gaugler R. Molecular comparisons of entomopathogenic nematodes using randomly amplified polymorphic DNA (RAPD) markers. Fundamen Applied Nematol. 1996;19:399-406.
17.Peteira B, Pérez S, Fraga Y. Modificación de un micrométodo de extracción de ADN para el análisis por RAPD de la variabilidad genética de Alternaria solani. Rev Protección Veg. 1999;14(2):121-124.
18.Liu J, Berry RE. Phylogenetic analysis of the genus Steinernema by morphological characters and randomly amplified polymorphic DNA fragments. Fundamental and Applied Nematology. 1996;19:463-469.
19.You-Jin Hao, Montiel R, Lucena MA, Costa Simoes. Genetic diversity and comparative analysis of gene expression between Heterorhabditis bacteriophora Az29 and Az36 isolates: Uncovering candidate genes involved in insect pathogenicity. Experimental Parasitology. 2012;130(2):116-125.
20.Melo Elsa L, Ortega CA, Susurluk A, Gaig A, Bellotti AC. Poblaciones nativas de nematodos entomopatógenos (Rhabditida) en cuatro departamentos de Colombia. Revista Colombiana de Entomología. 2009;35(1):28-33.
21.Byron JA, Fodor A, Koppenhöfer Heather S, Stackebrandt E, Stock P, Klein MG. Biodiversity and systematics of nematode-bacterium entomopathogens. Biological Control. 2006;37:32-49.
22.Kibugi JM. Molecular differentiation of desiccation and heat tolerant strains of the entomopathogenic nematode Heterorhabditis bacteriophora. Master dissertation submitted in partial fulfilment of the requirements for the degree of Master of Science in Nematology, 2010.
23.Kumar SV, Mulla SR, Suresh CK. Isolation and molecular characterization of symbiotic bacterial isolates associated with entomopathogenic nematodes in agro climatic zone 5 of Karnataka. J of Phytology. 2011;3(11):25-29.
24.Moyib OK, Odunola OA, Dixon AGO. SSR markers reveal genetic variation between improved cassava cultivars and landraces within a collection of Nigerian cassava germplasm. African J of Biotechn. 2007;6(23):2666-2674.
25.Asare PA, Galyuon IKA, Sarfo JK, Tetteh JP. Morphological and molecular based diversity studies of some cassava (Manihot esculenta crantz) germplasm in Ghana. African J of Biotechn. 2011;10(63):3901-3908.
26.Turyagyenda LF, et al. Genetic diversity among farmer-preferred cassava landraces in Uganda. African Crop Science Journal. 2012;20(s1):15-30.
27.Shapiro DI, Gouge DH, Piggott SJ, Patterson Jane F. Application technology and environmental considerations for use of entomopathogenic nematodes in biological control. Biological Control. 2006;38:124-133.
28.Mwaniki SW, Nderitu JH, Olubayo F, Kimenju JW, Nguyen K. Factors influencing the occurrence of entomopathogenic nematodes in the Central Rift Valley Region of Kenya. Afr J Ecol. 2008;46(Suppl. 1):9-84.
29.Shapiro DI, Glazer I, Segal D. Genetic diversity in wild and laboratory populations of Heterorhabditis bacteriophora as determined by RAPD-PCR analysis. Fundamental and Applied Nematology. 1997;20:581-585.
30.Hashmi G, Gaugler R. Genetic Diversity in Insect-Parasitic Nematodes (Rhabditida: Heterorhabditidae). J of Invertebrate Pathol. 1998;72(3):185-189.
31.Kaya HK, et al. Status of entomopathogenic nematodes and their symbiotic bacteria from selected countries or regions of the world. Biological Control. 2006;38:134-155.
32.Cutler CG, Stock Patricia. Steinernema websteri n. sp. (Rhabditida: Steinernematidae), a new entomopathogenic nematode from China. Nematol Medit. 2003;31:215-224.
33.Stock P, Griffin CT, Chaenari R. Morphological and molecular characterization of Steinernema hermaphroditum n. sp. (Nematoda: Steinernematidae), an entomopathogenic nematode from Indonesia, and its phylogenetic relationship with other closely related taxa. Nematology. 2004;6:401-413.
34.Stock P. New trends in entomopathogenic nematode systematics: Impact of molecular biology and phylogenetic reconstruction. In: Proc. International Congress of Parasitology. (Ed.) Monduzzi. Canada, 2002;1-8 pp.
Recibido: 21-5-2014.
Aceptado: 21-7-2014.
1 Resultado obtenido en el marco del Proyecto de Investigación: Producción de nematodos entomopatógenos para el manejo de plagas agrícolas. Convenio Integral de Cooperación Cuba-Venezuela. 2007-2009.