Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Protección Vegetal
versión impresa ISSN 1010-2752versión On-line ISSN 2224-4697
Rev. Protección Veg. vol.30 supl.1 La Habana dic. 2015
ARTÍCULO ORIGINAL
Validación de indicadores de calidad para la cría masiva de Tamarixia radiata Waterston (Hymenoptera: Eulophidae) en Cuba
Validation of quality indicators for the mass rearing of Tamarixia radiata Waterston (Hymenoptera: Eulophidae) in Cuba
Heyker L. Baños DíazI, María de los Ángeles Martínez RiveroI, Ileana Miranda CabreraI, Margarita Ceballos VázquezII
IDireccción de Sanidad Vegetal. Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, Mayabeque, Cuba. Correo electrónico: hlellani@censa.edu.cu.
IIDepartamento Gestión de Calidad. Centro Nacional de Sanidad Agropecuaria (CENSA), Apartado 10, San José de las Lajas, Mayabeque, Cuba. Correo electrónico: margara@censa.edu.cu.
RESUMEN
El objetivo del presente trabajo fue evaluar los indicadores de calidad de la cría de Tamarixia radiata Waterston en condiciones de laboratorio. A partir de los indicadores de calidad ya establecidos, se procedió a evaluar el comportamiento de los parámetros para el control de la calidad de Tamarixia radiata sobre Diaphorina citri Kuwayama. Se analizaron la talla y la longitud de la tibia media, el porcentaje de emergencia, longevidad del adulto con y sin alimento, las deformaciones, el porcentaje de parasitoidismo y la relación de sexos en el 10% de las poblaciones obtenidas en los lotes durante el proceso de cría. Con la información obtenida se construyeron los gráficos de Shewart. En todas las generaciones evaluadas la relación de sexos fue favorable a las hembras, con una media de 0,90±0,39. El análisis de los sexos evidenció que en todos los casos la hembra alcanzó un mayor tamaño que los machos. No se observaron deformaciones en las alas de los individuos. La sobrevivencia promedio para las hembras fue de 2,47 días y de 1,98 días para los machos. La media de parasitoidismo fue de 62,58% y la emergencia osciló entre 74,56 y 80,2%. Los indicadores estuvieron dentro de los valores de aceptación, por lo que se demostró que el proceso de producción estaba bajo control. El promedio de adultos de T. radiata en los 10 lotes evaluados fue 340, valor que se encuentra dentro del esperado; lo anterior evidenció la eficiencia en el proceso de cría bajo las condiciones impuestas.
Palabras clave: control de calidad, Tamarixia radiata, gráficos de Shewart.
ABSTRACT
The aim of this study was to evaluate the quality indicators for the mass rearing of Tamarixia radiate Waterston under laboratory conditions. From established quality indicators, the behaviour of the routine parameters for the quality control of Tamarixia radiate on Diaphorina citri Kuwayama was evaluated. Size and length of the middle tibia, emergency percentage, longevity of adults with and without food, deformations, parasitism percentage, and sex ratio were analyzed in 10% of the populations obtained per batch during the rearing process Shewart graphics were constructed with the information obtained. In all generations evaluated, the sex ratio was favorable to females, with a mean of 0.90 ± 0.39. In all cases, the gender analysis showed the female to reach a larger size than the males. No deformations were observed on the wings of the specimens. The mean survival was 2.47 days for females and 1.98 days for males. The parasitism average was 62.58% and the emergency ranged between 74.56 and 80.2%. The indicators were within acceptance values, demonstrating that the production process was under control. The adult average of T. radiata in the ten batches tested was of 340 parasitoids, a value which is within the expected ones, which showed the efficiency of the rearing process under the conditions imposed.
Key words: quality indicators, Tamarixia radiata, Shewart graphics.
INTRODUCCIÓN
Mantener a un grupo de insectos por varias generaciones en un insectario, donde se proporcionan factores abióticos estables (temperatura, luz, humedad, viento) y factores bióticos constantes (alimento); además de la ausencia de competencia por hospedante, por pareja sexual, por sitios de apareamiento y sin necesidad de desplazamiento para localizar los recursos, son factores que pueden provocar que los insectos reproducidos en el laboratorio pierdan algunas de las características deseables que presentaban los especímenes que dieron origen a la colonia (1).
No fue hasta la década de los 80 que se comenzó el uso del control de la calidad en las crías de insectos y, paulatinamente, se convirtió en un tema más popular (2,3). Durante el periodo comprendido entre los años 1991 y 1998, la Organización Internacional de Control Biológico (IOBC) determinó los parámetros para el control de la calidad de 20 especies de enemigos naturales (4). Estos parámetros comprenden, por lo general, características relativamente fáciles de determinar en el laboratorio (emergencia, relación sexual, longevidad, fecundidad, tamaño del adulto y tasas de predación o parasitoidismo) (5, 6). Posteriormente, se iniciaron los estudios que permitieron desarrollar los criterios para evaluar la capacidad de vuelo y relacionar esa característica, determinada en el laboratorio, con la eficiencia en campo (7).
De acuerdo con van Lenteren (8), existen algunas recomendaciones básicas que se deben considerar para tener una cría de insectos que conserven características deseables y se disminuya el impacto de la domesticación, como son el establecimiento de estándares de calidad y determinar las pruebas que proporcionen los indicadores a evaluar durante el proceso de cría. Este mismo autor señaló, como criterios generales para el control de calidad para parasitoides, el número de parasitoides vivos (si se envían como adultos), el número de adultos que emergen en cierto tiempo (si son estados inmaduros), la relación sexual, la fecundidad, el grado de mortalidad del hospedante, la longevidad, el tamaño de los adultos, la capacidad de vuelo y el desempeño en campo.
Es importante resaltar que los ensayos de fecundidad, longevidad y capacidad de parasitoidismo se pueden combinar entre ellos. Por otra parte, el control de calidad debe ser realizado bajo condiciones específicas de temperatura, humedad relativa y fotoperiodo para cada organismo (8). Es muy probable que algunos de los efectos de la domesticación sean inevitables, pero tomar ciertas precauciones puede minimizar su impacto (1).
El control de calidad en la cría de cualquier insecto debe involucrar a todos los aspectos que puedan influir en su desempeño final. Estos aspectos deben tener revisiones periódicas y recomendaciones para mejorar el sistema de cría (9).
El parasitoideTamarixia radiata Waterston se empleó con éxito en numerosos programas de control de Diaphorina citri Kuwayama, vector de la bacteria Candidatus liberibacter spp., agente causal de Huanglongbing (HLB), y se utiliza actualmente a escala mundial para la regulación de las poblaciones del fitófago; además, aunque no se comercializa aún, se produce en varios países: Taiwan (10), Brasil (11), Argentina (12), México (13) y Venezuela (14).
En Cuba se determinó la efectividad de este parasitoide frente al vector y se estableció un sistema de cría sobre un sustrato alternativo bajo condiciones de laboratorio (15). Sin embargo, no se evaluaron los parámetros para el control de la calidad sobre este hospedante y el sustrato alternativo, aspectos que deben ser estudiados pues permitirán la introducción de este control biológico dentro de la estrategia de manejo del HLB en Cuba, descrita por Batista (16). Por lo que el objetivo del presente trabajo fue evaluar los indicadores de calidad de la cría de Tamarixia radiata en condiciones de laboratorio.
MATERIALES Y MÉTODOS
El estudio se realizó a partir de crías masivas del parasitoide T. radiata establecidas en jaulas de 60x50x50cm, colocadas en un cuarto climatizado del laboratorio de Entomología-Acarología del Centro Nacional de Sanidad Agropecuaria (CENSA), Cuba, a temperatura de 28 ±1oC y 60±10%, humedad relativa y un régimen de luz de 14 horas. Como hospedante se utilizó D. citri y, como sustrato, muralla (Murraya paniculata L. (Jack)).
En cinco jaulas con plantas de muralla sembradas en macetas de cinco litros, previamente infestadas con ninfas de D. citri, se liberaron 30 parejas del parasitoide por 72 horas. Transcurrido ese tiempo y con ayuda de un exahuster, se transfirieron a una nueva jaula para repetir la operación, para lo cual se mantuvo el mismo número de jaulas y parasitoides en cada una de las generaciones.
Las plantas con ninfas, supuestamente parasitadas, se colocaron en jaulas que se rotularon con la fecha y el número de parentales, el número de la descendencia de cada lote y los datos recogidos en un registro. A partir de que los parasitoides comenzaron a emerger, se colectaron y contabilizaron para determinar el rendimiento por lote.
Indicadores de calidad: Según los criterios de diversos autores (5, 6, 8,17,18) y la función que desempeña el parasitoide, se seleccionaron como indicadores morfológicos la talla y la longitud de la tibia media y, como indicadores fisiológicos, el porcentaje de emergencia, la longevidad del adulto (con y sin alimento), las deformaciones, el porcentaje de parasitoidismo y la relación de sexos en T. radiata. Como lote, se definió cada una de las jaulas con individuos nacidos un mismo día y se evaluaron los indicadores antes seleccionados en el 10% del lote; se tuvieron en cuenta, además, las características biológicas de la especie en cada lote.
Para evaluar los indicadores antes mencionados se siguieron los procedimientos que se refieren a continuación:
Cociente sexual: Se contabilizó la cantidad de hembras y machos de la descendencia y se calculó la proporción de sexos, a través de la fórmula: hembras/hembra +macho.
Talla de los adultos: Para comparar la talla (largo y ancho) de ambos grupos, se midieron el largo y el ancho de 20 hembras e igual número de machos provenientes de poblaciones de laboratorio. La longitud (mm) se midió desde el extremo anterior de la cabeza hasta el extremo posterior del abdomen y el ancho por la parte dorsal más ancha del insecto. Ambas medidas se tomaron con un microscopio estereoscopio ZeissStemi DV 4 con ocular micrométrico a 8 aumentos.
Los resultados obtenidos se compararon con preparaciones fijas de adultos procedentes de campo que se conservan en la colección de los insectos del laboratorio de Entomología-Acarología del Centro Nacional de Sanidad Agropecuaria (CENSA).
Deformaciones en los adultos:
Según diseño completamente al azar, se contó con 10 parasitoides adultos por cada generación evaluada a los que se les observó si presentaban deformaciones en las alas.
Sobrevivencia: Para determinar la sobrevivencia se tomaron en los lotes 20 insectos (10 hembras y 10 machos), los cuales se sexaron e individualizaron en viales tapados con mallas antiáfidos. En la parte superior de los viales se depositó una mota de algodón embebida en agua y, en los casos que se evaluó la alimentación, se suministró miel y polen de flores de majagua (Hibiscus elatus Sw.). El experimento se replicó tres veces para un total de 60 muestras en cada caso. Se registraron los momentos de la emergencia del adulto y de su muerte; además, se calculó la sobrevivencia.
Para los análisis estadísticos se empleó el paquete InfoStat versión 2010. Las diferencias entre los indicadores de calidad se obtuvieron mediante un análisis de varianza simple y las medias se compararon según la prueba de rangos múltiples de Tukey para p£0,01.
Con el objetivo de detectar los cambios ocurridos durante el proceso de producción se elaboraron los Gráficos de Shewart, según las normas ISO 2002 (19), para el coeficiente sexual, el por ciento de parasitoidismo y el rendimiento. De esta forma se garantiza mantener la efectividad del parasitoide en el control de la plaga, después que se reproduzca por un número de generaciones en el laboratorio.
RESULTADOS Y DISCUSIÓN
Es importante resaltar que los valores de los indicadores de calidad obtenidos tienen gran importancia práctica, pues permiten mejorar y ajustar las condiciones y las características del sistema de cría que se implementó, al mismo tiempo que permiten conocer el estado de la producción del parasitoide.
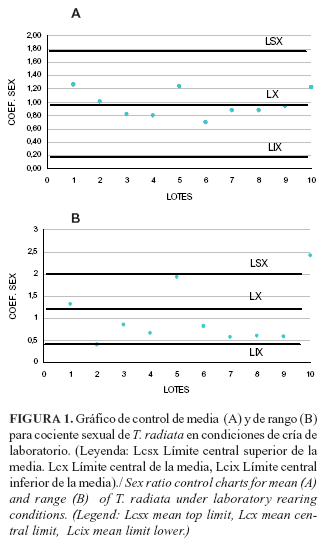
Cociente sexual: En todas las generaciones evaluadas la relación de sexos fue favorable a las hembras, con una media de 0,90±0,39, sin diferencias significativas entre ellas. Esto sugiere un comportamiento homogéneo del parámetro biológico entre los lotes muestreados.
Por otra parte, el cociente sexual promedio obtenido en este estudio fue superior al establecido dentro de los estándares para la cría (15), condición que puede resultar ventajosa, pues determina un mayor número de huevos y, por consiguiente, mayor tasa de parasitoidismo.
Según Ceballos (17), estas oscilaciones se consideran favorables para las crías masivas de artrópodos que se reproducen sexualmente, donde es muy deseable un aumento de las hembras, con vistas a incrementar la obtención de descendientes, lo que contribuye a mantener alto el nivel de población en las unidades de cría y garantizará las posteriores liberaciones en campo para los programas de control biológico.
Este resultado se corresponde con lo informado por Baños et al. (20), ya que obtuvieron 2 hembras por cada macho. De igual forma, el hecho de encontrar una tasa favorable a las hembras indica que las condiciones de cría fueron adecuadas (7,17).
Kairo (21) planteó que una desviación rápida del cociente sexual hacia cualquiera de los dos sexos puede indicar un problema en la talla de los hospedantes y, por tanto, una dificultad con el material vegetal utilizado, lo cual se debe revisar.
Aunque una inclinación favorable del coeficiente sexual hacia las hembras es deseable en las crías, se debe tener especial cuidado, pues un número excesivo de estas puede reducir la cantidad de progenitores machos efectivos. En este contexto, se puede decir que la proporción de hembras y machos influye en la capacidad de reproducción de una cría, porque un número muy bajo de machos, o su esterilidad, resultará peligroso para la especie.
No se encontraron diferencias significativas entre los lotes; sin embargo, los gráficos de control (Fig. 1a) mostraron que los límites de aceptación o rechazo de las medidas de tendencia central y los recorridos oscilaron entre 0,15-1,84 y 0,50-1,92, respectivamente, con un valor para el límite central de la media de 0,90.
Las oscilaciones de los puntos de la media mostraron regularidad, mientras que el de los rangos mostró mayor dispersión, aunque el lote 10 estuvo por encima del límite superior (Fig. 1b). En realidad sería interesante seleccionar lotes que tuvieran una mayor cantidad de hembras, pues si tenemos en cuenta que es la hembra de T. radiata la que cumple con la función de parasitar cuando deposita sus huevos en el hospedante, una proporción de sexos a favor de las hembras en la cría garantizará una mejor regulación al ser liberados.
Talla de los adultos: El análisis de los sexos evidenció que en todos los casos la hembra alcanzó un mayor tamaño que los machos. La longitud, el ancho y la longitud de la tibia posterior de las hembras adultas de poblaciones de campo y de laboratorio no mostraron diferencias significativas. Sin embargo, en los machos se encontraron diferencias entre el ancho de los insectos de ambas poblaciones, con valores ligeramente superiores en las poblaciones de laboratorio (Tabla 1).
El hecho de que no existan diferencias significativas entre la talla de las hembras de laboratorio y la calculada para la población inicial de campo confirma que la población mantuvo de forma óptima este indicador en los lotes evaluados. Estos resultados demostraron que no es necesario, hasta la generación evaluada, realizar cruzamientos como una de las medidas que se recomienda para incrementar la calidad de diversas especies que se mantienen bajo condiciones de cría artificial durante determinado tiempo (8).
Al respecto, Kairo (21) afirmó que una desviación en el tamaño del adulto sugiere que se debe revisar el tamaño del hospedante suministrado en las crías o quizás un cambio en el régimen de temperatura utilizado. Este indicador resulta de un valor inestimable, pues según van Lenteren (8), la talla es, frecuentemente, un buen indicador para la longevidad, la fecundidad y la capacidad de parasitoidismo/predación.
Deformaciones de los adultos: No se observaron deformaciones en las alas en los individuos evaluados, lo que constituye un signo positivo del estado de salud genético de las poblaciones en desarrollo. No obstante, cuando aparezcan deformaciones se evaluará hasta qué por ciento puede ser admitido, aspecto que luego será objeto de estudio.
Sobrevivencia: La sobrevivencia promedio para las hembras, procedentes de la cría en el laboratorio con alimento, osciló entre 1-6 días y 1-3 días para los machos, con una media de 2,47 y 1,98, respectivamente. Por otra parte, el comportamiento de este indicador sin suministro de alimento osciló entre 1-3 días para las hembras y 1-2 días para los machos; las medias resultaron de 1,34 y 1,26, respectivamente.
Estos indicadores fueron mayores que los estándares obtenidos a partir de los insectos de campo en la variante con alimento (15). El resultado es ventajoso, pues durante el envío y el traslado de los insectos para la liberación en lugares alejados de los centros de producción, estos benéficos se pueden mantener con la dieta por espacio de 2 días; a su vez, se evidenció que estos insectos son susceptibles al estrés causado por la ausencia de alguna fuente de alimento, por tanto, su traslado hacia los puntos de liberación debe ser rápido.
El aspecto más importante de esta prueba está dado porque una sobrevivencia mayor en los individuos, sobre todo en las hembras, indica que estos tendrán mayor posibilidad de éxito a las adversidades del ambiente (22).
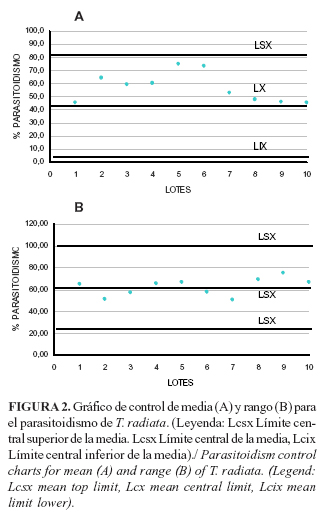
Parasitoidismo: Este indicador osciló entre 32,17% y 95,09%, con una media de 62,58% de parasitoidismo. Los gráficos de control mostraron oscilaciones erráticas por debajo del límite de la media, lo cual es típico de los procesos que no se han sometido, con anterioridad, a procesos de control (Fig. 2). El límite central de la media fue de 62,58 y los límites de aceptación y rechazo de las medidas de tendencia central y los recorridos estuvieron entre 58,4 y 97,85 y 40,8 y 89,65 en cada caso.
No obstante, los valores que estuvieron fuera de los límites se encontraban por debajo del límite central de la media, por lo que en la práctica no se rechazarían los lotes, debido a que el límite de rechazo de la media es superior. Sería de interés seleccionar lotes que tuvieran un porcentaje de parasitoidismo cada vez más alto en correspondencia con los alcanzados en condiciones naturales.
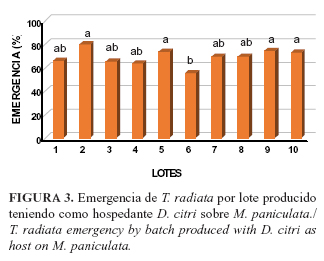
Emergencia: El mayor número de parasitoides emergidos se obtuvo en la segunda generación, seguida por la quinta, novena y décima (Fig. 3); de forma general, la emergencia resultó alentadora, pues osciló entre 74,56 y 80,2%, valores superiores a los alcanzados en condiciones naturales (23).
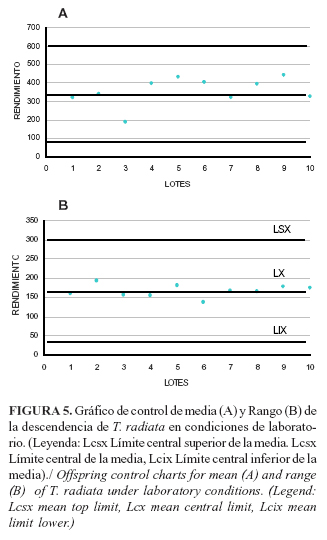
Rendimiento: Este indicador, que está relacionado con el número de entomófagos /contenedor, osciló entre 184 y 749 individuos, y el mayor número de parasitoides se obtuvo en las generaciones 6 y 15; sin embargo, el rendimiento disminuyó por debajo de los 300 individuos en las generaciones 3, 13 y 20, debido a la baja población de ninfas. Este fenómeno estuvo relacionado con la disminución en el número de brotes en el sustrato vegetal, lo que es indicativo de la necesidad de efectuar un control de proceso durante la producción. No obstante, de forma general, los rendimientos alcanzados en el presente estudio están dentro de los deseados, teniendo en cuenta la fecundidad de la especie y a pesar de las disminuciones en las generaciones 3, 13 y 20. (Fig. 4).
Al analizar los valores promedios de la descendencia por generación (Tabla 2), se evidenciaron diferencias significativas. En las generaciones 14, 16, 6, 4, 15 y 5 se alcanzaron los valores más altos, en relación con las restantes, en las 13 y 3 se observaron los valores más bajos; no obstante, la descendencia promedio, desde la primera hasta la última, estuvieron dentro de los valores esperados, al igual que el rendimiento total.
El gráfico de control de medias y rangos (Figura 5) muestra que los valores de rendimiento de los lotes 1 y 3 están por debajo de la línea central (LC) con una tendencia hacia el límite inferior en el caso del lote 3, comportamiento esperado en el inicio de cualquier proceso productivo, donde es necesario la identificación y las corrección de todas las causas que provocan una desviación anormal (17), por ello esta etapa se considera «fase de ajuste». Los límites de rechazo, para las medidas de tendencia central, estuvieron entre 327 y 152 y, para el caso de la dispersión, entre 535 y 151, con un límite central para la media de 340 y de 334 para los recorridos.
Hasta la generación evaluada no se evidenciaron cambios.No obstante, se deben seguir evaluando más allá de la generación 20, pues no se puede aseverar que no se producirán variaciones en los parámetros de control de la calidad. Existe la posibilidad de que el entrecruzamiento, la consanguinidad y la deriva genética (el azar de los cruzamientos puede conceder ventajas a ciertos genes y eliminar otros), causen pérdida de la calidad biológica del enemigo natural producido, lo cual conllevaría restablecer los estándares en los indicadores de calidad a través de poblaciones salvajes.
El control de la calidad, para más de 30 especies de enemigos naturales, incluyeron aspectos básicos que se pueden determinar fácilmente en condiciones de laboratorio, entre ellos están el cociente sexual, el ciclo de vida, la fecundidad, y la tasa de predación/parasitoidismo (24).
Para el fitófago y el parasitoide, los indicadores de calidad se tendrán en cuenta para cada uno de los lotes producidos, se estimará el 10% de las poblaciones obtenidas como tamaño de muestra. Una vez que se completen los 20 lotes, se emplearán los valores para el ajuste de los indicadores iniciales de cría. A partir de ese momento la frecuencia de mediciones será en lotes alternos, valores que se emplearán para la construcción de gráficos de calidad.
Desde el punto de vista estructural, los elementos, que se incluyen en el desarrollo de la cría del parasitoide están en correspondencia, de forma general, con lo descrito por Chien et al. (10), Etienne et al. (25) y Gómez (11), además de los criterios aportados por van Lenteren (8), autor que hace una compilación de la experiencia acumulada a través de los años de la crianza y control de la calidad de agentes de control biológico.
REFERENCIAS
1. Martínez ML. El control de calidad en la cría de insectos. In: Bautista MN, Vejar CG y Carrillo SJ. (eds.), Técnicas para la Cría de Insectos. Colegio de Postgraduados, Montecillo, Edo. de Méx. 1994; 25-37.
2. van Lenteren JC. Integrated pest management in protected crops. In: Dent D. (Ed.). Integrated pest management. London: Chapman and Hall. 1995; 311-343.
3. Leppla NC. Building partnership to support research on commercial natural enemies. Global IOBC Bulletin, 2007;3:82-85.
4. van Lenteren JC. Quality control guidelines for 21 natural enemies. Sting, Newsletter on Biological Control in Greenhouses, Wageningen. 1994;3:24.
5. van Lenteren JC. Designing and implementing quality control of beneficial insects: towards more reliable biological pest control. In: Quality control meeting. Paris: Antibes, 1996; 64.
6. van Lenteren JC, Hale A, Klapwijk JN, Schelt Tvan, Steinberg S. Guidelines for quality control of commercially produced natural enemies. In: van Lenteren JC (Ed.). Quality control and production of biological control agents: theory and testing procedures. Wasllingford: CABI, 2003; 265-303.
7. Van Lenteren JC. Criterios de Seleção de Inimigos Naturais. En: Bueno VEP. (Eds). Controle Biologico de Pragas: Produção Massal e Controle de Qualidade. Editora UFLA. 2009; 11-32.
8. Van Lenteren JC. Controle de Cualidade de Agentes de Controle Biologico produzidos massalmente. En: Bueno VEP. (Eds). Controle Biologico de Pragas: Produção Massal e Controle de Qualidade. Editora UFLA. 2009; 311-337.
9. Boller EF, Chambers DL. Quality aspects of mass-reared insects. In Ridgway RL, Vinson SB. (Eds). Biological control by augmentation of natural enemies. 1977; 219-236.
10.Chien CC, Chu YI, Ku HC. Parasitic Strategy, Morphology and Life History of Tamarixia radiata (Hymenoptera: Eulophidae). Chinese Journal of Entomology. 1991;11:264-281.
11.Gómez TML. Estudos bioecológicos de Tamarixia radiata (Waterston, 1922) (Hymenoptera: Eulophidae) para o controle de Diaphorina citri Kuwayama, 1907 (Hemiptera: Psyllidae). Tesis en Opción al grado científico de Doctor en Ciencias. Escuela superior de Agricultura Luis de Queiroz (ESLAQ). Piracicaba. Brasil. 2009.
12.SENASA. Diaphorina citri Psílido Asiático de los Citrus. 2006. (En línea). Disponible en http://www.sinavimo.gov.ar. Consulta 3-9-2007.
13.SENASICA. Psílido asiático de los cítricos, Diaphorina citri Kuwayama (Hemiptera: Psyllidae). Programa PAC. 2010. Disponible en: http://www.senasica.gob.mx/?id=1013). Consulta: 3-9-2013.
14.Morales VP, Fonseca O, Noguera Y, Cabaña W, Ramos F, Escalona E, et al. Evaluación del ciclo de vida del psílido asiático de los cítricos en cinco plantas hospederas. Agronomía Trop. 2010;60(3):283-286.
15.Baños HL. Tamarixia radiata Waterson (Hymenoptera: Eulophidae): agente de control biológico para la regulación de Diaphorina citri Kuwayama (Hemiptera: Liviidae). [Tesis presentada en opción al grado científico de Doctor en Ciencias Agrícolas]. Centro Nacional de Sanidad Agropecuaria. Universidad Central de Las Villas Martha Abreu. Cuba. 2013. 100pp.
16.Batista L. Epidemiologia y Manejo de Huanglongbing en Cuba, Taller internacional de la RIAC Habana 2011.
17.Ceballos M. Encírtidos asociados a pseudocóccidos, elección y método de reproducción de un agente de control biológico. [Tesis presentada en opción al grado científico de Doctor en Ciencias Agrícolas]. Centro Nacional de Sanidad Agropecuaria. Universidad Central de Las Villas Martha Abreu. Cuba. 2006. 100pp.
18.van Driesche R, Hoddle M, Center T. Midiendo El Impacto De Los Enemigos Naturales sobre Las Plagas. Capítulo 20: Evaluación de los enemigos naturales. En: Control de Plagas y Malezas por enemigos naturales. USA, USDA, 2007; 358-376.
19.Norma Cubana. ISO 8258: 2002. Gráficos de control de Shewhart (Publicada por la ISO, 1991) ICS: 03.120.30 1. Edición Mayo 2002. Oficina Nacional de Normalización (NC).
20.Baños HL, Miranda I, Martínez MA. Biology and life table of Tamarixia radiata Waterston under controlled conditions. Rev Protección Veg. 2013;28(2):120-126.
21.Kairo MTK, Cross AE, Lopez VF, Petekin DD, Ram P. International Institute of Biological Control. Rearing the hibiscus mealybugs, Maconellicoccus hirsutus, and the parasite Anagyrus kamali Moursi. CAB International. 1997; 33 p.
22.Rodríguez H, Ramos M. Parámetros para el control de la calidad en la cría de Amblyseius largoensis (Muma) sobre Panonychus citri (McGregor). Rev Protección Veg. 2005;20(3):185-191.
23.Baños HL, Miranda I, Rodríguez MH, Sánchez A, Chico R, Martínez M. Parámetros Poblacionales de Diaphorina citri Kuwayama (Hemiptera: Liviidae) y Tamarixia radiata Waterston (Hymenoptera: Eulophidae) en condiciones naturales. Rev Protección Veg. 2015;30(1):30-39.
24.Van Lenteren JC. IOBC Internet Book of Biological Control, version 6. Waage J. and Greathead D. 1986. Insect parasitoids. 2012. Academic Press, INC. San Diego. 375.
25.Etienne J, Quilici S, Marival D, Franck A. Biological control of Diaphorina citri (Hemiptera: Psyllidae) in Guadeloupe by imported Tamarixia radiata (Hymenoptera: Eulophidae). Fruits. 2001;56(5):307-315.
Recibido: 12-5-2015.
Aceptado: 15-12-2015. ^rND^sMartínez^nML^rND^svan Lenteren^nJC^rND^sLeppla^nNC^rND^svan Lenteren^nJC^rND^svan Lenteren^nJC^rND^svan Lenteren^nJC^rND^sHale^nA^rND^sKlapwijk^nJN^rND^sSchelt^nTvan^rND^sSteinberg^nS^rND^sVan Lenteren^nJC^rND^sVan Lenteren^nJC^rND^sBoller^nEF^rND^sChambers^nDL^rND^sChien^nCC^rND^sChu^nYI^rND^sKu^nHC^rND^sGómez^nTML^rND^sMorales^nVP^rND^sFonseca^nO^rND^sNoguera^nY^rND^sCabaña^nW^rND^sRamos^nF^rND^sEscalona^nE^rND^sBaños^nHL^rND^sCeballos^nM^rND^svan Driesche^nR^rND^sHoddle^nM^rND^sCenter^nT^rND^sBaños^nHL^rND^sMiranda^nI^rND^sMartínez^nMA^rND^sRodríguez^nH^rND^sRamos^nM^rND^sBaños^nHL^rND^sMiranda^nI^rND^sRodríguez^nMH^rND^sSánchez^nA^rND^sChico^nR^rND^sMartínez^nM^rND^sVan Lenteren^nJC^rND^sEtienne^nJ^rND^sQuilici^nS^rND^sMarival^nD^rND^sFranck^nA^rND^1A01^nBenedicto^sMartínez^rND^1A01^nDanay^sInfante^rND^1A01^nBelkis^sPeteira^rND^1A01^nBenedicto^sMartínez^rND^1A01^nDanay^sInfante^rND^1A01^nBelkis^sPeteira^rND^1A01^nBenedicto^sMartínez^rND^1A01^nDanay^sInfante^rND^1A01^nBelkis^sPeteira
ARTÍCULO RESEÑA
Taxonomía polifásica y variabilidad en el género Trichoderma
Polyphasic taxonomy and variability in the genus Trichoderma
Benedicto Martínez, Danay Infante*, Belkis Peteira
Dirección de Sanidad Vegetal, Centro Nacional de Sanidad Agropecuaria (CENSA). Apartado10, San José de las Lajas, CP 32700, Mayabeque, Cuba.
RESUMEN
El objetivo del trabajo fue resumir aspectos teóricos vitales para la identificación de especies y aislados de Trichoderma, así como algunos elementos a tener en cuenta para el estudio de la variabilidad inter e intraespecífica del género. En la actualidad, aún se utilizan aspectos morfológicos para la ubicación taxonómica de las especies, pero estos resultan insuficientes y se recurre al uso de otras técnicas como son los estudios fisiológicos, de compatibilidad vegetativa y de herramientas moleculares, los cuales contribuyen a la identificación específica y certera. En los estudios de variabilidad se indica tener en cuenta aspectos fisiológicos y ecológicos, incluyendo los modos de acción de este agente de control biológico, así como la variabilidad a nivel de ADN. Ambos temas tributan directamente al conocimiento necesario para realizar una adecuada selección de las cepas que resulten promisorias, su protección como ingrediente activo de productos comerciales, monitoreo, registro y comercialización.
Palabras clave: morfología, compatibilidad vegetativa, identificación molecular.
ABSTRACT
The objective was to summarize essential theoretical aspects for the identification of Trichoderma species and isolates, as well as some aspects to be considered for studying the inter and intraspecific variability in the genus. Although the morphological aspects are currently used for the taxonomic status of the species, they are insufficient. Thus, the use of molecular techniques and other vegetative compatibility and physiological studies are important. Such techniques contribute to a specific and accurate identification. Physiological and ecological aspects, including the modes of action of this biological control agent, and the variability at the DNA level as well, must be considered in studies of variability. Both issues point directly at the necessary knowledge for the appropriate selection of promising strains, their protection as active ingredient of commercial products, monitoring, register, and marketing.
Key words: morphology, vegetative compatibility, molecular identification.
INTRODUCCIÓN
Los hongos pertenecientes al género Trichoderma son saprofitos de crecimiento muy rápido (1). Las especies se encuentran ampliamente distribuidas por todas las latitudes y se presentan naturalmente en diferentes ambientes, especialmente en aquellos que contienen materia orgánica o desechos vegetales en descomposición. Son reconocidos a nivel mundial como excelentes agentes de control biológico, debido a diferentes modos de acción, elementos esenciales para la selección eficaz de cepas promisorias para un control más eficiente y duradero (2, 3, 4).
El género Trichoderma se describió por Persoon en 1794 (5). Posteriormente, Rifai en 1969 (6) lo revisó y propuso nueve especies agregadas: Trichoderma piluliferum Webster & Rifai, Trichoderma polysporum (Link ex Pers) Rifai, Trichoderma hamatum (Bon) Bain, Trichoderma koningii Rifai, Trichoderma aureoviride Rifai, Trichoderma harzianum Rifai, Trichoderma longibrachiatum Rifai, Trichoderma pseudokoningii Rifai y Trichoderma viride Pers ex S. F Gray. Estas especies se identificaron teniendo en cuenta diferencias morfológicas y fisiológicas; sin embargo, con la taxonomía establecida sobre caracteres morfológicos no se diferencian satisfactoriamente las especies en el género.
En la actualidad se utilizan herramientas moleculares para la identificación y clasificación de los organismos. La Reacción en Cadena de la Polimerasa (PCR por sus siglas en inglés) específica de las regiones ITS1 e ITS2 y del factor de elongación, así como la secuenciación de estas para su comparación con las secuencias depositadas en GenBank TrichoBlast y en otras bases de datos, facilitan la identificación del aislamiento.
A partir de esos nuevos conocimientos se ratificaron especies y se determinaron también otras nuevas, teniendo en cuenta la variación intra e interespecífica notificada para especies del género Trichoderma en el GenBank. Esto se evidenció por Lieckfeldt et al. (7) en su estudio sobre T. viride, una de las primeras especies caracterizada e identificada únicamente por la rugosidad de la pared del conidio que presenta dos tipos morfológicamente distintos (I y II), ya que cada uno de esos tipos posee un diseño de ADN mitocondrial diferencial. Posteriores estudios moleculares, morfológicos y fisiológicos designaron al tipo I como el «verdadero» T. viride, anamorfo de Hypocrea rufa (Pers.: Fr.) y al II como una nueva especie, Trichoderma asperellum Samuels, Lieckfeldt & Nirenberg, la cual, en términos moleculares, se considera cercana al neotipo de T. hamatum. Consecuentemente, el taxa en Trichoderma incrementó de nueve a más de 100 especies en años recientes (8).
Las bondades que presentan las cepas del antagonista Trichoderma hicieron posible la elaboración de productos biológicos con características amigables con el ambiente. Pero el éxito de estas cepas como productos está amparado por una precisa selección de cepas, la cual debe partir de una certera identificación.
La correcta identificación, a través de la conjunción de diferentes técnicas, se conoce como identificación polifásica y se impone teniendo en cuenta las complejidades del género antes mencionadas. Este aspecto es vital para el registro de un producto cuyo ingrediente activo es Trichoderma, además de la importancia que reviste para la autenticación y protección del mismo, así como su monitoreo una vez liberado en campo. De igual forma, es preciso el conocimiento de la variabilidad entre los aislamientos de una colección o, incluso, entre especies del género, a fin de hacer una correcta selección de las cepas más promisorias según los aspectos fisiológicos, ecológicos, modos de acción y la misma variabilidad molecular.
Teniendo en cuenta estos aspectos el objetivo de este trabajo fue realizar una revisión actualizada sobre la identificación de aislados del género Trichoderma a partir de las diferentes técnicas disponibles para esto.
PARTE GENERAL
Trichoderma. Aspectos generales
El género Trichoderma se presenta naturalmente en diferentes hábitats, aparece distribuido por todas las latitudes desde zonas polares a ecuatoriales. Se caracteriza por ser saprófito, sobrevive en suelos con diferentes cantidades de materia orgánica debido a su gran capacidad de descomponerla. En determinadas condiciones, puede ser anaerobio facultativo. Estos atributos le permiten tener una mayor plasticidad ecológica (9). Tal capacidad de adaptación a diversas condiciones ambientales y sustratos le confiere a este género la posibilidad de ser utilizado en la industria biotecnológica (10,11).
Su amplia distribución y su plasticidad ecológica se encuentran estrechamente relacionadas con la alta capacidad enzimática que posee para degradar sustratos, un metabolismo versátil y resistencia a inhibidores microbianos (9). Conocer cuáles, en qué momento y bajo qué condiciones se favorece la expresión de estas enzimas, permite, además de la selección de cepas, orientar la forma de producción y aplicación de las mismas (12).
Identificación de aislados del género Trichoderma
· Taxonomía
La Taxonomía de este género es complicada. En 1794, Persoon describió el género Trichoderma y aún en la actualidad se continúa profundizando en este aspecto. Según Villegas (1), el género Trichoderma se ubica en la clase Hyphomycetes, orden Moniliales, familia: Moniliaceae.
Su fase sexual (estado Teleomorfo) se encuentra ubicado en la clase Ascomycetes, serie Pyrenomycetes, orden Hypocreales, género Hypocrea (1, 13, 14). Las especies del género Trichoderma son un grupo de derivados clonales de Hypocrea que han perdido la capacidad de completar un ciclo sexual (15).
Según Samuels (14), Trichoderma se clasifica como un hongo anamórfico. El estado teleomorfo se ha detectado en pocas especies. En este sentido se han identificado: Hypocrea lixii Chaverri como el estado teleomorfo de Trichoderma harzianum Rifai, Hypocrea atroviridis Dodd como el teleomorfo de Trichoderma atroviride P.karst (Bissett) e Hypocrea virens Kullnig-Gradinger como el teleomorfo de Trichoderma virens (Miller, Giddens & Foster) Arx (16, 17, 18). Las especies antes mencionadas se utilizan ampliamente como agentes de control biológico (19).
Especies
De las cuatro especies originalmente descritas por Persoon, solo se conserva Trichoderma viride Pers.: Fr., lo cual evidencia la dificultad en la identificación en este género. En trabajos posteriores, Rifai (5) determinó nueve especies agregadas sobre la base de los caracteres morfológicos, así como el tipo de ramificación del conidióforo y forma, y tamaño de los conidios, entidades morfológicamente muy difíciles de separar a nivel de especie (5). Este autor, además, señaló que T. hamatum podía incluir dos o más especies morfológicamente distintas. Todas estas especies han estado en constante variación, por lo que sus cualidades no se han definido con exactitud. A pesar de que la clasificación propuesta por Rifai sigue en uso, solo se acepta parcialmente.
Posteriormente, Bissett (20, 21, 22, 23) reexaminó los criterios de Rifai y definió cinco secciones: Trichoderma, Longibrachiatum, Saturnisporum, Pachybasium e Hypocreanum. Estas divisiones se realizaron, fundamentalmente, con el empleo de la taxonomía tradicional, según el tipo de ramificación del conidióforo, la forma del conidio, crecimiento y coloración de la colonia, entre otras.
Samuels et al. (24) realizaron otras investigaciones dentro del complejo Trichoderma, sección Longibrachiatum, las cuales permitieron un avance importante en el conocimiento de la taxonomía del género y en la comprensión de algunas especies no abordadas con anterioridad por Rifai en 1969 y Bissett en 1984.
Para depurar ambigüedades taxonómicas que aún persisten, se propusieron claves adicionales que condujeron a la redefinición de la especie T. harzianum y de su neotipo (25) y la neotipificación de T. koningii (26).
En la actualidad, la taxonomía basada en caracteres morfológicos no permite una identificación adecuada de las especies del género. Es por ello que la introducción de técnicas moleculares tiene una función decisiva en la identificación y clasificación de las especies de Trichoderma. El uso de estas herramientas por Samuels et al. en 1999 (27) trajo como consecuencia la nueva caracterización de T. viride y la descripción de Trichoderma asperellum Samuels, Lieckfeldt & Nirenberg y, en 2010, de Trichoderma asperelloides Samuels como nueva especie (28).
Las técnicas moleculares mostraron su gran potencial en estudios a nivel inter específico y, combinadas con criterios morfológicos, condujeron a la reubicación de algunas especies (7), la definición de otras nuevas (27) y/o conectar fases anamorfas con su teleomorfo (15).
Debido a la importancia del género y la complejidad para su identificación hasta nivel de especie, así como los resultados obtenidos de forma individual por diferentes autores, se evidencia la necesidad de abordar la taxonomía con un enfoque más amplio, teniendo en cuenta los aspectos morfológicos, fisiológicos, compatibilidad vegetativa y moleculares.
· Morfología
Las colonias de los aislamientos de Trichoderma presentan rápido crecimiento. En 1969 Rifai señaló que poseen color blanco y se tornan verde oscuro con abundante esporulación. En general, en medio Papa Dextrosa Agar (PDA) este hongo no presenta micelio aéreo y su pigmentación puede variar desde verde oscuro hasta verde claro y, en ocasiones, tornarse amarillento. Algunos aislamientos tienen olor típico a coco (29, 30). Durante su desarrollo y crecimiento producen hifas de 5 -10µm de ancho (31) que conforman el micelio septado, con paredes compuestas por quitina y glucano.
Los conidióforos de Trichoderma tienen aspecto cónico cuando se observan al microscopio. Producen gran cantidad de conidios asexuales unicelulares de color verde o hialinos, lisos o con paredes muy poco ásperas, subglobosos, cilíndricos, oblongos, con diámetro promedio de 3 a 5 µm. Estos se forman a partir de células conidiógenas y fiálides (singulares o en grupos), que se ubican en los extremos de los conidióforos, los cuales son hialinos muy ramificados y no verticilados. Además, este hongo puede producir clamidosporas (unicelulares), globosas en sustratos naturales, las que pueden ser intercalares y, en ocasiones, terminales en los extremos de las hifas, de tono verde y menores de 15 µm de diámetro. Estas esporas pueden perdurar a través del tiempo, por ello son consideradas estructuras de sobrevivencia (32, 33).
Para la identificación morfológica de las especies se tuvo en cuenta, principalmente, la clave de Rifai (6). Se emplea la caracterización cultural y el crecimiento de las colonias sobre diferentes medios de cultivo (Agar Extracto Malta, PDA, Cornmeal Agar), la morfometría del conidióforo, fiálides, conidios, clamidosporas, entre otros caracteres obtenidos a partir de colonias del hongo en estos medios. Más recientemente, otros autores (14, 34) diseñaron claves taxonómicas para la identificación de otras especies.
A pesar de las inexactitudes que puedan tener las claves taxonómicas, estas representan la base primaria de identificación y el punto de partida para la identificación molecular.
· Compatibilidad vegetativa
La compatibilidad e incompatibilidad somática o vegetativa, es frecuente en hongos filamentosos. De manera general, los mecanismos que la rigen permiten o evitan la coexistencia de núcleos genéticamente diferentes dentro de un citoplasma común (35).
El control genético de la incompatibilidad se estudió en Ascomycetes, y la definen diferencias en uno o más loci het (heterocarión). La existencia de numerosos loci het se dedujo a partir del gran número de grupos de compatibilidad presentes en hongos como: Neurospora crassa (Shear & B.O. Dodge), Podospora anserina (Rabenh.), Aspergillus nidulans (G Winter) y otros (35). Por ejemplo, en N. crassa la compatibilidad del heterocarión es conferida por al menos 11 loci distribuidos en 5 de los 7 grupos de ligamiento. Esto significa que para que dos aislados de esta especie sean compatibles vegetativamente, los alelos de todos los loci deben ser idénticos. De ahí que la incompatibilidad se manifiesta como una falla de fusión entre las hifas o más frecuentemente como inviabilidad del producto de la fusión, lo cual se identifica por la vacuolación, disolución de núcleos y generación de pigmentos, lo que se describió como barrera de reacción (36).
Los aislamientos agrupados, de acuerdo a los patrones de compatibilidad micelial, muestran patrones genéticos similares. Por ello, la técnica se considera como una herramienta alternativa, simple, económica y rápida para inferir similitudes entre individuos (36).
Estos grupos de compatibilidad vegetativa se consideran como una herramienta muy útil para estudiar diversidad y dinámica poblacional (37); además representan un indicador de la estrategia reproductiva, y posiblemente de la estructura genética de la población (36).
Son varios los trabajos realizados para determinar grupos de compatibilidad en Trichoderma, teniendo en cuenta que las células vegetativas pueden llegar a poseer más de 100 núcleos (24). Según Stasz et al. (38), se caracterizaron varias progenies (fusión de protoplastos) de T. harzianum, T. hamatum, T. koningii y T. viride. Varios autores describieron, dentro del género Trichoderma, la formación de heterocarión entre varias especies por medio de la anastomosis hifal, fusión de protoplastos o las transferencias nucleares (39).
· Identificación Molecular
La identificación de las especies del género Trichoderma resultó ambigua mediante la caracterización morfológica con el uso de claves tradicionales. Por estas razones se introdujeron técnicas moleculares que apoyaron y facilitaron la caracterización e identificación de las especies del género con mayor precisión. No obstante, es posible que la ambigüedad en la identificación morfológica sea una de las causas por lo que existan secuencias en las bases de datos del GenBank que no se correspondan con las especies nominadas, lo que exige una revisión de las mismas.
Dentro de las herramientas moleculares más usadas para la identificación se incluyen: cariotipos electroforéticos y la secuenciación de las regiones ITS1 e ITS2 del ADN ribosomal (ADNr) y del Factor de Elongación.
Estas técnicas, además de resolver problemas taxonómicos, proporcionaron marcadores específicos de cepas que permiten evaluar la estabilidad genética a través de diferentes generaciones sucesivas de propagación y el monitoreo de la supervivencia y destino ambiental, después de su liberación en campo (40, 41).
Actualmente, las secuencias de las especies de Trichoderma identificadas por métodos moleculares, referidas en el sitio www.isth.info de la International Commission on the Taxonomy of Fungi subcommision on Trichoderma and Hypocrea (42, 43, 44), están depositadas en: http://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=29859.
Espaciador interno del transcrito (ITS) de la región del ADN ribosomal
Las regiones del ADN de mayor interés, tanto en estudios de filogenia como en taxonomía de hongos, son las que codifican para el ARN ribosómico (45) que se encuentran en mitocondrias, cloroplastos y el núcleo. Este ADNr contiene la información para la síntesis del ARN que conforma los ribosomas, por lo que se transcribe y no se traduce (46). Este tipo de ADN está presente en más de 90 copias por genoma, puede ser amplificado fácilmente y contiene regiones que no codifican para proteínas (47).
La región del ADNr está conformada por el gen 18S (SSU), el espaciador intergénico ITS1, el gen 5.8S, el espaciador ITS2 y el gen 28S (LSU). Los genes 18S, 5.8S y 28S están relativamente conservados en los hongos y proporcionan una base molecular para investigar relaciones filogenéticas a diferentes niveles. No obstante, sus pequeños tamaños limitan su utilidad en comparaciones filogenéticas. Los espaciadores ITS, cuya función es desconocida (48), son mucho más variables porque sus transcriptos se cortan desde el ARNr y no llegan a formar parte del ribosoma (49, 50, 51). A partir de estas regiones se diseñaron cebadores universales, los cuales permiten realizar estudios evolutivos a nivel de género y familia (52).
Samuels et al. (27, 29) y Lieckfeldt et al. (7) fueron los primeros que amplificaron las regiones del ITS1-ITS2, incluyendo la región del ADNr 5.8S de la especie de T. viride. Kuhls et al. (53) lograron definir la sec. Longibrachiatum como un grupo monofilético por la secuenciación de las regiones ITS1 e ITS2, mientras que las secciones Pachybasium y Trichoderma quedaron definidas como grupos polifiléticos. Por lo informativo y la fácil amplificación, la región ITS del ADNr ha sido utilizada en numerosos estudios y, en consecuencia, ha permitido evaluar con mayor detalle la actual taxonomía de Trichoderma.
Lieckfeldt y Seifert (54) plantearon que el uso de este marcador fue desacreditado por la presencia de copias parálogas de los ITS de otros géneros de hongos relacionados con las plantas. A pesar que en la mayoría de las especies de Trichoderma / Hypocrea se encontró prueba de ello y, aunque los ITS proporcionan una pobre resolución filogenética en algunos clados, particularmente de la sección Pachybasium B (55), este marcador continúa usándose en la actualidad (14, 17, 18, 28, 33, 36, 56, 57, 58, 59, 60).
Factor de Elongación durante la Traducción (TEF)
El largo intrón de la trascripción (tef1) (56) es un fragmento de casi 2 Kb, del cual se pueden usar sus exones o intrones para el análisis filogenético. El fragmento del gen más estudiado y utilizado para la identificación de especies se conoce como tef1, el cual incluye al 4to. Intrón, el 5to. Exón, el 5to. Intrón y parte del 6to Exón, y tiene una longitud entre 600 y 650 pb. La secuenciación del factor de elongación de diferentes aislados de Trichoderma facilitó la identificación de especies (14, 17, 18, 28, 33, 55, 56, 58, 59, 60, 61, 62, 63), así como la reubicación de especies que por otras técnicas estaban erróneamente ubicadas (64)
Variabilidad en el género Trichoderma
Las especies de Trichoderma presentan una amplia plasticidad ecológica. Esta característica implica que es posible encontrar variabilidad genética importante, la cual debe ser explorada, caracterizada y finalmente explotada, con el fin de realizar una selección adecuada de las cepas promisorias. Para el uso de Trichoderma, como agente de control biológico, es necesario conocer cómo influyen estos factores bioecológicos en la fisiología de los aislados de este género, su comportamiento en cuanto a los diversos mecanismos de acción que presenta frente a las dianas que puede controlar y su variabilidad genética.
· Efecto de factores bióticos y abióticos
Dentro de estos factores se encuentran temperatura, pH, humedad relativa, iluminación y los diferentes sustratos que pueden utilizar las diferentes especies y los aislados pertenecientes al género Trichoderma. El conocimiento de la influencia de todos estos factores bioecológicos en la fisiología y las potencialidades de Trichoderma como agente de control biológico (ACB) son vitales en los procesos de selección de cepas promisorias, su reproducción masiva y su introducción en diferentes agroecosistemas para lograr una máxima efectividad del hongo en el manejo de plagas.
También existe una gran variabilidad en cuanto a los mecanismos de acción que tienen estos aislados como ACB. Entre ellos se citan la competencia por espacio y nutrientes, el micoparasitismo y la antibiosis (65).
Además de su acción biorreguladora, Trichoderma actúa de forma indirecta sobre los patógenos a través de la elicitación de mecanismos de defensa fisiológicos y bioquímicos de la planta, entre los cuales se encuentran: estimulación del crecimiento vegetal y desarrollo del sistema radicular, solubilización y absorción de nutrientes inorgánicos (N y P), inducción de resistencia y tolerancia a estreses bióticos y abióticos (66, 67).
Estos aspectos se analizaron profundamente en un artículo reciente por Martínez et al. (4), donde se hace una revisión de diferentes aspectos relacionados con la función de aislados del género Trichoderma como ACB y sus potencialidades.
· Variabilidad genética
La variabilidad inter e intraespecífica también puede ser analizada directamente en el ADN. El desarrollo de marcadores moleculares condujo a una intensa investigación y caracterización en plantas, virus, hongos, bacterias, artrópodos, nematodos. El uso de marcadores moleculares dominantes en estudios filogenéticos y de diversidad se justifica porque: 1) a diferencia de los marcadores morfológicos, los marcadores moleculares dominantes exploran múltiples loci a través de todo el genoma, por lo que son más informativos; 2) en el gen analizado pudiera encontrarse una variación de secuencias insuficiente, la cual se expresa a nivel de fenotipo o esta expresión pudiera estar influenciada por el ambiente; 3) en ocasiones los datos de la secuencia del gen estudiado pueden ser insuficientes para resolver las relaciones filogenéticas entre los grupos de estudio (68).
Es por esto que los marcadores moleculares se convirtieron en una herramienta ampliamente usada en estudios de diversidad y filogenéticos (69, 70, 71), mapeo y mejoramiento (72).
Algunas especies del género Trichoderma se caracterizaron con el uso de estas herramientas. Los cuatro tipos de marcadores moleculares más usados para determinar la diversidad genética son: RFLP (del inglés Restriction Fragment Length Polymorphism, Polimorfismo de la Longitud de los Fragmentos de Restricción) (73) (marcador codominante), RAPD (del inglés Random Amplified Polymorphic DNA, Polimorfismo del ADN Amplificado al Azar) (74), SSR (del inglés Single Sequencies Repeat, Secuencias Simples Repetidas) (75) y AFLP (del inglés Amplified Fragments Length Polymorphism, Polimorfismo de la Longitud de los Fragmentos Amplificados) (76). Cada uno de estos marcadores tiene un poder de discriminación diferente (77).
RFLP
Botstein et al. (78) describieron esta técnica, que constituyó el primer marcador de ADN utilizado para construir mapas genéticos de organismos superiores. Dicha técnica se basa en la digestión del ADN con enzimas de restricción (endonucleasas). Cada una de estas endonucleasas (de origen bacteriano), reconoce y corta una secuencia nucleotídica específica de 4-8 pb nitrogenadas en el ADN, siempre y cuando estas no estén protegidas (metiladas). Por consiguiente, cualquier ADN que no esté metilado puede ser reconocido y cortado en fragmentos de longitud definida. Cualquier mutación puntual dentro de esos sitios ocasionará pérdida o creación de un nuevo sitio de restricción en una región del genoma (79).
Es una técnica de alta repetibilidad, que permite detectar variaciones en la secuencia de ADN de 4 a 8 pb reconocidas por las enzimas utilizadas. Se aplicó a un gran número de estudios de construcción de mapas genéticos y mapeo de caracteres de interés. También se usó para resolver problemas taxonómicos o filogenéticos en Trichoderma (75, 80).
Posee como ventaja la rápida identificación de rutina, especialmente en los casos de genomas menores; es económica y confiable (si el patrón de bandas coincide con los previamente notificados). Entre las principales desventajas de la técnica se halla el requerimiento de grandes cantidades de ADN de buena calidad para la detección de loci de copias únicas. Si no se encuentra un patrón de bandas igual al descrito no se logra identificar el organismo y, además, exige de personas especializadas (81, 82).
RAPD
Esta técnica fue descrita por primera vez por Williams et al. (83) y Welsh y McClelland (84). Se basa en una PCR (del inglés Polymerase Chain Reaction, Reacción en Cadena de la Polimerasa), caracterizada esencialmente por condiciones de baja astringencia, con el uso de solo un iniciador arbitrario, con longitud de 9 a 10 nucleótidos y con un contenido de 60-70% de G + C. Las regiones del ADN, homólogas con el iniciador, son amplificadas y generan productos múltiples de loci que se distribuyen en todo el genoma. Dicha característica permitió su utilización en el mapeo, así como en la generación de huellas genéticas (36).
Esta técnica posee diversas ventajas como son la obtención de un alto número de bandas, los cebadores arbitrarios pueden ser adquiridos fácilmente sin necesidad de información nucleotídica inicial, solo se requieren pequeñas cantidades de ADN diana para la amplificación y los costos por ensayo son bajos. El poder de esta técnica se pone de manifiesto cuando se utiliza para identificar variaciones en la secuencia del ADN entre individuos. Por tal motivo, los RAPDs son una técnica muy poderosa para el estudio de la diversidad en la secuencia de una población y la localización de genes (82).
Entre las desventajas se encuentran que son esencialmente dominantes, no hay un conocimiento a priori sobre la identidad de los productos de amplificación, existen problemas de comigración y también de baja reproducibilidad de los resultados obtenidos. No obstante, esta última, aparente y discutida desventaja, se supera de manera satisfactoria al establecer adecuadamente los protocolos (85).
Esta herramienta se ha usado para estudios genéticos y taxonómicos en varios hongos, entre los que se incluyen especies del género Trichoderma (36). En este sentido, Kumar y Sharma (74) obtuvieron 100% de polimorfismo con cuatro cebadores RAPD para 12 aislados de Trichoderma spp. Latha y Mukherjee (86) encontraron, con cinco iniciadores RAPD, gran variabilidad intraespecífica entre las cepas examinadas de Trichoderma spp., excepto para dos de T. hamatum, que resultaron ser exactamente idénticas. También Hernández et al. (87) obtuvieron gran variabilidad intraespecífica en diez aislados de Trichoderma spp. con cuatro iniciadores RAPD.
SSR
Los microsatélites son secuencias de uno a diez pares de bases repetidas y adyacentes distribuidas en el genoma. Generalmente consisten en dinucleótidos (AC)n, (AG)n, (AT)n, trinucleótidos (TCT) n, (TTG) n; tetranucleótidos (TATG)n, donde n es el número de unidades repetidas dentro de un locus de microsatélite. Estos loci se encuentran en regiones codificantes y no codificantes del ADN y es probable que se formen por eventos de rompimiento que generan polimorfismos con valores superiores al 90% (88). Debido a esto, la técnica muestra altos niveles de variación genética basados en la diferencia del número de unidades repetidas en el locus. En ella, el número de productos amplificados depende de la frecuencia en que se encuentran los microsatélites utilizados como cebadores. El nivel polimórfico detectado en los productos amplificados puede estar relacionado con la diversidad genómica dentro de la especie.
Al igual que con los RAPDs, los microsatélites tienen la desventaja de que los productos obtenidos varían con las combinaciones usadas, la temperatura de acoplamiento, la concentración del cebador y del magnesio. Se demostró que ocurren problemas con el sitio de reconocimiento del cebador, aún a altas temperaturas de acoplamiento. En este caso, para incrementar la especificidad se recomienda ajustar la temperatura de acoplamiento o aplicar un PCR con decrecimiento de la misma (89). Las reacciones donde se involucran los microsatélites son más específicas que los RAPDs debido a la mayor longitud de los cebadores (15-20mers), por lo que resulta de mayor repetibilidad.
En general, los marcadores desarrollados por microsatélites son altamente polimórficos, generan gran cantidad de información debido al número y frecuencia de los alelos detectados. De aquí que los SSR se han usado satisfactoriamente para estudiar variabilidad genética en especies del género Trichoderma (74, 90).
AFLP
La técnica se describió por primera vez por Vos et al. (91) y demostró ser extremadamente versátil debido a su confiabilidad, reproducibilidad y potencia; además, porque detecta polimorfismo en todo el genoma. El principio de la técnica es utilizar Amplificación Selectiva de Fragmentos de Restricción de ADN genómico.
Valadez y Kahl (79) mencionaron que los AFLPs surgen a partir de: A) polimorfismos en los sitios de restricción, donde una secuencia específica por el reconocimiento de una endonucleasa de restricción está presente o ausente; B) polimorfismos en la longitud de la secuencia, donde el número de las secuencias repetidas arregladas en serie («tandem») tienen sitios variables; C) cambios en los pares de bases de ADN no asociados con sitios de restricción. Como desventaja, con respecto al RAPD, esta técnica requiere de más experiencia para su manipulación y, principalmente, de los geles de poliacrilamida (92).
Según Galdames (36), ambas técnicas (RAPD y AFLP) fueron lo suficientemente poderosas para detectar variabilidad genética entre aislados de Trichoderma. Sin embargo, a partir de los dendrogramas generados en cada caso, llegaron a conclusiones diferentes acerca de las relaciones genéticas entre individuos. Como resultado obtuvo que las cuatro combinaciones de los cebadores AFLP amplificaron casi el doble del producto en comparación con el número de amplicones obtenidos con los 12 iniciadores RAPDs utilizados. Aunque el polimorfismo que se detectó por los RAPDs fue mayor que el evidenciado por los AFLPs, los dendrogramas generados por este último marcador fueron más robustos, precisos y confiables.
Los resultados obtenidos hasta el momento de los estudios de variabilidad, indican que se debe usar más de un tipo de marcador, teniendo en cuenta los materiales a analizar y las ventajas y desventajas de estas técnicas. En este caso fue posible, mediante los AFLPs, detectar diferencias entre los aislados cubanos con relación a las cepas usadas como referencia, pero no entre ellos, lo que se logró con la técnica de RAPD (datos no publicados).
Debido a la alta variabilidad morfológica existente en las especies del género Trichoderma, se han introducido secuencias erróneamente identificadas por caracteres morfológicos en el Genbank; esto debe ser tomado en cuenta cuando se utilicen técnicas moleculares para su identificación.
En general, para realizar una certera identificación, tanto a nivel de especie como para llegar a la clasificación de aislamientos en el género Trichoderma, se precisa en la actualidad de la taxonomía polifásica a partir de datos morfofisiológicos y de la introducción de las técnicas moleculares y, sobre todo, de la secuenciación de ácidos nucleicos, teniendo en cuenta los nuevos avances en el tema y los cambios que se están produciendo en los últimos años.
REFERENCIAS
1. Villegas M. Trichoderma pers. Características generales y su potencial biológico en la agricultura sostenible. 2005. Disponible en: http://www.oriusbiotecnologia.com/tecnica/128-Trichoderma-pers-caracteristicas-y-su-potencialbiologico-en-la-agricultura-sostenible Consultado: 11 de marzo 2010.
2. Martínez B, Reyes Y, Infante D, González E, Baños H, Cruz A. Selección de aislamientos de Trichoderma spp. candidatos a biofungicidas para el control de Trichoderma sp. en arroz. Rev Protección Veg. 2008;23(2):118-125.
3. Infante D, Martínez B, González N, Reyes Y. Mecanismos de acción de Trichoderma frente a hongos fitopatógenos. Rev Protección Veg. 2009;24(1):14-21.
4. Martínez B, Infante D, Reyes Y. Trichoderma spp. y su función en el control de plagas en los cultivos. Rev Protección Veg. 2013;28(1):1-11.
5. Persoon CH. Disposita methodica fungorum. Römer's Neues Mag Bot. 1794;1:81-128.
6. Rifai M. A revision of the genus Trichoderma. Mycol Pap. 1969;116:1-56.
7. Lieckfeldt E, Samuels G, Nirenberg H. A morphological and molecular perspective of Trichoderma viride: is it one or two species? Applied and Environmental Microbiology. 1999;65:2418-2428.
8. Druzhinina I, Kopchinskiy A, Kubicek C. The first 100 Trichoderma species characterized by molecular data. Mycoscience. 2006;47:55-64.
9. Howell C. Mechanisms employed by Trichoderma species in the biological control of plant diseases: The history and evolution of current concepts. Plant Disease. 2003;87(1):4-10.
10.Harman G. Overview of mechanisms and uses of Trichoderma spp. Phytopathology. 2006;96(2):190-194.
11.Páez O. 2006. Uso Agrícola del Trichoderma. Disponible en: http://www.soilfertility.com/trichoderma/espagnol/index.shtml. [Consultado: 19 de marzo de 2009].
12.González I, Infante D, Martínez B, Arias Y, González N, et al. Induction of chitinases and glucanases in Trichoderma spp. strains intended for biological control. Biotecnología Aplicada. 2012;29:12-16.
13.Druzhinina I, Kubicek C. Species concepts and biodiversity in Trichoderma and Hypocrea: from aggregate species to species clusters? J Zhejiang Univ SCI. 2005;6B(2):100-112.
14.Samuels G, Dodd S, Lu B, Petrini O, Schroers H, Druzhinina I. The Trichoderma koningii aggregate species. Studies in Mycology. 2006;56:67-133.
15.Kuhls K,et al. Molecular evidence that the asexual industrial fungus Trichoderma reesei is a clonal derivative of the ascomycete Hypocrea jecorina. Proc Natl Acad Sci USA. 1996;93:7755-7760.
16.Kullnig-Gradinger C, Szakacs G, Kubicek C. Phylogeny and evolution of the genus Trichoderma: A multigene approach. Mycol Res. 2002;106:757-767.
17.Chaverri P, Castlebury L, Samuels G, Geiser D. Multilocus phylogenetic structure within the Trichoderma harzianum / Hypocrea lixii complex. Molecular Phylogenetics and Evolution. 2003;27(2):302-313.
18.Dodd S, Lieckfeldt E, Samuels G. Hypocrea atroviridis sp. nov. the teleomorph of Trichoderma atroviride. Mycologia. 2003;95:27-40.
19.Monte E. Understanding Trichoderma: Between biotecnology and microbialecology. Int Microbiol. 2001;4:1-4.
20.Bissett J. A revision of the genus Trichoderma. I. Section Longibrachiatum sect. nov. Can J Bot. 1984;62:924-931.
21.Bissett J. A revision of the genus Trichoderma. II. Infrageneric classification. Can J Bot. 1991;69(11):2357-72.
22.Bissett J. A revision of the genus Trichoderma. III. Section Pachybasium. Can J Bot. 1991;69(11):2373-417.
23.Bissett J. A revision of the genus Trichoderma. IV. Additional notes on section Longibrachiatum. Can J Bot. 1991;69:2418-2420.
24.Samuels G, Petrini O, Kuhls K, Lieckfeldt E, Kubicek C. The Hypocrea schweinitzii complex and Trichoderma sect. Longibrachiatum. Stud Mycol. 1998;41:2-54.
25.Gams W, Meyer W. What exactly is Trichoderma harzianum? Mycologia. 1998;90:904-915.
26.Lieckfeldt E, Samuels G, Börner T, Gams W. Trichoderma koningii: Neotypification and Hypocrea teleomorph. Can J Bot. 1998;76:1507-1522.
27.Samuels G, Lieckfeldt E, Nirenberg H. Trichoderma asperellum, a new species with warted conidia, and redescription of Trichoderma viride. Sydowia. 1999;51(1):71-88.
28.Samuels G, Ismaniel A, Bon M, De Respinis S, Petrini O. Trichoderma asperellum sensu lato consists of two cryptic species. Mycologia. 2010;102:944-966.
29.Samuels GJ. Trichoderma: a review of biology and systematic of the genus. Mycol Res. 1996;100(8):923-935.
30.Harman G. Trichoderma harzianum, T. viridis, T. koningii, T. hamatum (Deuteromycetes: Moniliales), 2003. Disponible en: http://www.ibun.unal.edu.co/r2r7e.html Consultado: 2 de Febrero 2007.
31.Rodríguez VJ. Efecto antagónico y biocontrolador de algunos microorganismos saprofíticos contra Rhizoctonia solani un fitopatógeno causante del (damping off) en plantas de tomate. Tesis, Universidad Nacional Mayor de San Marcos, Perú. 2002. Disponible en: http://72.30.186.56/search/cache?p=investigaciones+con+trichoderma Consultado: 30 de mayo de 2007.
32.Harman G. Trichoderma spp., including T. harzianum, T. viride, T. koningii, T. hamatum and other sp. Deuteromycetes, Moniliales (asexual classification system). 2001. Disponible en: http://www.nysaes.cornell.edu/ent/biocontrol/pathogens/trichoderma.htm. Consultado: 2 de febrero de 2010.
33.Pérez CM. Aislamientos de Trichoderma spp., nativos de Venezuela, promisorios para el control de Rhizoctonia solani (Kühn) en maíz (Zea Mays l.). 2012. Trabajo de Grado presentado como requisito final para optar al título de Magister Scientarium en Agronomía. Venezuela.
34.Jaklitsch W, Samuels G, Dodd S, Lu B-S, Druzhinina I. Hypocrea rufa/Trichoderma viride: a reassessment, and description of five closely related species with and without warted conidia. Stud Mycol. 2006;56:135-177.
35.Begueret J, Turcq B, Clavé C. Vegetative incompatibility in filamentous fungi: het genes begin to talk. Trends in Genetics. 1994;10:441-446.
36.Galdames R. Análisis genético-molecular de la diversidad del hongo patógeno Sclerotium cepivorum Berk., y del biocontrolador Trichoderma spp. Tesis en Opción al grado científico de Doctor en Ciencias Agrícolas- Biotecnología de Plantas. 2001. Guanajuato. México.
37.Leslie JF. Fungal vegetative compatibility. Annu Review Phytopathol. 1993;31:127-150.
38.Stasz TE, Nixon K, Harman GE, Weeden NF, Kuter GA. Evaluation of phenetic species and phylogenetic relationships in the genus Trichoderma by cladistic analysis of isozyme polymorphism. Mycologia. 1989;81:391-403.
39.Barcellos FG, Hungria M, Pizzirani-Kleiner AA. Limited vegetative compatibility as a cause of somatic recombination in Trichoderma pseudokoningii. Brazilian J of Microbiology. 2011;42:1625-1637.
40.Brimmer TA, Boland GJ. «A review of the non - target effects of fungi used biologically control plants diseases». Agriculture Ecosystems & Environment. 2003;100:3-16.
41.Punja ZK, Utkhede RS. Using fungi and yeast to manage vegetable crop diseases. Trends in Biotechnology. 2003;21(9):400-407.
42.Gomes EM, Kasuya E, De Barros A, Borges AM. Polymorphism in the internal transcribed spacer (ITS) of the ribosomal DNA of 26 isolates of ectomycorrhizal fungi. Genetics and Molecular Biology. 2002;25(4):477-483.
43.Zervakis GI, Moncalvo J, Vilgalys R. Molecular phylogeny, biogeography and speciation of the mushroom species Pleurotus cystidiosus and allied taxa. Microbiology. 2004;150(3):715-726.
44.Villalobos A, Escobar M, Guzmán G, Guzmán-Dávalos L. Extracción de ADN y amplificación de secuencias de ITS en Psilocybe (Agaricales, Fungi). Avances en la Investigación Científica en la CUCBA. XVI Semana de la investigación Científica. Universidad de Guadalajara, Zapopan. 2005; 548-553.
45.Iturralde J. Identificación genética de hongos. Sociedad Micológica de Madrid. España. 2005. Disponible en: http://www.socmicolmadrid.org/noti/noticias30.html. Consultado: 23 de julio 2014.
46.Rentaría M. Herramientas Moleculares. Breve revisión de los marcadores moleculares. 2007; Cap. 18: 541-566.
47.Calle J. Caracterización morfológica y molecular de hongos fitopatógenos de suelo e identificación de bacterias foliares en el cultivo de cebolla. Tesis en Opción al Grado de Maestro en Ciencias en Agronomía. Recinto Universitario de Mayagüez. Universidad de Puerto Rico. 2005.
48.Peintnen UJ, Moncalvo JM, Vilgalys R. Toward a better understanding of the intrageneric relationships in Cortinarius (Agaricales, Basidiomycota). Mycologia. 2004;96(5):1042-1058.
49.Guevara GF, Garza OF, Cázares E. Estudio del ITS nuclear en algunas especies del género Cantharellus de México. CIENCIA UANL. 2004;VII(3):371-378.
50.Tedersoo L, Suvi T, Larsson E, Köljalg U. Diversity and community structure of ectomycorrhizal fungi in a wooded meadow. Mycol Res. 2006;110:734-748.
51.Herrera P. Aislamiento de Tulasnella spp. (Basidiomycota) a partir de raíces de cuatro especies de orquídeas epifitas. Tesis previa a la obtención del título de Bioquímico-Farmacéutico Universidad Técnica Particular de Loja. 2007.
52.White TJ, Bruns T, Lee S, Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: Innis, M.A. et al. (Eds.), PCR Protocols: A guide to Methods and Applications. Academic Press, San Diego. 1990; 315-322.
53.Kuhls K, Lieckfeldt E, Samuels GJ, Meyer W, Kubicek CP, Börner T. Revision of Trichoderma section Longibrachiatum including related teleomorphs based on analysis of ribosomal DNA internal transcribed spacer sequences. Mycologia. 1997;89(3):442-60.
54.Lieckfeldt E, Seifert KA. An evaluation of the use of ITS sequences in the taxonomy of the Hypocreales. Stud Mycol. 2000;45:35-44.
55.Druzhinina I, Kopchinskiya A, Komoña M, Bissett J, Szakacsc G, Kubicek C. An oligonucleotide barcode for species identification in Trichoderma and Hypocrea. Fungal Genetics and Biology. 2005;42(10):813-828.
56.Taylor J, et al. Phylogenetic species recognition and species concepts in fungi. Fungal Genetics Biol. 2000;31:21-32.
57.Mukherjee P, Verma A, Latha J. PCR fingerprinting of some Trichoderma isolates from two Indian type culture collection-a need for re-identification of these economically importance fungi. Science Correspondence. 2002;83(4):372-374.
58.Hoyos-Carvajal L, Orduz S, Bissett J. Genetic and metabolic biodiversity of Colombia and adjacent neotropic regions. Fungal Genetics and Biology. 2009; pag. 303-320.
59.Hoyos-Carvajal L, Bissett J. Biodiversity of Trichoderma in Neotropics. The Dynamical Processes of Biodiversity - Case Studies of Evolution and Spatial Distribution. Fungal Genetics and Biology. 2010;46(9):615-631.
60.Devi P, Prabhakaran N, Kamil D, Pandey P, Lekha BJ. Characterization of Indian native isolates of Trichoderma spp. and assessment of their bio-control efficiency against plant pathogens. African J of Biotechnology. 2012;11(85):5150-15160.
61.Seibel C, Tisch D, Kubicek CP, Schmoll M. The role of pheromone receptors for communication and mating in Hypocrea jecorina (Trichoderma reesei). Fungal Genetics and Biology. 2012;49:814-824.
62.Druzhinina I, et al. Molecular phylogeny and species delimitation in the section Longibrachiatum of Trichoderma. Fungal Genet Biol. 2012;49(5):358-368.
63.Barrera VA. El género Hypocrea Fr. (Hypocreales, Ascomycota) en la Argentina. Estudio de la variabilidad molecular de su estado anamórfico Trichoderma. Tesis en Opción al Grado de Doctor en Ciencias. Fuente: Biblioteca Digital de la Facultad de Ciencias Exactas y Naturales - Universidad de Buenos Aires. 2012.
64.Martínez B, Infante D, Reyes Y. About to identification of some Trichoderma isolates reported in Revista de Protección Vegetal. Rev Protección Veg. 2010;25(2):135.
65.Harman G. Myths and dogmas of biocontrol: changes in perceptions derived from research on Trichoderma harzianum T-22. Plant Dis. 2000;84:377-393.
66.Harman G. Overview of mechanisms and uses of Trichoderma spp. Phytopathology. 2006;96(2):190-194.
67.Vinale F, Sivasithamparamb K, Ghisalbertic EL, Marraa R, Woo L, Lorito M. Trichoderma-plant-pathogen interactions. Soil Biology & Biochemistry. 2008;40:1-10.
68.Simmons SL, Bazylinski DA, Edwards KJ. Population dynamics of marine magnetotactic bacteria in a meromictic salt pond described with qPCR. Environ Microbiol. 2007;9(9):2162-2174.
69.Marhold K, Lihova J, Perny M, Bleeker W. Comparative ITS and AFLP analysis of diploid Cardamine (Brassicaceae) taxa from closely related polyploid complexes. Annals of Botany. 2004;93:507-20.
70.Sasanuma T, Chabane K, Endo TR, Valkoun J. Characterization of genetic variation in and phylogenetic relationships among diploid Aegilops species by AFLP: incongruity of chloroplast and nuclear data. Theoretical and Applied Genetics. 2004;108:612-618.
71.Furini A, Wunder J. Analysis of eggplant (Solanum melongena)-related germplasm: morphological and AFLP data contribute to phylogenetic interpretations and germplasm utilization. Theoretical and Applied Genetics. 2004;108:197-208.
72.Thabuis A, et al. Marker-assisted introgression of 4 Phytophthora capsici resistance QTL alleles into a bell pepper line: validation of additive and epistatic effects. Molecular Breeding. 2004;14:9-20.
73.Ali A, Bajwa R, Mehmood N, Jabeen R. Molecular Screening of Trichoderma isolates. J Bio-Sci. 2009;17:117-122.
74.Kumar MA, Sharma P. Molecular and morphological characters: An appurtenance for antagonism in Trichoderma spp. African J of Biotechnology. 2011;10(22):4532-4543.
75.Kretzner AM, Molina R, Spatafora JW. Microsatellite markers for the ectomycorrhizal basidiomycete Rhizopogon vinicolor. Mol Ecol. 2000;9:1190-1191.
76.Buhariwalla HK, Srilakshmi P, Kannan S, Kanchi RS, Chndra S, Satyaprasad K, et al. AFLP analysis of Trichoderma spp. from India compared with sequence and morphological-based diagnostics. J of Phytopathology. 2005;153(7-8):389-400.
77.Couteaudier Y, Viaud M, Neuveglise C, Bridge P, Clarkson J. Combination of different independent molecular markers to understand the genetic structure of Beauveria populations. In: Bridge P. and Couteaudier Y. (eds) Molecular variability of Fungal Pathogens. CAB International, Wallinford. 1998; pp 95-104.
78.Botstein D, White RL, Skolnick M, Davis RV. Construction of a genetic linkage map in man using restriction fragment length polymorphism. Amer J Hum Genetic. 1980;32:314-331.
79.Valadez E, Kahl G. Huellas de ADN en genomas de plantas (Teoría y protocolos de laboratorio). 1era edición. Mundi-Prensa. 147p. 2000.
80.Ahmad Israr, Bhagat Someshwar, Kumar Krishna, Birah Ajanta, Tripathi AK, Madhuri K, et al. PCR-RFLP marker based DNA amplified fragments and diversity assessment of Trichoderma spp. from Andaman and Nicobar Islands. J of Mycopathological Research. 2012;50(1):55-59.
81.Segura G, Luis E, Kirchmayr Manuel R, Flores B, Ericka P, Gschaedler M, et al. PCR-RFLP de las regiones ITS-5.8S como herramienta de identificación de levaduras: ventajas y desventajas. e-Gnosis. 2010;8:1-12.
82.Cornide MT, Arencibia RA, Berovides VA, Calvo DP, Canales EL, Coto OA, et al. Marcadores moleculares. Nuevos horizontes en la genética y la selección de plantas. Ed. Félix Varela.367 Págs., 2002.
83.Williams JGK, Kubelik AR, Livak KJ, Rafalski JA, Tingey SV. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res. 1990;18(22):6531-6535.
84.Welsh J, McClelland M. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Res. 1990;18(24):7213-7218.
85.Rifalski JA. Randomly amplified polymorphic DNA (RAPD) analysis. En: Caetano-Anollés, G. y P.M. Gresshoff (eds.) DNA markers: protocols, application and overviews. Wiley-VCH. 1997;5:75-83.
86.Latha J, Verma A, Mukherjee P. PCR-fingerprinting of some Trichoderma isolates from two Indian type culture collections - a need for re-identification of these economically important fungi. Current Science. 2002;83(4):372-374.
87.Hernández A, Jiménez M, Arcia A, Ulacio D, Méndez N. Caracterización molecular de doce aislamientos de Trichoderma spp. mediante RAPD y rADN-ITS. Bioagro. 2013;25(3):167-174.
88.Coltman DW, Bowen DW, Weith JM. PCR primers for harbour seal (Phoca vitulina concolour) microsatellites amplify polymorphic loci other pinniped species. Molecular Ecology. 1996;5:161-163.
89.Vogel JM, Scolnilk PA. Direct amplification from microsatellites: detection of simple sequence repeat- based polymorphisms without cloning. En Caetano-Anollés, G. y P.M. Gresshoff (eds.). DNA markers: protocols, application and overviews. Wiley-VCH. 1997;9:133-150.
90.Stenglein SA. Caracterización de Trichoderma con marcadores moleculares ISSR y RAPD. Conferencia en Taller Latinoamericano: Biocontrol de fitopatógeno con Trichoderma y otros antagonistas. Ciudad de La Habana Cuba 28-31 marzo 2006. Abstract. Fitosanidad. 2006;10(2):138-139.
91.Vos AP, et al. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res. 1995;23:4407-4414.
92.Macías FO, Delgado CY, Peña ME, León BR, Elías BR. Técnicas para el diagnóstico y determinación de variabilidad genética de fitopatógenos. 30p. 2006. Disponible en: http://www.bibliociencias.cu/gsdl/collect/libros/index/assoc/HASH01d2/441186ed.dir/doc.pdf. Consultado 12 de junio 2014.
Recibido: 20-11-2014.
Aceptado: 10-9-2015.
* Correspondencia: Danay Infante. Correo electrónico: danay@censa.edu.cu.