INTRODUCCIÓN
La soya (Glycine max (L), Merril) se encuentra ampliamente distribuida en el mundo y es afectada por una amplia variedad de fitopatógenos. Entre las principales enfermedades se encuentran las causadas por fitoplasmas (1) y begomovirus (2,3), que disminuyen la productividad de los cultivos y ocasionan pérdidas económicas de gran valor.
En Cuba, se informó la infección por fitoplasmas en cultivos de soya de la región oriental del país (4) y la presencia de begomovirus (5) en plantas sintomáticas colectadas en la provincia Las Tunas (región oriental de Cuba). Posteriormente, Chang et al. (6) identificaron a Rhynchosia golden mosaic Yucatan virus (RhGMYuV) en áreas comerciales del cultivo en la provincia Mayabeque y estudios recientes confirman la presencia del Tobacco yellow crinkle virus (TbYCV) (Leyva R.M et al; datos no publicados) en plantas de soya de la región oriental del país. De igual modo, otros estudios muestran la presencia de 15 % de infecciones mixtas de begomovirus y fitoplasmas (Leyva R.M; Datos no publicados) en el cultivo.
La Reacción en Cadena de la Polimerasa (PCR, del inglés Polymerase Chain Reaction) es ampliamente utilizada para el diagnóstico de fitopatógenos (7). En el caso de los fitoplasmas, que están distribuidos irregularmente y presentan bajas concentraciones en el floema de plantas infectadas, su diagnóstico se realiza con técnicas de alta sensibilidad como la PCR anidado (nPCR, del inglés nested PCR) (8). La nPCR permite la detección más sensible y específica de estos patógenos (9). De igual modo, para la detección de begomovirus se evalúan variantes de nPCR para lograr una detección de mayor sensibilidad y especificidad (10).
La detección simultánea de varios patógenos en una simple reacción de PCR múltiple demostró que es una herramienta valiosa para la detección de fitopatógenos, porque en la naturaleza más de un patógeno infecta un cultivo u hospedante (11). Estas herramientas facilitan la detección e identificación simultánea de distintos agentes de interés, con lo que contribuyen sustancialmente a obtener un diagnóstico rápido y preciso. Estos ensayos reducen los costos, ahorran tiempo y son más eficientes para la detección de este tipo de infecciones (12).
El objetivo del trabajo fue desarrollar un ensayo de nPCR múltiple para la detección simultánea de fitoplasmas y begomovirus identificados en cultivos de soya en Cuba.
MATERIALES Y MÉTODOS
La estandarización del método de nPCR múltiple se basó en la combinación del uso de los cebadores universales para la detección de fitoplasmas (R16mF2 (5’-CATGCAAGTCGAACGGA-3’) / R16mR1 (5’-CATGCAAGTC GAACGGA-3’) que amplifican un fragmento de 1450 pb del gen ARNr 16S en la primera reacción. En la segunda reacción se utilizaron los cebadores R16F2n (5’-GAACGGCGGTGTGTACAAACCC-3’)/R16R2 (5’-TGACGGGCGGTGTGT ACACCCG-3’) que amplifican un fragmento de 1250 pb) y las condiciones descritas en la literatura (13) con la utilización de cebadores diseñados en este estudio para la detección de begomovirus mediante nPCR.
Análisis in silico de la capacidad de los cebadores diseñados para la detección de los begomovirus Rhynchosia golden mosaic Yucatan virus y Tobacco yellow crinkle virus
Para la detección de los begomovirus se procedió al diseño de cebadores tomando como base las secuencias de los begomovirus Rhynchosia golden mosaic Yucatan virus (RhGMYuV) (KT192632, KT381193) y Tobacco yellow crinkle virus (TbYCV) (KU562964) identificados en cultivos de soya en Cuba y secuencias de referencia de las mismas especies: RhGMYuV (KP641349, KP641347, EU02121, GQ352453) y de TbYCV (FJ222587, FJ213931, KP641345), disponibles en la base de datos del Genbank.
Los cebadores se diseñaron teniendo en cuenta los parámetros establecidos en la literatura (14). Para el análisis se separaron las secuencias de nucleótidos de las proteínas de la cápsida (CP) y de replicación (Rep) de los virus, mediante el programa ORF Finder (http://www.ncbi.nlm.nih.gov/projets/gorf/orfig.cgi). Las secuencias nucleotídicas de las proteínas se alinearon con el módulo MUSCLE, implementado en el programa MEGA (versión 6) (15).
Mediante el programa SnapGene (versión 1.1.3) se comprobó la amplificación de una única secuencia diana en el genoma de los begomovirus identificados y la generación de amplicones de tamaños diferentes a los que se producen en la PCR anidada para fitoplasmas. Se comprobó que los cebadores alinearan a la secuencia molde con el programa MEGA (versión 6); se analizaron mediante la herramienta Blastn y se sintetizaron por el Centro de Ingeniería Genética y Biotecnología de Cuba (CIGB).
Ensayo de PCR anidada preliminar para la detección de begomovirus
A partir del resultado del análisis in silico de los cebadores diseñados para la detección de los begomovirus identificados, se realizó un ensayo preliminar de la PCR, como base para la optimización de los parámetros críticos.
Como moldes se utilizaron controles positivos (ADN plasmídico) de los begomovirus Rhynchosia golden mosaic Yucatan virus (RhGMYuV) y Tobacco yellow crinkle virus (TbYCV), disponibles en el laboratorio. Estos controles se usaron, además, para el desarrollo de los ensayos de estandarización de la técnica. Como control negativo se utilizó un ADN plasmídico que contiene el genoma del Bean golden yellow mosaic virus (BGYMV) obtenido de plantas de frijol positivas, disponible en el laboratorio de Virología Vegetal del Centro Nacional de Sanidad Agropecuaria (CENSA). Los controles se evaluaron según las condiciones descritas para la nPCR de fitoplasmas (13).
La primera reacción se realizó en un volumen final de 25 µl que contenía la solución amortiguadora de PCR 10X (Invitrogen, Brasil); MgCl2 50 mM; dNTPs 10 mM; cebadores 10 mM cada uno; 2U de Taq ADN Polimerasa (Invitrogen), agua libre de nucleasas (Promega, Medison, WI, E.U.A.) y ADN plasmídico (10 ng/µl). En la segunda reacción, la mezcla contuvo los mismos componentes a la misma concentración utilizada en la primera reacción. Se adicionó 1 μl de una dilución 1:30 del producto obtenido en la primera reacción.
Para ambas amplificaciones, se usó un termociclador programable (eppendorf Mastercycler gradient). Los productos de la PCR (10,0 μl de cada producto) se analizaron mediante electroforesis en gel de agarosa al 1 %, en tampón TBE 1X y en cámara de electroforesis durante 45 minutos a 70 voltios, teñido con 1,0 µg. ml-1 de bromuro de etidio, de acuerdo al procedimiento descrito por Sambrook et al. (16) y se observaron bajo un transiluminador de luz ultravioleta (UV) Syngene InGenius L (Syngene, EE.UU). Para determinar la talla aproximada de los productos se incluyó un marcador de peso molecular (1 Kb DNA Plus Ladder) (Invitrogen).
Evaluación de los parámetros críticos para la nPCR para la detección de begomovirus
Se realizaron ensayos de PCR para la evaluación de los parámetros críticos de la técnica; estos fueron: temperatura óptima de alineamiento de los cebadores al molde, sensibilidad analítica y especificidad de los cebadores.
Para los ensayos se utilizaron, como controles positivos, ADN plasmídico de los begomovirus RhGMYuV y TbYCV; como control negativo un ADN plasmídico que contiene el genoma del BGYMV. A su vez, en cada etapa de la estandarización se varió una condición y las restantes se mantuvieron constantes. Se realizaron dos réplicas de cada evaluación.
Evaluación de diferentes temperaturas de alineamiento de los cebadores diseñados para detección de los begomovirus
Los cebadores diseñados se ensayaron a diferentes temperaturas de alineamiento (Tabla 1) mediante la evaluación de un gradiente de temperatura en un termociclador programable (Eppendorf Mastercycler gradient), en una reacción constituida por: una solución tamponada al 10X, MgCl2 (50 mM), dNTPs (10 mM): 2 u de Enzima Taq ADN polimerasa (Invitrogen); ADN plasmídico (10 ng); 10 pmol de cada cebador diseñados para la primera reacción, para un volumen final de reacción de 25 µl. Para la segunda reacción solo variaron los cebadores y los perfiles térmicos utilizados. Para este ensayo solo se usó como control positivo el ADN plasmídico de RhGMYuV. El resto de las condiciones de los ensayos de PCR se realizaron de acuerdo a lo descrito previamente para la PCR anidada preliminar.
Tabla 1 Perfiles térmicos utilizados para la evaluación de un gradiente de temperaturas para la nPCR con los cebadores diseñados para la detección de begomovirus./ Thermal profiles used for the evaluation of a temperature gradient for nPCR with primers designed for the detection of begomoviruses
| Cebadores | Desnaturalización inicial | 35 ciclos | Extensión final | ||
|---|---|---|---|---|---|
| Desnaturalización | Alineamiento | Extensión | |||
| PrepIcu-15F | 950C/4min | 950C/ 1min | 530c, 540c, 550c, 570c | 720C/2 Min | 720C/5 Min |
| PcpIcu-15R | |||||
| PrepEcu-15F | 950C/4min | 950C/ 1min | 570c, 590c, 600c, 630c | 720C/2 Min | 720C/5 Min |
| PcpEcu-15R | |||||
Determinación de la sensibilidad analítica de los cebadores diseñados
Para determinar la sensibilidad analítica de los cebadores diseñados para la detección de begomovirus, utilizados en la primera reacción, se empleó como control positivo ADN plasmídico de RhGMYuV de concentración conocida: 100; 10; 1; 0,10; 0,01 y 0,001 ng/μl. La sensibilidad analítica de los cebadores diseñados para la reacción anidada se evaluó a partir de concentraciones de 0,1; 0,01; 0,001 ng/μl del plásmido. El resto de las condiciones del ensayo se realizaron de acuerdo a lo descrito previamente para la PCR anidada preliminar.
Evaluación de la especificidad de los cebadores diseñados
Para evaluar la especificidad de los cebadores diseñados se tomaron ADN total de dos controles positivos a BGYMV, ADN plasmídico de dos controles positivos a RhGMYuV y dos a TbYCV. El resto de las condiciones del ensayo se realizaron de acuerdo a lo descrito previamente para la PCR anidada preliminar.
Evaluación de las características diagnósticas de un ensayo de PCR múltiple anidada para la detección de begomovirus y fitoplasmas
Evaluación de los parámetros críticos para la PCR múltiple anidada
Se realizaron ensayos de PCR para la evaluación de los parámetros críticos del ensayo, estos fueron: evaluación de diferentes combinaciones de concentraciones de los cebadores diseñados con las condiciones establecidas para nPCR de fitoplasmas, sensibilidad analítica y la repetitividad intra e interensayo. En cada ensayo se varió una condición y las restantes se mantuvieron constantes. Se realizaron dos réplicas de cada evaluación.
Evaluación de diferentes combinaciones de concentraciones de los cebadores diseñados con las condiciones establecidas para nPCR de fitoplasmas
La evaluación se realizó a partir de la segunda reacción de la PCR. Se evaluaron diferentes combinaciones de concentración de los cebadores (PrepIcu-15F/PcpIcu-15R: 10 pmol, 5 pmol, 3 pmol y 2 pmol); mientras que, la concentración de los cebadores R16F2n/R16R2 se mantuvo constante (10 pmol). Como controles positivos se utilizaron el ADN plasmídico de los begomovirus RhGMYuV y TbYCV, así como ADN plasmídico de fitoplasmas. Todos los controles se evaluaron a una concentración de 10 ng/µl. El resto de las condiciones del ensayo se realizaron de acuerdo a lo descrito previamente para la PCR anidada preliminar. Como control negativo se utilizó agua ultrapura.
Evaluación del límite de detección o sensibilidad analítica de la PCR anidada múltiple
A partir de la mezcla de 1 ul de ADN total, de concentración conocida (100 ng/µl), obtenido de plantas de soya positivas a begomovirus (TbYCV) y de ADN total de muestras positivas a fitoplasmas se realizó el análisis mediante nPCR múltiple. A un microlitro del producto de la primera reacción, se le realizaron diluciones de 1:30, 1:100 y de 1:1000 en agua ultra-pura y estas se sometieron a la reacción anidada de la PCR múltiple.
Evaluación de la repetitividad intra e interensayo
Se seleccionaron tres controles positivos que presentaban infección mixta a begomovirus y fitoplasmas (ADN total de plantas de soya positivas a TbYCV y fitoplasmas) y un control negativo (planta de soya negativa a ambos patógenos). Se realizaron dos repeticiones intraensayos e interensayos de la nPCR con las condiciones seleccionadas de acuerdo a los resultados de los experimentos anteriores.
Evaluación de parámetros de desempeño del ensayo de nPCR múltiple
Se evaluaron por el método estandarizado 52 muestras como controles positivos (muestras de plantas de soya evaluadas previamente en las que se determinó infección mixta por fitoplasmas y begomovirus, utilizando los métodos descritos anteriormente) y 50 muestras negativas a ambos patógenos, evaluadas previamente para confirmar su negatividad.
Se calcularon los índices o parámetros de desempeño analíticos del ensayo (17); estos se muestran a continuación:
Sensibilidad Diagnóstico (DSe)= Vp / (Vp + Fn)
Especificidad Diagnóstico (DSp)= Vn / (Vn + Fp)
Eficacia (E)= Vp + Vn / Vp + Vn + Fp +Fn
Valor Predictivo Positividad (Vpp)= Vp / (Vp + Fp)
Valor Predictivo Negatividad (Vpn)= Vn / (Vn + Fn)
Clave: Vp=Verdaderos positivos, Vn=Verdaderos negativos, Fn=Falsos negativos y Fp=Falsos positivo
RESULTADOS Y DISCUSIÓN
Análisis in silico de la capacidad de los cebadores diseñados para la detección de los begomovirus Rhynchosia golden mosaic Yucatan virus y Tobacco yellow crinkle virus
Como resultado de la evaluación in silico se seleccionaron dos parejas de cebadores que se denominaron PrepEcu-15F/PcpEcu-15R para la primera reacción y PrepIcu-15F/PcpIcu-15R para la reacción anidada como las adecuadas para el diagnóstico específico múltiple de los begomovirus identificados infectando soya en Cuba. Este análisis permitió conocer, de manera teórica, que los cebadores diseñados amplifican un fragmento que comprende una parte de la proteína de la replicación viral (Rep), toda la región intergénica y parte de la proteína de la cápsida (CP) de los begomovirus RhGMYuV y TbYCV (Tabla 2).
Tabla 2 Cebadores diseñados en este estudio para la detección de los begomovirus RhGMYuV y TbYCV/ Primers designed in this study for detection of RhGMYuV and TbYCV begomoviruses
| Reacción | Cebadores | Secuencia 5´-3´ | Posición de alineamiento RhGMYuV | Posición de alineamiento TbYCV | RhGMYuV | TbYCV | ||
|---|---|---|---|---|---|---|---|---|
| Tm | fragmento | Tm | fragmento | |||||
| ºC | (pb) | ºC | (pb) | |||||
| Primera | PrepEcu-15F | GAGGAGAGTTGAAACGGAGG | 1877-1906 | 1885-1904 | 55 | 1007 | 55 | 1026 |
| PcpEcu-15R | CTGGGCTTCCTRTACATRGGC | 291-311 | 289-309 | 61 | 61 | |||
| Anidada | PrepIcu-15F | CATGGTTCCGGAGCCTT | 1911-1927 | 1909-1925 | 55 | 968 | 55 | 988 |
| PcpIcu-15R | CTRTACATRGGCCTRTTAA CC | 282-302 | 280-300 | 57 | 54 | |||
El análisis de los cebadores permitió confirmar que no forman estructuras secundarias ni dímeros; además presentan enlaces fuertes en los extremos 3´. La secuencia de los cebadores y las posiciones que ocupan en el genoma viral se muestran en la Tabla 2; así como las temperaturas de alineamiento (Tm) de los cebadores diseñados y el tamaño de los fragmentos a amplificar. Las mismas se encuentran en el rango en que alinean los cebadores utilizados en la detección genérica de fitoplasmas.
La evaluación in silico de los cebadores diseñados para el diagnóstico específico de los begomovirus, posibilitó predecir el rango de evaluación de la temperatura adecuada de hibridación de los cebadores PrepEcu-15F/PcpEcu-15R (57, 59, 61, 63ºC) y PrepIcu-15F/PcpIcu-15R (53, 54, 55, 57ºC), la posición de hibridación de los cebadores al molde, la longitud de la secuencia diana, la temperatura de fusión y el porcentaje de guanina-citosina (Resultados no mostrados).
Estos resultados teóricos permitieron establecer el punto de partida para la optimización de los parámetros críticos. Todo ello evidenció, desde un punto de vista teórico, que los cebadores son capaces de hibridarse a las regiones de interés, lo que sugiere su utilidad para el diagnóstico específico de los begomovirus RhGMYuV y TbYCV.
Ensayo de PCR anidada preliminar para la detección de begomovirus
La Figura 1 muestra, de forma representativa, la evaluación de controles positivos mediante el nPCR preliminar. El ensayo mostró una banda de aproximadamente 988 pb para la detección del begomovirus TbYCV y 968 pb en RhGMYuV, para las réplicas de ADN evaluadas en la reacción con las condiciones empleadas. En el control negativo no se observó amplificación, lo cual garantiza la confiabilidad de esta detección. (Fig. 1)
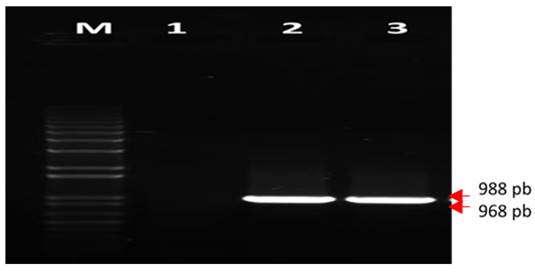
Figura 1 Análisis electroforético en un gel de agarosa al 1 %, que muestra los resultados de la PCR preliminar para la detección de los begomovirus TbYCV y RhGMYuV. M: marcador de peso molecular. Carril 1 control negativo. Carriles 2: control positivo al begomovirus TbYCV y carril 3: control positivo al begomovirus RhGMYuV / Electrophoretic analysis on 1% agarose gel that shows the results of the preliminary PCR for detection of the TbYCV and RhGMYuV begomoviruses. M: molecular weight marker. Lane 1 negative control. Lane 2: TbYCV begomovirus positive control and lane 3: RhGMYuV begomovirus positive control
El ensayo preliminar de nPCR mostró las bandas esperadas, coincidiendo con los resultados obtenidos en el análisis in silico de los cebadores diseñados, bajo las condiciones empleadas en la reacción. Este resultado demuestra la capacidad y pertinencia de los cebadores para detectar los begomovirus TbYCV y RhGMYuV.
Evaluación de los parámetros críticos para la nPCR para la detección de begomovirus
Evaluación de diferentes temperaturas de alineamiento de los cebadores diseñados para detección de los begomovirus
Los ensayos correspondientes a la PCR gradiente, realizadas a temperaturas de hibridación de 59, 60 y 63ºC para los cebadores PrepEcu-15F/PcpEcu-15R (Fig. 2A), amplificaron un fragmento de igual talla (aproximadamente 1007 pb), mientras que en la temperatura de 57ºC no se observó amplificación. El análisis electroforético mostró bandas intensas y bien definidas.

Figura 2 Resultados del análisis electroforético en un gel de agarosa al 1% que muestra las temperaturas de alineamiento de los cebadores diseñados. Figura 2-A, productos de PCR de los cebadores PrepEcu-15F/PcpEcu-15R a Tm de 57, 59, 60 y 63ºC. Figura 2-B, productos de PCR de los cebadores PrepIcu-15F/PcpIcu-15R a Tm de 53, 54, 55 y 57ºC/ Results of the electrophoretic analysis on 1% agarose gel that shows the alignment temperatures of the designed primers.Figure 2-A, PCR products of the PrepEcu-15F / PcpEcu-15R primers at Tm of 57, 59, 60 and 63°C.Figure 2-B, PCR products of PrepIcu-15F / PcpIcu-15R primers at Tm of 53, 54, 55, and 57°C.
El ensayo demostró la capacidad de los cebadores diseñados para detectar los begomovirus en estudio al amplificar el fragmento esperado a Tm de 59, 60 y 63ºC. Se seleccionó como valor adecuado 60ºC, por coincidir con la temperatura de amplificación utilizada en la detección genérica de fitoplasmas.
Para el caso de cebadores diseñados para la reacción anidada PrepIcu-15F/PcpIcu-15R, estos fueron capaces de amplificar el fragmento esperado (aproximadamente 968 pb) en las cuatro temperaturas evaluadas (Fig. 2-B). Se tomó como valor adecuado la temperatura de 55ºC, valor que coincide con los valores previstos en los análisis in silico de los cebadores y los perfiles térmicos utilizados en la nPCR para la detección genérica de fitoplasmas.
Estos resultados demuestran que la temperatura adecuada de alineamiento al ADN molde puede calcularse de manera teórica; sin embargo, es preciso determinarla experimentalmente. Los resultados obtenidos por Majumder et al. (18) confirman este planteamiento en la estandarización de una RT-PCR, los que permitieron determinar temperaturas adecuadas de 46 y 48ºC al evaluar un rango de 44 a 52ºC, respectivamente.
Los resultados expuestos confirman la capacidad que ofrecen los cebadores diseñados para la detección de los begomovirus bajo las condiciones establecidas para la detección de fitoplasmas.
Evaluación de la sensibilidad analítica de los cebadores diseñados para la detección de begomovirus
La pareja de cebadores (PrepEcu-15F/PcpEcu-15R) diseñados para primera reacción de PCR permitió la detección de hasta 0,1 ng de ADN del patógeno (Fig. 3A); mientras que los cebadores (PrepIcu-15F/PcpIcu-15R) diseñados para la reacción anidada de la PCR posibilitaron detectar hasta 0,01 ng de ADN molde (Fig. 3-B).
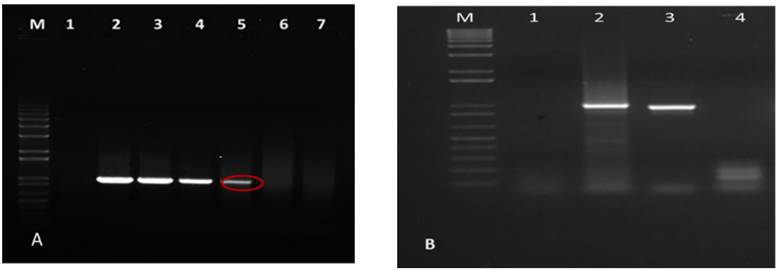
Figura 3 Resultados de la electroforesis en un gel de agarosa al 1 % que muestra el límite de detección de los cebadores diseñados. Figura 3-A PrepEcu-15F/PcpEcu-15R. Carril: 1- control negativo. Carril: 2=100 ng, Carril: 3= 10ng, Carril: 4= 1 ng, Carril: 5- 0,1 ng. Figura 3-B. PrepIcu-15F/PcpIcu-15R. Carril 1- control negativo. Carril: 2- 0,1. Carril: 3 -0,01 ng. Carril: 4- 0,001 ng. Carriles M: marcadores de peso molecular 1kb plus DNA Ladder./ Electrophoresis results on 1% agarose gel that shows the detection limit of the designed primers.Figure 3-APrepEcu-15F / PcpEcu-15R. Lane: 1- negative control. Lane: 2 = 100 ng, Lane: 3 = 10ng, Lane: 4 = 1 ng, Lane: 5- 0.1 ng.Figure 3-BPrepIcu-15F / PcpIcu-15R. Lane 1- negative control. Lane: 2- 0.1. Lane: 3 -0.01 ng. Lane: 4- 0.001 ng. Lanes M: 1kb plus DNA Ladder molecular weight markers
Este resultado demuestra la alta sensibilidad analítica de los cebadores diseñados para la detección de los begomovirus. La sensibilidad analítica de los cebadores es un factor importante en la detección del patógeno en cuestión, más aún en el caso de patógenos cuyas concentraciones en el hospedante son bajas o presentan una distribución irregular debido a la influencia de muchos factores, como las diferentes partes de las plantas analizadas, las condiciones del ambiente y el momento del cultivo al ser infectado (19).
Evaluación de la especificidad de los cebadores diseñados para la detección de begomovirus
En la evaluación de la especificidad analítica, mediante nPCR de los cebadores diseñados para la detección de begomovirus, no se amplificaron fragmentos de ADN en los controles positivos utilizados de la especie del begomovirus BGYMV, ni en el control negativo utilizado en la reacción, mientras que en los controles positivos utilizados de las especies de begomovirus TbYCV y RhGMYuV se amplificó una banda del tamaño esperado. (Fig. 4)

Figura 4 Resultados de la electroforesis en un gel de agarosa al 1 %, que muestra la detección de begomovirus mediante nPCR. Carriles. M: Marcador de peso molecular 1kb plus DNA Ladder. Carril 1: control negativo. Carriles 2: control positivo (TbYCV). Carriles 3-4 controles positivos a BGYMV. Carriles 5-6: controles positivos a TbYCV y carriles 7-8: controles positivos a RhGMYuV./ Electrophoresis results on 1% agarose gel that shows the detection of begomovirus by nPCR. Lane M: Molecular weight marker 1kb plus DNA Ladder. Lane 1: negative control. Lane 2: positive control (TbYCV). Lanes 3-4 positive controls to BGYMV. Lanes 5-6: TbYCV positive controls, and lanes 7-8: RhGMYuV positive controls
Este resultado demuestra la especificidad analítica del método de diagnóstico, ya que los cebadores y las condiciones evaluadas permiten la detección específica del ADN molde específico de los begomovirus TbYCV y RhGMYuV; y no de otros patógenos utilizados como controles en el ensayo (20).
Evaluación de las características diagnósticas de un ensayo de PCR múltiple anidada para la detección de begomovirus y fitoplasmas
Evaluación de diferentes concentraciones utilizando combinaciones de los cebadores mediante nPCR múltiple
Para determinar la concentración adecuada de los cebadores, el ensayo amplificó un fragmento de igual talla (1250 pb para fitoplasmas y 968 pb para begomovirus) en todas las réplicas de ADN donde se evaluaron las combinaciones R16F2n/R16R2 (10 pmol) /PrepIcu-15F/PcpIcu-15R (5 pmol); R16F2n/R16R2 (10 pmol) / PrepIcu-15F/PcpIcu-15R (3 pmol) y R16F2n/R16R2 (10 pmol) /PrepIcu-15F/ PcpIcu-15R (2 pmol). (Figura 5 A y B)
El análisis electroforético mostró que, a medida que disminuyó la concentración de los cebadores PrepIcu-15F/PcpIcu-15R desde 5 pmol a 2 pmol, aumentó ligeramente la intensidad de las bandas para fitoplasmas; en todas las combinaciones las bandas para begomovirus resultaron intensas y bien definidas. (Fig. 5B)
Los resultados alcanzados permitieron seleccionar como las combinaciones más adecuadas las R16F2n/R16R2 (10 pmol)/PrepIcu-15F/PcpIcu-15R (3 pmol) y R16F2n/R16R2 (10 pmol)/ PrepIcu-15F/PcpIcu-15R (2 pmol), utilizando como molde de 100 ng/µl de ADNt, al amplificar bandas intensas y bien definidas, tanto para la detección de fitoplasmas como begomovirus. (Fig. 5B)

Figura 5 Resultados de la electroforesis en un gel de agarosa a 1,5 % que muestra la evaluación de las concentraciones de cebadores a 10 pmol para la detección de fitoplasmas (1250 pb) y 10, 5, 3 y 2 pmol para la detección de begomovirus (968 pb). Figura 5-A. Carriles 2-5: Muestras de ADNt de plantas por amplificación con iniciadores R16F2n/R16R2 (10 pmol) y PrepIcu-15F/PcpIcu-15R (10 pmol), carriles 10-13: muestras ADN plantas por amplificación con iniciadores R16F2n/R16R2 (10 pmol) y PrepIcu-15F/PcpIcu-15R (5 pmol) carriles 6 y 14, 7 y 15 controles positivos a begomovirus y fitoplasmas respectivamente Carril 1: Control negativo. Carril M: 1 Kb plus DNA Ladder, (Invitrogen Life Technologies, EE.UU) carril 8 vacío. Figura 5-B. Carriles 2-5: Muestras ADN plantas por amplificación con iniciadores R16F2n/R16R2 (10pmol) y PrepIcu-15F/PcpIcu-15R (3pmol), carriles 8-11: muestras ADN plantas por amplificación con iniciadores R16F2n/R16R2 (10pmol) y PrepIcu-15F/PcpIcu-15R (2pmol) carriles 6-12 y 7-13 controles positivos a begomovirus y fitoplasmas respectivamente Carril 1: Control negativo. Carril M: 1 Kb plus DNA Ladder, (Invitrogen Life Technologies, EE.UU)./ Results of electrophoresis on 1.5% agarose gel showing the evaluation of the concentrations of 10 pmol primers for the detection of phytoplasms (1250 bp) and 10, 5, 3 and 2 pmol for the detection of begomovirus ( 968 bp).Figure 5-A. Lanes 2-5: Plant cDNA samples by amplification with R16F2n / R16R2 (10 pmol) and PrepIcu-15F / PcpIcu-15R (10 pmol), lanes 10-13: plant DNA samples by amplification with R16F2n / R16R2 (10pmol) initiators and PrepIcu-15F / PcpIcu-15R (5 pmol) lanes 6 and 14, 7 and 15 positive controls for begomoviruses and phytoplasmas respectively Lane 1: Negative control. Lane M: 1 Kb plus DNA Ladder, (Invitrogen Life Technologies, USA) lane 8: empty.Figure 5-BLanes 2-5: Plant DNA samples by amplification with primers R16F2n / R16R2 (10pmol) and PrepIcu-15F / PcpIcu-15R (3pmol), lanes 8-11: plant DNA samples by amplification with initiators R16F2n / R16R2 (10pmol) and PrepIcu -15F / PcpIcu-15R (2pmol) lanes 6-12 and 7-13 positive controls to begomoviruses and phytoplasmas, respectively Lane 1: Negative control. Lane M: 1 Kb plus DNA Ladder, (Invitrogen Life Technologies, USA)
Este resultado pudiera estar dado a que la concentración de los begomovirus en plantas de soya es superior a la concentración de fitoplasmas cuando coexisten en el mismo hospedante; razón por la cual la intensidad de las bandas en la detección de fitoplasmas es tenue a iguales concentraciones de cebadores. También pudiera estar asociado a que el tamaño de la banda a amplificar en los begomovirus es menor que la que se obtiene para fitoplasmas y con esto la sensibilidad en la detección de begomovirus es superior.
Por otra parte, la concentración de los cebadores es un parámetro que influye en el funcionamiento adecuado de la PCR, debido a que debe favorecer el desplazamiento de la reacción hacia la formación de los productos; sin embargo, una alta concentración podría provocar la formación de estructuras secundarias o las uniones inespecíficas al ADN molde (21).
Teniendo en cuenta los resultados obtenidos en cada ensayo de evaluación, se propone establecer las siguientes condiciones para la primera reacción de PCR: para un volumen de la mezcla de la reacción de 25 μl: buffer (10X): MgCl2 (50 mM): dNTPs (10 mM), cebadores (10 pmol), 2 u Taq ADN polimerasa (invitrogen), ADNt (100 ng/µl).
Para la reacción anidada se propone que un volumen final de 25 μl contenga: buffer (10X), MgCl2 (50 mM), dNTPs (10mM), cebadores para detección de fitoplasmas (10 pmol), cebadores para la detección de begomovirus (0.2 pmol), Taq ADN polimerasa (invitrogen), 1 ul de producto de la primera reacción de PCR (dilución 1:30). Los perfiles térmicos del programa de amplificación para el método propuesto son los utilizados para la detección genérica de fitoplasmas (13).
Evaluación de la sensibilidad analítica de la PCR anidada múltiple
La nPCR múltiple presentó un límite de detección de 1:30 para productos de PCR de la primera amplificación de fitoplasmas, mientras que para begomovirus el fragmento esperado se observó hasta la dilución 1:1000 (Fig. 6). Los resultados alcanzados no coinciden con Acosta et al. (22), quienes detectaron las bandas esperadas para productos de PCR de la primera amplificación de fitoplasmas hasta diluciones de 1:1000 al evaluar la sensibilidad analítica en ensayos múltiples para la detección de fitoplasmas y rickettsias en plantas de papayo.
Este resultado puede estar dado a que los fitoplasmas se encuentren en menor concentración que los begomovirus en los tejidos de las plantas, así como a una distribución irregular dentro de los haces vasculares.

Figura 6 Resultados de la electroforesis en un gel de agarosa a 1,5 %. Se muestran productos de nPCR a partir de diferentes diluciones de los productos de la primera reacción. Carriles M: marcador de peso molecular (1Kb DNA Plus Ladder), carril 1: control negativo; carriles 2-3 (1:30); 6-7 (1:100); 10-11 (1:1000). Los carriles 4, 8 y 12 controles positivos a begomovirus y los carriles 5, 9 y 13 controles positivos a fitoplasmas. / Results of the electrophoresis on 1.5 % agarose gel showing nPCR products from different dilutions of the first reaction products. Lanes M: Molecular weight marker (1kb plus DNA Ladder), lane 1: negative conrol, lanes 2-3 (1:30); 6-7 (1:100); 10-11 (1:1000). Lanes 4, 8, and 12: controls positive to begomoviruses, and lanes 5, 9, and 13:controls positive to phytoplasms.
Evaluación de la repetitividad intra e interensayo
Las repeticiones efectuadas intra e interensayo mostraron 100 % de concordancia (Fig. 7), por lo que no fue necesario realizar análisis estadísticos que determinaran diferencias entre ellos.

Figura 7 Análisis electroforético en gel de agarosa al 1,5 % de los productos de la nPCR múltiple, para evaluar la repetitividad del método. A: Intraensayos; B: Interensayos. M: Patrón de peso molecular 1 KB DNA Plus ladder Carril 1: control negativo carriles 2-4 repetición de 3 muestras de ADN total extraídos a partir de los controles positivos. / Electrophoretic analysis on 1.5 % agarose gel of the nPCR products to evaluate the method repeatability. A: Intra-assays, B: Inter-assays. M: Molecular weight marker (1kb plus DNA Ladder). Lane 1: negative control; lanes 2-4: repetition of 3 DNA samples extracted from the positive controls.
La repetitividad del ensayo proporciona información importante sobre el ensayo antes de que se lleve a cabo una validación posterior. Si se encuentra una excesiva variabilidad, habrá que corregirla antes de continuar con el proceso de validación (23).
Evaluación de parámetros de desempeño del nPCR múltiple y comparación con ensayos de nPCR
En la Tabla 3 se observa el comportamiento de los controles positivos y negativos de fitoplasmas y begomovius utilizados para la comparación del nPCR múltiple con los nPCR individuales para ambos patógenos, los cuales permitieron realizar el cálculo de los parámetros de desempeño. (Tabla 4)
Al comparar las técnicas de nPCR y nPCR múltiple se demostró la factibilidad de ambas para detectar fitoplasmas y begomovirus, tanto en forma simple como simultánea. El ensayo propuesto mostró una eficacia de 96 % para detectar los fitoplasmas y de 98 % para detectar los begomovirus; cifras que demuestra la factibilidad de su aplicación en el diagnóstico múltiple de estos patógenos.
Tabla 3 Resultados de la evaluación de controles positivos y negativos para la detección de fitoplasmas y begomovirus por nPCR y nPCR múltiple./ Results of the comparison of the techniques for detection of phytoplasmas and begomoviruses individually and simultaneously by nPCR and multiple nPCR
| Muestras | nPCR | nPCR múltiple | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Fitoplasmas | Begomovirus | Fitoplasmas | Begomovirus | |||||||||
| + | - | + | - | + | - | + | - | |||||
| Positivas | 52 | 50 | 2 | 52 | 51 | 1 | 52 | 49 | 3 | 52 | 50 | 2 |
| Negativas | 50 | 0 | 50 | 50 | 0 | 50 | 50 | 0 | 50 | 50 | 0 | 50 |
Los parámetros de desempeño de la técnica se muestran en la Tabla 4, los cuales oscilaron con valores superiores a 96,1 % para nPCR y 92,6 % para nPCR múltiple. Los valores de sensibilidad de la nPCR fueron superiores a los valores obtenidos mediante la nPCR múltiple. Los valores obtenidos para los parámetros evaluados en la nPCR múltiple se correlacionaron por la presencia de tres falsos negativos en la detección de fitoplasmas y dos falsos negativos en la detección de begomovirus (Tabla 3). Este resultado pudiera estar asociado a la distribución irregular de los patógenos en la planta o a muy baja concentración en el material vegetal utilizado como muestra experimental, lo cual dificulta el diagnóstico (24).
Tabla 4 Comparación de los parámetros evaluativos entre nPCR para fitoplasmas y nPCR para begomovirus con nPCR múltiple. / Comparison of the evaluation parameters between nPCR for phytoplasmas and nPCR for begomoviruses with multiple nPCR
| Parámetros Evaluativos (%) | nPCR | nPCR | nPCR múltiple | |
|---|---|---|---|---|
| Fitoplasma | Begomovirus | Fitoplasma | Begomovirus | |
| Sensibilidad diagnóstica (DSe) | 96,2 | 98,1 | 92,3 | 96,1 |
| Especificidad diagnóstica (DSp) | 100 | 100 | 100 | 100 |
| Eficacia (E) | 98 | 99 | 96,1 | 98 |
| Valor Predictivo Positivo (Vpp) | 100 | 100 | 100 | 100 |
| Valor Predictivo Negativo (Vpn) | 96,1 | 98,0 | 92,6 | 96,1 |
Los resultados demuestran que es posible la utilización de la nPCR múltiple para la detección de infecciones mixtas de fitoplasmas y los begomovirus identificados infectando el cultivo de soya en Cuba, con porcentajes de DSe, DSp y eficacia superior a 90 %; similar a los resultados alcanzados mediante nPCR para su identificación de forma independiente. Este resultado argumenta lo obtenido por otros autores en la evaluación de parámetros para el establecimiento de métodos de diagnóstico a virus (25) y bacterias (26), por lo que los ensayos descritos en este trabajo demuestran adecuados valores de sensibilidad y especificidad; indican, además, que ambas son factibles de implementar para la detección de ambos patógenos.
El empleo de la nPCR múltiple permitirá reducir costos y riesgos de contaminación e incrementar la rapidez del diagnóstico (27). La PCR múltiple ha sido utilizada en la detección de fitoplasmas y begomovirus en cultivos de tomate en México (28), del begomovirus Corchorus golden mosaic virus (CoGMV), un fitoplasma perteneciente al grupo 16SrV-C (29) y en cultivos de Berenjena (12), entre otros.
El PCR anidado múltiple desarrollado en este trabajo para la detección de fitoplasmas y begomovirus que infectan soya en Cuba constituye una herramienta valiosa a utilizar en la detección de ambos patógenos en los programas de manejo integrado y mejoramiento genético de este cultivo, ya que permite un diagnóstico confirmativo de estas entidades. De igual modo, es de gran utilidad para un laboratorio de diagnóstico, pues permite disminuir considerablemente el tiempo en que se obtiene el resultado, el esfuerzo, los costos y el riesgo de contaminación cruzada, así como aumentar la capacidad diagnóstica de la detección.














