Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Vaccimonitor
versión impresa ISSN 1025-028X
Vaccimonitor vol.21 no.1 Ciudad de la Habana ene.-abr. 2012
ARTÍCULO ORIGINAL
Normalización de un sistema de reacción en cadena de la polimerasa en tiempo real para la cuantificación de papilomavirus humano de alto riesgo oncogénico
Standardization of a real-time based polymerase chain reaction system for the quantification of human papilomavirus of high oncogenic risk
Yudira Soto,1* Vivian Kourí,1 Pedro Ariel Martínez,1 Consuelo Correa1 , Griselda Torres,2 Adibel Goicolea,3 Luis Morier,1 Virginia Capó,2 Lissette Pérez1, Yoan Alemán 1 , Hermis Rodríguez1, Alina Álvarez1
1Laboratorio de Infecciones de Transmisión Sexual, Departamento de Virología. Instituto de Medicina Tropical «Pedro Kourí» (IPK). Centro Colaborador OPS/OMS para el Control de Enfermedades Virales. Novia del Mediodía Km 6 ½. La Habana, Cuba.
2Laboratorio de Anatomía Patológica, Subdirección de Atención Médica, Instituto de Medicina Tropical "Pedro Kourí" (IPK). Centro Colaborador OPS/OMS para el Control de Enfermedades Virales. Novia del Mediodía Km 6 ½. La Habana, Cuba.
3Hospital Ginecobstétrico "Eusebio Hernández". Marianao, La Habana, Cuba
email:yudira@ipk.sld.cu
* Máster en Ciencias, Licenciada en Microbiología, Investigadora Agregada y Profesora Asistente.
RESUMEN
El objetivo de este trabajo fue normalizar e implementar un sistema de reacción en cadena de la polimerasa en tiempo real, para determinar la carga viral de 7 genotipos de papilomavirus humano (PVH) de alto riesgo oncogénico. Se evaluó la especificidad del sistema y se construyeron las curvas estándar para PVH 16 y 18, que se emplearon para la cuantificación de ADN viral en diferentes muestras de pacientes identificados como positivos a PVH, mediante reacción en cadena de la polimerasa (RCP) cualitativa y secuenciación nucleotídica. Se obtuvieron dos curvas estándar para PVH 16 y 18, a partir del ADN genómico de las líneas celulares SiHa y HeLa, las que mostraron una buena correlación lineal ( r = -0,99) y valores bajos de error. El límite inferior de detección a partir del ADN de las líneas celulares fue de hasta 10 copias para ambos genotipos. No se obtuvo reacción cruzada entre los diferentes tipos de PVH ni con otros virus ADN. La reacción en cadena de la polimerasa en tiempo real (RCP-TR) normalizada probó ser un sistema simple, rápido, específico y altamente sensible. Además, permitirá desarrollar investigaciones sobre la prevalencia de infección por PVH en Cuba, con vistas a la aplicación de las vacunas que se encuentran disponibles en el mercado internacional, así como la evaluación de otros candidatos vacunales diseñados en el futuro.
Palabras clave: Papilomavirus humano, lesiones intraepiteliales, reacción en cadena de la polimerasa en tiempo real.
ABSTRACT
The objective of the present study is to standardize a real-time based polymerase chain reaction system in order to detect and quantify 7 high risk human papillomavirus in different clinical samples from patients suspected of this type of infection. The validation of a 5´ exonuclease fluorescent probe real-time PCR assay (TaqMan format) for the detection and quantification of the 7 most frequent HR-HPV types (16, 18, 31, 33, 45, and 58) which account for over 87% of cervical carcinomas world-wide was carried out. Simultaneous PCR reactions are required to detect the designated HPV types. Specificity tests for each HPV type and other DNA viruses were performed. Standard external curve constructions were achieved, which allow determining the number of target DNA copies in the previously HPV tested samples. HPV 16 and 18 standard curves were obtained from purified genomic DNA of SiHa and HeLa cell lines, respectively. The pattern curves were constructed on the basis of each of the resulting standard DNA, which showed good linear correlation (r = -0, 99) and low error values. The lower detection limit was 10 copies for both HPV 16 and 18. No cross reactions between HPV types and other DNA viruses were observed. Real-Time Polymerase Chain Reaction system, standardized for 7 HPV types, proved to be a rapid, specific and highly sensitive system for better diagnosis and follow-up of patients with high grade intraepithelial lesions. In addition, this assay will allow the development of coming researches in relation with the prevalence and pathogenesis of human papillomavirus infections in different samples from Cuban patients.
Keywords : Human papillomavirus, Intraepithelial Lesions, Real-time Polymerase Chain Reaction.
INTRODUCCIÓN
Actualmente se ha establecido que los papilomavirus humanos (PVH) son los agentes causales de más del 90% de los cánceres del cuello uterino. Además, se han visto asociados con el cáncer anal, carcinoma esofágico, así como con el desarrollo de carcinomas celulares escamosos de cabeza y cuello, no relacionados con el consumo de alcohol o tabaco (1).
Los PVH se dividen en dos grupos, según su asociación con las lesiones malignas: de alto y bajo riesgo oncogénico. Los tipos 6 y 11 de bajo riesgo se detectan hasta en el 90% de los casos de verrugas anogenitales, mientras que los tipos 16 y 18 de alto riesgo oncogénico se pueden diagnosticar entre el 70% y el 80% de los casos de cáncer cervicouterino (2).
El incremento de la carga viral es un indicador que se ha podido correlacionar con la progresión del cáncer cervical (3). También ha sido posible correlacionar los niveles altos en la carga viral de PVH 16 con el desarrollo de carcinomas celulares escamosos de la orofaringe (4). La asociación entre la presencia de genotipos de alto o bajo potencial oncogénico, la cuantificación de la carga viral y el desarrollo de las lesiones constituyen herramientas valiosas para el monitoreo de la progresión de la enfermedad, así como para definir conductas y tratamientos (4).
Un argumento de gran relevancia práctica que apoya la necesidad de la genotipificación y cuantificación viral es que se requiere de ensayos eficientes en cualquier laboratorio para poder definir la eficacia del empleo de las vacunas que se encuentran disponibles en el mercado mundial: la tetravalente contra PVH 6, 11, 16 y 18 (Gardasil® /Silgard®) y la bivalente contra PVH 16 y 18 (Cervarix®) (5).
La RCP constituye un método sensible para la detección de ADN de PVH. Actualmente, el uso de oligonucleótidos degenerados se combina con reacciones de RCP anidada para ganar en sensibilidad y especificidad (6, 7). También se usa la secuenciación nucleotídica directamente, a partir del producto de la RCP de la primera reacción, con el fin de identificar los genotipos mayoritarios en las muestras clínicas (8). Existen, además, estuches comerciales que permiten hibridar los productos de la RCP con sondas de ADN específicas para cada genotipo, las cuales permiten identificar los genotipos de PVH presentes en cada muestra clínica, como es el caso de los sistemas Amplicor o Linnear Array, comercializados por la Roche (9). Todos estos ensayos son laboriosos, consumen tiempo, no permiten la cuantificación del ADN viral y por lo tanto no aportan datos sobre el papel de la carga viral en la progresión de la enfermedad.
La RCP cuantitativa o RCP en tiempo real (RCP-TR) permite la cuantificación del ADN viral hasta 8 órdenes de magnitud con la ventaja de que los procesos de amplificación y detección se producen de manera simultánea en el mismo tubo de reacción, sin necesidad de manipulación posterior; se elimina el riesgo de liberación al ambiente de ácidos nucleicos amplificados que pueden ser la causa de contaminaciones en la RCP convencional (10).
La mayoría de los ensayos que se han desarrollado para la cuantificación de la carga viral de PVH han estado dirigidos a la detección de PVH 16 o 18 (11, 12). Sin embargo, se han publicado relativamente pocos métodos capaces de detectar y cuantificar otros genotipos de PVH y muy pocos trabajos dirigidos a la detección y cuantificación de la carga viral de diferentes genotipos de PVH de alto y bajo riesgo oncogénico.
Estos ensayos difieren en el diseño de oligos y sondas. Además, emplean diferentes plataformas o sistemas, incluyendo la tecnología Roche y Applied Biosystem , que son las que más ensayos clínicos han publicado (13-15).
Recientemente, Schmitz y cols describieron un novedoso método que permite detectar y cuantificar 7 de los genotipos de alto riesgo oncogénico (PVH 16, 18, 31, 33, 45, 52 y 58 ) que son los reportados mundialmente en el 87, 4% de las lesiones malignas (5).
En dicho estudio reportan que se puede lograr la detección diferencial de estos genotipos y de un control interno para el gen de la â globina humana en dos mezclas de reacción, empleando la metodología TaqMan o sonda de hidrólisis a través de la plataforma Applied Biosystem .
Al no contar con técnicas cuantitativas para el diagnóstico y monitoreo de las infecciones por PVH en pacientes infectados con estos virus, ni para evaluar el uso futuro de la vacuna contra los PVH que incluye los tipos 16 y 18, el colectivo de autores se propuso normalizar el protocolo antes mencionado (5).
De esta manera, implementar una RCP-TR para identificar 7 genotipos de PVH de alto riesgo oncogénico (PVH 16, 18, 31, 33, 45, 52 y 58), adaptando la metodología a la plataforma Light Cycler de Roche e introducir dicha herramienta diagnóstica en el laboratorio de Infecciones de Transmisión Sexual (ITS) del Departamento de Virología del Instituto de Medicina Tropical «Pedro Kourí» (IPK).
MÉTODOS
Líneas celulares y muestras clínicas
Líneas celulares: Se emplearon las líneas de carcinoma cervical humano de la Colección Americana de Cultivos Tipo (American Type Culture Collection, ATCC) transformadas por PVH 16, SiHa (HTB 35, ATCC, Rockville, Md) y por PVH 18, HeLa (CRL 1550, ATCC). Estas líneas celulares contienen entre 1 y 2 copias de PVH 16, y entre 10 y 50 copias de PVH 18 por célula, respectivamente (5, 15). Dichas células fueron cultivadas en medio Eagle-MEM suplementado con suero fetal bovino al 10%, con 100 unidades/mL de penicilina y 100 µg/mL de estreptomicina a 37 °C en atmósfera de CO2 al 5%.
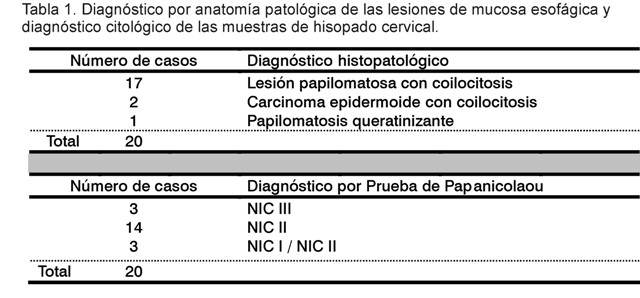
Muestras clínicas: Para la implementación del sistema se analizaron 20 muestras de mucosa esofágica obtenidas por biopsia, mediante ponche. Dichas muestras tenían un diagnóstico histológico previo, sugestivo de infección por PVH, realizado en el Laboratorio de Anatomía Patológica del IPK. Además, se estudiaron 20 muestras de células cervicouterinas a partir de un diagnóstico preliminar por la prueba de Papanicolaou, realizado en el hospital ginecobstétrico "Eusebio Hernández", donde se identificaron como neoplasia intraepitelial cervical de alto grado. Cada uno de los diagnósticos citológicos e histológicos aparecen en la Tabla 1.
Todas las muestras seleccionadas fueron analizadas para la detección de PVH mediante RCP cualitativa (6, 7). La genotipificación se realizó empleando secuenciación nucleotídica. Luego de realizar el diagnóstico de PVH a cada paciente, se informaron los resultados al médico de asistencia, quién indicó el tratamiento y seguimiento adecuado.
Ética: El empleo de muestras clínicas en esta investigación fue aprobado por el Comité de Ética de la institución auspiciadora. En todos los casos se obtuvo el consentimiento informado de los pacientes para formar parte la misma. Se les informó que este estudio se realizaría teniendo en cuenta los principios éticos de la investigación clínica en humanos, máxima confidencialidad; que los resultados solo serían utilizados con fines científicos sin revelar su identidad y se les brindó, además, la oportunidad de salir del estudio en cualquier momento que lo consideraran.
Extracción del ADN: Para la purificación de ADN genómico (ADNg) de las líneas celulares se contaron las células en cámara de Newbauer, y partiendo de una cantidad conocida de células se extrajo el ADNg de 10 6 células. Para la obtención del ADN de las muestras clínicas a partir de tejido fresco, se procesaron fragmentos de mucosa esofágica de aproximadamente 2 mm3 . Las muestras obtenidas por hisopado de células cervicales se colectaron y transportaron en un 1 mL de medio de trasporte para PVH (DIGENE, EEUU). Para la purificación del ADNg de las líneas celulares y de todas las muestras clínicas se empleó el estuche comercial QIAamp® DNA Mini Kit (QIAGEN, Alemania ) , según las indicaciones del fabricante. El ADN obtenido se resuspendió en 100 µL de tampón de elusión.
RCP cualitativa y secuenciación nucleotídica: El ADN obtenido de las muestras clínicas fue sometido a una primera RCP con los oligos MY09/MY11 (7) y a una segunda reacción o RCP anidada empleando los cebadores GP5+/GP6+, según los protocolos descritos previamente (6). Los productos amplificados a partir de la primera reacción fueron purificados empleando el estuche comercial QIAquick® PCR Purification (QIAGEN, Alemania ) y siguiendo las instrucciones del fabricante.
Posteriormente, dichos productos fueron empleados en reacciones de secuenciación nucleotídica, con el fin de establecer el genotipo de PVH mayoritario en cada muestra clínica. Para las reacciones de secuenciación se emplearon los mismos cebadores utilizados en la RCP: MY09 (cebador en sentido +) y MY11 (cebador en sentido -), para obtener secuencias en ambos sentidos. El producto secuenciado fue de 450 pares de base, el cual se corresponde con un fragmento del gen más conservado para la familia Papillomaviridae que codifica para la proteína L1 de PVH que es la proteína mayoritaria de la cápside viral.
Se prepararon mezclas constituidas por 5 pmoles de cada uno de los cebadores especificados, 8 µL de la mezcla de reacción de secuencia DTCS Quick Star Master Mix (suministrada con el Dye Terminator Cycle Sequencing (DTCS) Quick Start Kit, de Beckman Coulter, EEUU), 5 µL de ADN purificado (aproximadamente 100 ng) y 6 µL de agua, libre de nucleasas, para RCP, hasta completar 20 µL de reacción.
La reacción de secuencia consta de una desnaturalización por dos min a 96 ºC, seguida de 50 ciclos con 20 s de desnaturalización a 96 ºC, 20 s de hibridación a 50 ºC y 4 min de extensión a 60 ºC. Una vez concluida la reacción de secuencia se realizó la purificación del producto siguiendo el protocolo descrito en el estuche comercial DTCS Quick Star Master Mix (Beckman Coulter, EEUU). La corrida de la reacción de secuencia se realizó en un secuenciador automático Beckman Coulter modelo CEQ TM 8800 (EEUU), utilizando el procedimiento de análisis de datos crudos para productos de RCP. Las secuencias nucleotídicas obtenidas fueron sometidas a un proceso básico de alineamiento (BLAST, del inglés Basic Local Alignment Search Tool ). Dicho programa permitió la comparación de las mismas con todas las secuencias disponibles en las bases de datos del sitio de internet www//ncbi.nih.gov , y finalmente definir el genotipo mayoritario de PVH presente en cada muestra clínica.
Normalización de la RCP-TR y preparación de las curvas estándar
En la implementación de este método se realizaron varias modificaciones, aunque se empleó la metodología TaqMan o Sonda de hidrólisis, al igual que en el protocolo original, se trabajó sobre la plataforma Light Cycler de Roche en lugar de Applied Biosystem (5). Esta modificación condujo a la eliminación del protocolo de reacciones de RCP múltiples, por lo que se emplearon reacciones simples de RCP-TR por muestra y por genotipo. Para este fin las sondas se marcaron con los fluorocromos BHQ-1a-6-FAM y TAMRA-6-FAM (BHQ: Black Hole Quencher; FAM: 6-Fam), ya que el equipamiento Light Cycler 1.5 de Roche solo consta de dos canales de lectura de fluorescencia.
Confección de las curvas estándar: Se estableció el número de copias de cada uno de los ADNg obtenidos a partir de cada línea celular. Para ello se correlacionó el número de copias de PVH por célula con el número de células que se sometió a extracción del ADNg. Se procesaron 10 millones de células para ambas líneas celulares.
Una vez conocido el número de copias de los diferentes ADNg se procedió a la confección de dos curvas estándar a partir de cada uno de ellos. Para esto se realizaron diluciones seriadas en base 10 en H 2 0 libre de ARNasa-ADNasas de los dos productos. En el caso de PVH 16 desde 105 copias hasta 10 copias de ADN viral/µL y en el caso de PVH 18 desde 106 copias hasta 10 copias de ADN viral/µL.
Se realizaron tres réplicas por cada una de las diluciones de los productos de ADNg. Se añadieron 5 µL de cada dilución a capilares que contenían 15 µL de una mezcla formada por: 8 µL de H 2O, 4 µL de mezcla universal de RCP ( Roche ), 10 pmoles y entre 1-5 pmoles de oligonucleótidos y sonda, respectivamente. Las secuencias de los cebadores y de las sondas fueron reportadas previamente por Schmitz y cols (5). Los parámetros del ciclaje fueron los siguientes: 94 °C por 10 min y 45 ciclos compuestos por 94 °C durante 15 s, 50 °C por 20 s y 60 °C durante 40 s.
Al finalizar el último ciclo, por medio de análisis de regresión lineal, el programa del Light Cycler 1.5 produce un gráfico de la curva estándar en el que relaciona la concentración logarítmica (eje x) con el punto de corte (Cp) de cada uno de los estándares (eje y), con 95% de intervalo de confianza. Cada uno de los ADN estándares obtenidos fue trabajado por separado en días diferentes.
Para la construcción de las curvas estándar se utilizaron los valores medios de los Cp de cada una de las réplicas y se siguieron las instrucciones del fabricante. Se empleó el método de la derivación secundaria máxima (SDMM: siglas del inglés Second Derivative Maximum Method ), versión 3.3 del programa del Light Cycler 1.5 de Roche . Posteriormente, las curvas externas estándar, se exportaron y archivaron en el equipo.
Aplicación de la RCP-TR a las muestras clínicas
En las muestras clínicas seleccionadas se identificaron y cuantificaron los 7 genotipos virales de PVH (PVH 16, 18, 31, 33, 45, 52 y 58), mediante el protocolo descrito anteriormente, para la confección de las curvas estándar. Las muestras se tipificaron y se cuantificó la carga viral para cada genotipo de PVH, empleando las curvas estándar previamente confeccionadas. En todos los casos se empleó como control interno un fragmento del gen que codifica para la â globina humana, para demostrar la calidad del ADN utilizado, según recomiendan los autores del protocolo original (5).
Análisis estadístico
Para la selección del mejor método para determinar la carga viral de PVH se tuvieron en cuenta varios parámetros, los cuales se calculan de manera automática por el programa del Light Cycler (versión 3.3), entre ellos están: pendiente de la curva (Slope) : se utiliza para evaluar la eficiencia de la reacción. Según las recomendaciones del fabricante para lograr una eficiencia de la curva estándar entre 1,5 y 2,2, su valor debe estar entre 5,7 y 2,9; Error : señala las variaciones entre capilar y capilar (ejemplo, por errores al pipetear). Un valor de 0,6 corresponde a una desviación del valor x (concentración) de hasta 50%. Intercepción (Intercept): se utiliza para evaluar la sensibilidad de la reacción de la RCP-TR; mientras menor sea el valor del Cp en la ecuación de regresión mayor será la sensibilidad del sistema; r: c oeficiente de regresión. Ofrece un control de la adecuada distribución lineal de la curva. Señala posibles errores sistemáticos ( ejemplo, error acumulado en las diluciones seriadas). También se estableció el límite de detección de la RCP-TR normalizada, se identificó la última dilución en la que el sistema fue capaz de detectar el ADN diana en el mismo Cp para todas las réplicas.
Especificidad del sistema
Para evaluar la especificidad del ensayo y descartar posibles falsos positivos se sometieron a amplificación, mediante la RCP-TR normalizada, los ADN previamente extraídos de tres cepas no relacionadas: citomegalovirus humano (CMVh; AD169, ATCC), virus de la varicela zóster [(VVZ) aislamiento clínico, Laboratorio de ITS, Virología, IPK)] y virus del herpes simple 2 [(VHS-2), aislamiento clínico, Laboratorio de ITS, Virología, IPK).
Se determinó, además, que no existieran amplificaciones inespecíficas entre los genotipos presentes en cada una de las líneas celulares empleadas como controles para la confección de las curvas estándar. Así mismo se evaluó la especificidad del sistema para los diferentes genotipos obtenidos en las muestras clínicas analizadas por RCP cualitativa-secuenciación nucleotídica y mediante el método normalizado.
RESULTADOS
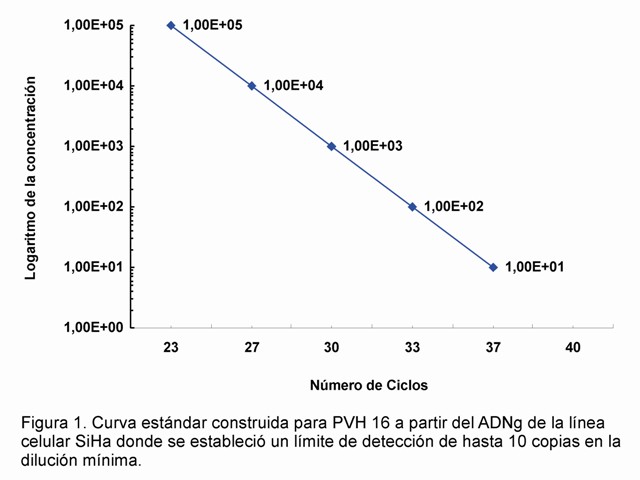
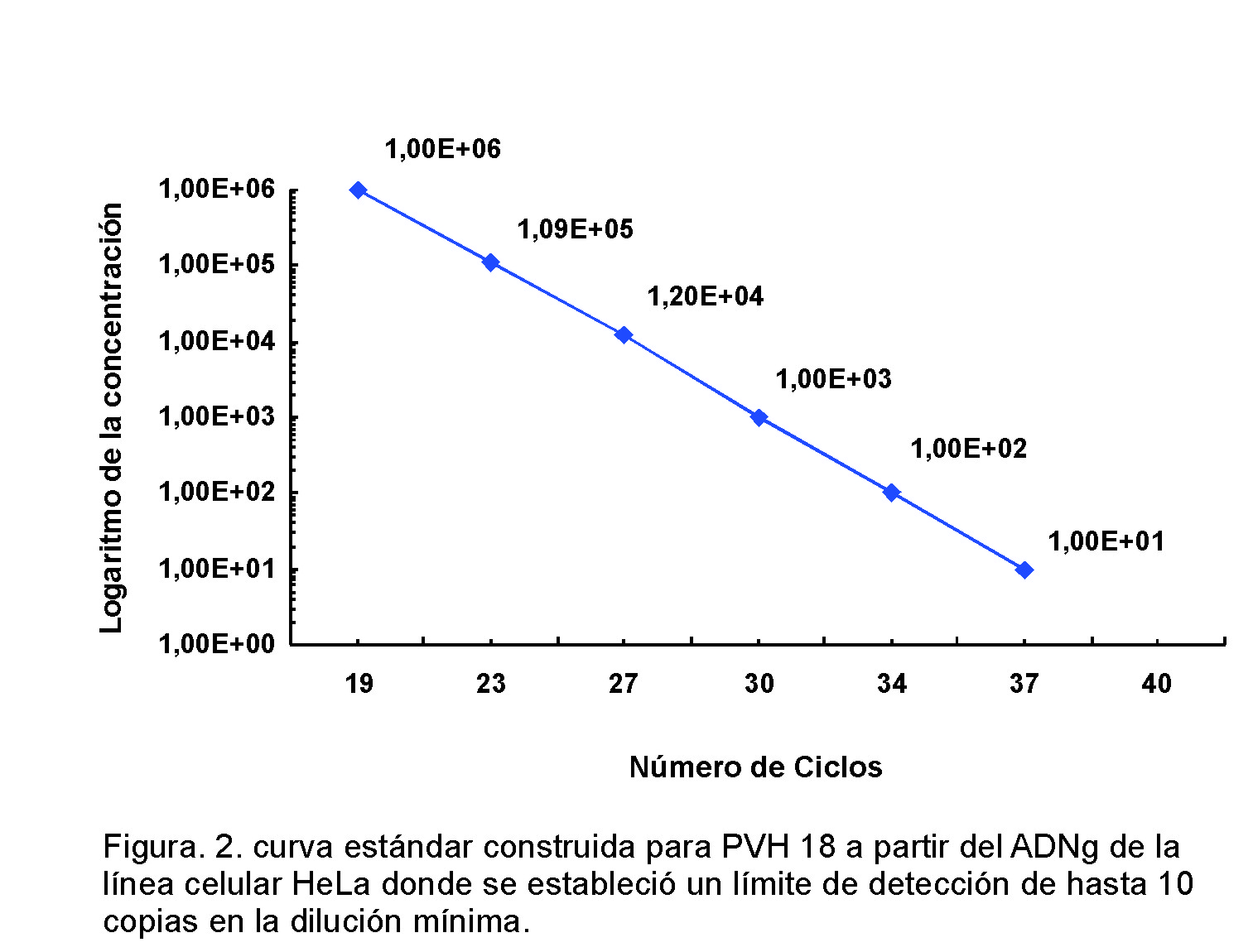
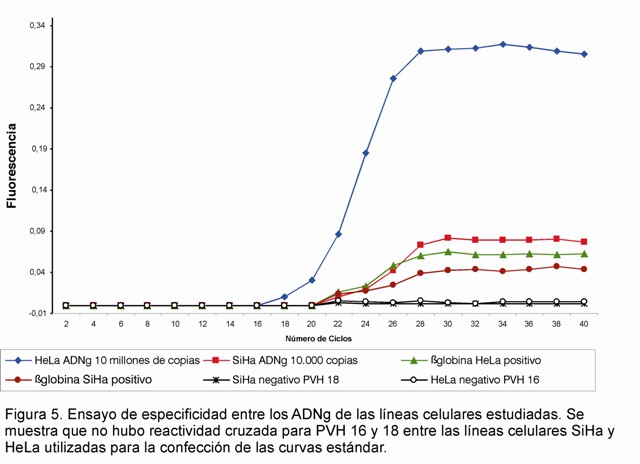
A través del presente estudio se logró obtener dos curvas estándar: para PVH 16, con ADNg de la línea celular SiHa y para PVH 18, con ADNg de la línea celular HeLa. Los resultados de la corrida de los ADNg estándares en la RCP-TR mostraron una buena correlación lineal (r= -0,99) con valores bajos de error a lo largo de 5 y 6 magnitudes de concentración de ADN diana, respectivamente, en cada una de las curvas estándar construidas. Con esto se denota la confiabilidad de los 2 ADN estándar obtenidos y su utilidad para ser empleados en la cuantificación de PVH en diferentes muestras clínicas. Cuando se evaluó el límite inferior de detección del sistema de RCP-TR normalizado se encontró que con las curvas estándar construidas, a partir del ADNg de las líneas celulares SiHa y HeLa, el sistema fue capaz de detectar hasta 10 copias de PVH 16 y PVH 18 en las tres réplicas de esta dilución (Fig. 1 y 2).
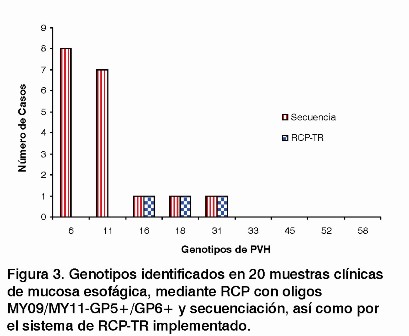
La RCP-TR normalizada probó ser muy específica entre los diferentes genotipos de PVH, al no ocurrir amplificaciones para otros genotipos que no coincidieran con los que se habían identificado mediante RCP cualitativa y secuenciación nucleotídica. Al estudiar las muestras de mucosa esofágica, en ninguno de los casos positivos a PVH 6 y 11 por secuenciación, hubo amplificación con los cebadores empleados en el sistema RCP-TR implementado. Los genotipos de PVH 6 y 11 fueron mayoritarios en estas muestras y el sistema de RCP-TR no contiene cebadores para amplificarlos. Sin embargo, sí se detectan otros genotipos de alto riesgo como el 16, 18 y 31 en este grupo de muestras.
En estos casos se evidenció una total coincidencia entre ambos métodos. A pesar de haberse analizado solo 20 muestras para normalizar el sistema, se debe destacar que las dos muestras en las que se detectaron los genotipos de alto riesgo PVH 16 y 18 estaban diagnosticadas previamente por histología como carcinoma epidermoide de esófago y la muestra en que se detectó PVH 31, como papilomatosis queratinizante.
Además, en las muestras cervicouterinas se mostró una absoluta coincidencia entre los dos métodos de detección de PVH estudiados. En el 100% de las muestras positivas a PVH 16, 18, 31, 33 y 58 por secuenciación, se pudo confirmar el mismo resultado por el método de RCP-TR implementado. La especificidad del sistema también se confirma, pues no hubo detección para los casos positivos a PVH 61 y 85 detectados por secuenciación, ya que no están incluidos en el protocolo normalizado. El genotipo predominante en un mayor número de casos fue PVH 16, seguido por el 33 y el 18. Los genotipos de PVH obtenidos mediante RCP cualitativa y posterior secuenciación, así como por el protocolo normalizado para cada grupo de muestras clínicas, aparecen en las Fig. 3 y 4.
Tampoco hubo reactividad cruzada para PVH 16 y 18 entre las dos líneas celulares empleadas (Fig. 5). Además, su especificidad también se comprobó pues no se detectó un incremento por encima del umbral de fluorescencia en los capilares que contenían ADN extraído de las cepas de CMVh, VHS-2 y VVZ.
DISCUSIÓN
Uno de los pasos fundamentales para lograr la puesta en marcha de la RCP-TR es la obtención de los productos estándares. Hasta el momento no se cuenta con ADN estándares universales que permitan normalizar este sistema. Es por ello que la inmensa mayoría de los ensayos que se han diseñado se normalizan en los laboratorios encargados del diagnóstico, a los que se les reconoce en la literatura como RCP-TR caseros (16). De esta forma, los dos productos obtenidos en el presente estudio representan una importante fuente reproducible de ADN estándar para la puesta en marcha de la RCP-TR en el laboratorio de ITS del IPK, en Cuba.
Al comparar los diferentes parámetros de las curvas estándar construidas en el presente trabajo con los obtenidos por los autores del protocolo original (5) se encontró que los valores relacionados con la eficiencia de la reacción y el coeficiente de regresión alcanzados en este trabajo se encuentran dentro de los valores aceptables, teniendo en cuenta que se modificaron varios marcajes en las sondas y se introdujeron variaciones en la plataforma empleada. Se han publicado diversos métodos de RCP-TR en los que se normaliza el sistema para la detección y cuantificación de diferentes genotipos de PVH (13).
Recientemente se reportó un estudio donde los límites mínimos de detección oscilan desde 44 hasta 700 copias para 15 genotipos de PVH de alto riesgo y 5 genotipos de bajo riesgo oncogénico, con la limitante de que este método solo discrimina en dos grupos de PVH de alto o bajo riesgo (14). En otro estudio para la detección y cuantificación de PVH 6, 11, 16 y 18 sí es posible discriminar entre genotipos, aunque el límite mínimo de detección es de 100 copias (13).
Considerando los buenos resultados obtenidos en el presente estudio con estas curvas estándar para PVH 16 y PVH 18, por permitir detectar un número mínimo de copias de secuencias dianas, 10 copias de ADN para ambos genotipos, se decidió utilizar dichas curvas estándar para cuantificar PVH 16, 18, 31, 33, 45, 52 y 58 en diferentes muestras clínicas de pacientes con sospecha de infección por estos virus.
En el caso de las muestras esofágicas, los resultados obtenidos en este estudio se corresponden con lo que se ha reportado mundialmente, pues los genotipos que predominan en las lesiones papilomatosas con presencia de coilocitos son PVH 6 y 11, de bajo potencial oncogénico.
Se detectaron genotipos de PVH de alto riesgo en lesiones malignas de esófago como es el caso de los carcinomas epidermoides. Dichas lesiones se han visto asociadas frecuentemente con la presencia de genotipos de PVH de alto riesgo oncogénico (17). En el caso de las 20 muestras de cuello uterino con lesiones intraepiteliales de alto grado se corrobora la presencia de PVH de alto riesgo oncogénico, con un predominio de PVH 16. Este genotipo se ha detectado en más del 70% de este tipo de lesiones (18).
El hecho de que no se detectara fluorescencia en los capilares que contenían ADN extraído de las cepas de CMVh, VHS-2 y VVZ justifica lo planteado por los autores que diseñaron las sondas y los cebadores utilizados para normalizar este sistema de RCP-TR, quienes demostraron que estos no poseían reactividad cruzada con el genoma de ningún otro patógeno ni con alguna secuencia de ADN humano (5).
La evidencia acumulada ha demostrado la utilidad de la genotipificación de PVH en el manejo clínico y seguimiento de los pacientes y como un indicador de la eficacia de candidatos vacunales de uso profiláctico. La determinación de la carga viral también se evalúa como un indicador de la progresión de las lesiones en aquellas pacientes con lesiones intraepiteliales cervicales de alto grado que han sido sometidas a algún tipo de tratamiento o en las que se evalúa algún candidato vacunal de uso terapéutico en estudio. En este trabajo se ha normalizado una RCP-TR para genotipificar y cuantificar los 7 tipos de PVH de alto riesgo oncogénico que se han publicado recientemente como los más comúnmente asociados al cáncer cervicouterino en el mundo (PVH tipos 16, 18, 31, 33, 45, 52 y 58).
El sistema normalizado demostró una coincidencia del 100% en los resultados, al ser comparado con los protocolos de RCP cualitativa que se emplean mundialmente. Al comparar los resultados obtenidos al normalizar esta metodología con otros estudios publicados, se puede afirmar que las modificaciones en los marcajes y en el equipamiento utilizado no afecta la sensibilidad y especificidad del sistema. (13, 19).
El uso de los cebadores MY09/MY11 y GP5+/GP6+ con la posterior secuenciación de los productos amplificados coincidió en todas las muestras clínicas analizadas con los resultados obtenidos por la RCP-TR normalizada.
Los cebadores MY y GP para RCP cualitativa, comúnmente se combinan con diversas técnicas de hibridación de los productos amplificados. En estos protocolos las sondas a hibridar se marcan por diferentes técnicas y todo el procedimiento resulta laborioso y relativamente largo, aunque son considerados con un alto nivel de reproducibilidad y elevada sensibilidad, por lo que constituyen sistemas de referencia (13).
Sin embargo, los ensayos de RCP-TR resultan rápidos, sensibles y mucho menos laboriosos, además de que permiten no solo el genotipado, sino también la cuantificación de la carga viral. Un aspecto que limita el uso de estos métodos en su modalidad de ensayo múltiple es el hecho de que no resultan muy eficientes en la detección de coinfecciones con más de tres genotipos, pues los estudios interlaboratorios reflejan estas diferencias. Este inconveniente se soluciona al diseñar ensayos múltiples, donde no se detecten más de cuatro genotipos en una misma mezcla de reacción, como se refiere en el protocolo original en el que se sustenta la presente investigación (20).
La carga viral se ha evaluado como un indicador del desarrollo y evolución de las lesiones de alto grado, sobre todo en aquellos casos sujetos a tratamiento (5). En estudios recientes se ha estimado el valor de la carga viral de PVH 16 con respecto a la evolución histológica y la eliminación de las lesiones de neoplasia intraepitelial cervical de diferente grado. Se ha demostrado que las variaciones de los valores de la carga viral en el tiempo pueden ser un indicador predictivo de la evolución histológica de las lesiones, incluso en pacientes que han sido vacunados con la vacuna tetravalente Gardasil® .
Esta vacuna, a pesar de no tener un diseño para uso terapéutico, ha mostrado efectos en la disminución de la carga viral y en la evolución clínica favorable de las lesiones intraepiteliales de alto y bajo grado (20). Este criterio puede ser evaluado en el diagnóstico y la investigación de los casos clínicos con NIC, no solo para PVH 16 sino también para el resto de los genotipos, de gran relevancia clínica, incluidos en este protocolo. Además, el ensayo puede ser útil para valorar clínicamente la persistencia viral de cada genotipo y así monitorear las lesiones recurrentes e investigar el efecto de la vacunación contra PVH en el futuro.
En el presente estudio se normalizó un sistema de RCP-TR rápido, específico y altamente sensible para la detección y cuantificación del PVH. La incorporación de este sistema al flujograma diagnóstico del Laboratorio de ITS del Departamento de Virología del IPK significará un salto cualitativo no solo para el diagnóstico de la infección por estos virus, sino también en el monitoreo de la respuesta terapéutica, ante la introducción de la vacunación y en estudios sobre la patogenia de la infección por este virus oncogénico.
Agradecimientos
Al doctor Ulrich Hengge, de la Universidad de Düserdolf, Alemania, por la donación del equipo Light Cycler 1.5 al Departamento de Virología del IPK.
REFERENCIAS
1. zur Hausen H. Papillomaviruses in the causation of human cancers a brief historical account. Virology 2009;384(2):260-5.
2. zur Hausen H. Perspectives of contemporary papillomavirus research. Vaccine 2006;24(Suppl 3):S3/iii-S3/iv.
3. Tucker RA, Unger ER, Holloway BP, Swan DC. Real-time PCR-based fluorescent assay for quantitation of human papillomavirus types 6, 11, 16 and 18. Mol Diagn 2001;6(1):39-47.
4. Kreimer AR, Clifford GM, Snijders PJ, Castellsague X, Meijer CJ, Pawlita M, et al. HPV16 semiquantitative viral load and serologic biomarkers in oral and oropharyngeal squamous cell carcinomas. Int J Cancer 2005;115(2):329-32.
5. Schmitz M, Scheungraber C, Herrmann J, Teller K, Gajda M, Runnebaum IB, et al. Quantitative multiplex PCR assay for the detection of the seven clinically most relevant high-risk HPV types. J Clin Virol 2009;44(4):302-7.
6. Jacobs MV, de Roda Husman AM, van den Brule AJ, Snijders PJ, Meijer CJ, Walboomers JM. Group-specific differentiation between high- and low-risk human papillomavirus genotypes by general primer-mediated PCR and two cocktails of oligonucleotide probes. J Clin Microbiol 1995;33(4):901-5.
7. Bauer HM, Greer CE, Manos MM. Determination of genital human papillomavirus infection using consensus PCR. In: Herrington CS, McGee JOD, editors. Diagnostic molecular pathology: a practical approach. Oxford, United Kingdom: Oxford University Press; 1992. p. 132-52.
8. Satra M, Vamvakopoulou DN, Sioutopoulou DO, Kollia P, Kiritsaka A, Sotiriou S, et al. Sequence-based genotyping HPV L1 DNA and RNA transcripts in clinical specimens. Pathol Res Pract. 2009;205(12):863-9.
9. van Ham MA, Bakkers JM, Harbers GK, Quint WG, Massuger LF, Melchers WJ. Comparison of two commercial assays for detection of human papillomavirus (HPV) in cervical scrape specimens: validation of the Roche AMPLICOR HPV test as a means to screen for HPV genotypes associated with a higher risk of cervical disorders. J Clin Microbiol 2005;43(6):2662-7.
10. Espy MJ, Uhl JR, Sloan LM, Buckwalter SP, Jones MF, Vetter EA, et al. Real-time PCR in clinical microbiology: applications for routine laboratory testing. Clin Microbiol Rev 2006;19(1):165-256.
11. Peitsaro P, Johansson B, Syrjanen S. Integrated human papillomavirus type 16 is frequently found in cervical cancer precursors as demonstrated by a novel quantitative real-time PCR technique. J Clin Microbiol 2002;40(3):886-91.
12. Gravitt PE, Peyton C, Wheeler C, Apple R, Higuchi R, Shah KV. Reproducibility of HPV 16 and HPV 18 viral load quantitation using TaqMan real-time PCR assays. J Virol Methods 2003;112(1-2):23-33.
13. Seaman WT, Andrews E, Couch M, Kojic EM, Cu-Uvin S, Palefsky J, et al. Detection and quantitation of HPV in genital and oral tissues and fluids by real time PCR. Virol J 2010;7:194. Disponible en: http://www.virologyj.com/content/7/1/194 .
14. Takacs T, Jeney C, Kovacs L, Mozes J, Benczik M, Sebe A. Molecular beacon-based real-time PCR method for detection of 15 high-risk and 5 low-risk HPV types. J Virol Methods 2008;149(1):153-62.
15. Guo M, Gong Y, Deavers M, Silva EG, Jan YJ, Cogdell DE, et al. Evaluation of a commercialized in situ hybridization assay for detecting human papillomavirus DNA in tissue specimens from patients with cervical intraepithelial neoplasia and cervical carcinoma. J Clin Microbiol 2008;46(1):274-80.
16. Speers DJ. Clinical applications of molecular biology for infectious diseases. Clin Biochem Rev 2006;27(1):39-51.
17. Feller L, Wood NH, Khammissa RA, Lemmer J. Human papillomavirus-mediated carcinogenesis and HPV-associated oral and oropharyngeal squamous cell carcinoma. Part 1: human papillomavirus-mediated carcinogenesis. Head Face Med 2010;6:14. Disponible en: http:// www.ncbi.nlm.nih.gov/pmc/articles/pmc2912877.
18. zur Hausen H. Human papillomavirus & cervical cancer. Indian J Med Res 2009;130(3):209.
19. Gnanamony M, Peedicayil A, Subhashini J, Ram TS, Rajasekar A, Gravitt P, et al. Detection and quantitation of HPV 16 and 18 in plasma of Indian women with cervical cancer. Gynecol Oncol 2010;116(3):447-51.
20. Sabol I, Salakova M, Smahelova J, Pawlita M, Schmitt M, Gasperov NM, et al. Evaluation of different techniques for identification of human papillomavirus types of low prevalence. J Clin Microbiol 2008;46(5):1606-13.
Recibido: Junio de 2011
Aceptado: Agosto de 2011