Introducción
El coronavirus 2 del síndrome respiratorio agudo grave o SARS-CoV-2 (del inglés, severe acute respiratory syndrome coronavirus 2) se reportó primero en 2019 en Wuhan, provincia china de Hubei; es el agente causal de la pandemia de COVID-19 (del inglés coronavirus disease 2019).1
El SARS-CoV-2 es uno de los siete tipos de coronavirus que infectan a los humanos y uno de los tres tipos de coronavirus que causan enfermedades respiratorias.2) Este virus posee cuatro proteínas estructurales que participan en la patogenia de la infección viral.3,4,5
La infección viral comienza principalmente por la unión de los virus a receptores funcionales en la superficie de la célula hospedera.6 Los receptores determinan la invasión y diseminación viral y las manifestaciones clínicas de los pacientes.
La proteína de la espícula (proteína S, proteína espicular; spike protein en inglés) está constituida por dos dominios funcionales: el dominio funcional subunidad 1 ó S1 y el dominio funcional subunidad 2 ó S2. Estos dominios intervienen en la unión del SARS-CoV-2 al receptor de membrana de la célula huésped y de la fusión de las membranas virales y celulares, respectivamente. El receptor del virus es la enzima convertidora de angiotensina-2 (ACE2).5,7
El receptor ACE2 forma parte del sistema renina-angiotensina-aldosterona que participa en la respuesta inflamatoria, presión sanguínea, regulación del volumen vascular y remodelación de tejidos. La unión de SARS-CoV-2 con ACE2 trastorna este sistema con efectos protrombóticos y proinflamatorios.8) La ACE2 se encuentra en la membrana celular de numerosos tejidos, lo que explica la gran variedad de síntomas que provoca la COVID-19.6,9
El propósito de esta revisión bibliográfica fue describir la relación entre la proteína de la espícula del SARS-CoV-2 y su receptor (ACE2) como desencadenante primario de la infección por la COVID-19.
Método
Se desarrolló una revisión bibliográfica basada en análisis de documentos y contrastación de la información científica sobre la temática, en bases de datos disponibles en Google Académico (https://scholar.google.com.cu), SciELO (https://scielo.org/es), PubMed (https://pubmed.ncbi.nlm.nih.gov) y en el DeCS (https://decs.bvsalud.org/es).
Los descriptores seleccionados fueron: enfermedad por coronavirus 2019, COVID-19, SARS-CoV-2, glicoproteína espicular del coronavirus, enzima convertidora de angiotensina 2, vacuna contra la COVID-19. Sus equivalentes en inglés: coronavirus disease 2019, COVID-19, SARS-CoV-2, spike glycoprotein, angiotensin-converting enzyme 2, COVID-19 vaccines. Se usaron operadores booleanos: “AND”, “OR” y “NOT” para buscar los descriptores del tema. El periodo de publicación seleccionado fue 2019-2021, sin restricciones en cuanto al tipo de artículos (originales, revisiones bibliográficas, revisiones sistemáticas, metaanálisis, ensayos clínicos, libros y documentos diversos); disponibles en idioma español e inglés a texto completo.
Se realizó un filtro de artículos dividido en tres partes: la primera en revisar los títulos de las publicaciones arrojadas por las bases de datos que debían tener al menos una de las palabras clave. La segunda parte correspondía a la lectura del resumen a fin de comprobar que el artículo académico contenía la información requerida. La tercera parte consistía en la lectura completa de los artículos que pasaron la segunda parte del filtro para verificar que la información era útil.
Desarrollo
Virus del SARS-CoV-2
El SARS-CoV-2 semeja una corona, de ahí, su nombre; es un virus envuelto con un genoma de ARN de 29 881 ribonucleótidos que codifica para 9 860 aminoácidos.5,10,11
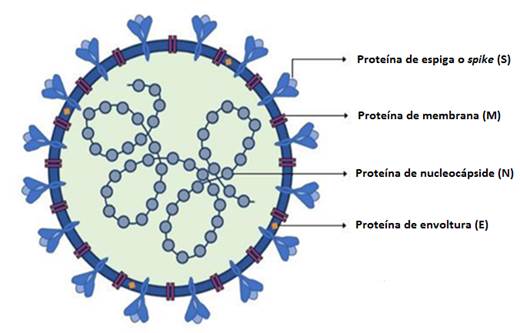
Los genes más importantes son estructurales: S (de spike o pico o espiga o espícula), E (envoltura), M (membrana) y N (nucleocápside).5,12) Como si fuese un erizo marino tiene muchas espinas (proteína espicular) que salen de su cuerpo; cada espina está formada por tres subunidades u homotrímero de dicha proteína espicular. El genoma viral codifica también, al menos, 16 proteínas no estructurales.13 En la Figura 1 aparece una representación esquemática del coronavirus.
La proteína de la espícula es la más grande de las proteínas estructurales y presenta el 76 % de homología de secuencia de aminoácidos con SARS-CoV, lo que sugiere que interactúa con similares proteínas diana.14
En el caso de SARS-CoV-2, esta proteína con 1 273 aminoácidos, permite a la membrana viral fusionarse con la membrana de la célula del huésped. Por su importancia en la infección viral sus características se desarrollarán más adelante.
La proteína N forma la nucleocápside helicoidal, uniéndose a lo largo de todo el genoma viral, que adopta una forma de rizo. Esta proteína pesa de 43-50 KDa y está fosforilada en un número discreto de serinas y treoninas. Aunque no se ha determinado el papel de esta fosforilación, se sugiere que está relacionado con funciones reguladoras y con su capacidad para unirse al genoma viral.13
La más abundante proteína, M, proporciona soporte estructural y la proteína E es una pequeña proteína de membrana esencial en el ensamblaje y liberación de los viriones.14
Enzima convertidora de angiotensina-2
La ACE2 forma parte del sistema renina-angiotensina-aldosterona cuyo funcionamiento interviene en la regulación de la función cardiovascular y renal.9,15 La ACE2 se descubrió como una homóloga de ACE en 2000.6 La ACE2 es una proteína transmembranal tipo I que presenta 805 aminoácidos con un dominio N terminal extracelular y un tallo C terminal corto intracelular.6
El extremo N terminal presenta un dominio de metaloproteinasa Zinc (HEMGH dominio), con un 41,8 % de homología de secuencia con el dominio amino de ACE.6 Este dominio es esencial para la formación del péptido vasodilatador ang (1e7) y la fijación de la proteína de la espícula del SARS-CoV-2.6
Como carboxipeptidasa, la ACE2 escinde muchos sustratos biológicos, además de la angiotensina II, para controlar la permeabilidad vascular.16) Es una metaloproteinasa homóloga de la enzima convertidora se angiotensina (ACE), pero con diferente especificidad de sustrato.6,16
La ACE escinde a la angiotensina I (ang I) para producir el potente octapéptido vasopresor angiotensina II (ang II) que, a su vez, es escindido por la ECA2 para producir ang 1-7.17) Juntas, la ACE y la ACE2 regulan la vasoconstricción y la vasodilatación en el sistema renina-angiotensina aldosterona. Además, la ACE y la ACE2 regulan el sistema cinina-calicreína para controlar la permeabilidad vascular.17
Además de los sistemas renina-angiotensina y cinina-calicreína, la ACE2 también escinde otros péptidos biológicos, como apelina-13, que activa el receptor de apelina para causar vasodilatación.18 A pesar de la importancia de la ACE2 en el sistema, hay un conocimiento limitado del impacto de la infección por coronavirus en la función fisiológica de la ACE2.
Proteína de la espícula
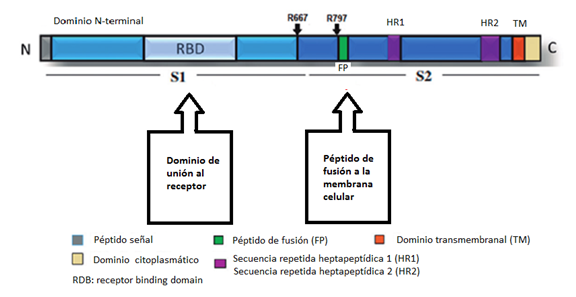
La proteína de la espícula es una glicoproteína estructural localizada en la superficie del virión del SARS-CoV-2.2,5,19) Es una proteína transmembranal de tipo I3,4,14,20,21) homotrimétrica, es decir, que está compuesta de tres partes o subunidades similares18,19,20,22) las cuales corresponden a tres dominios, de los mismos dos son funcionales S1 y S2 y un dominio transmembranal o segmento intraviral.
Esta proteína consta de un péptido señal (aminoácidos 1-13) situado en el N-terminal, la subunidad S1 (14-685 residuos) y la subunidad S2 (686-1273 residuos); las dos últimas regiones son responsables de la unión al receptor y de la fusión de la membrana, respectivamente.21) Las subunidades S1 y S2 forman la cabeza bulbosa y la región del tallo.
En la subunidad S1 hay un dominio N-terminal (14-305 residuos) y un dominio de unión al receptor (RBD, por sus siglas en inglés, receptor-binding domain, 319-541 residuos).21 La subunidad S2 comprende: el péptido de fusión (FP) (788-806 residuos), la secuencia repetida heptapeptídica 1 (HR1) (912-984 residuos), HR2 (1163-1213 residuos), el dominio transmembranal o TM (del inglés transmembrane, 1213-1237 residuos) anclado en la membrana viral y el dominio citoplasmático (1237-1273 residuos).
La importancia de la proteína espicular radica en el dominio RBD que es el sitio de unión del coronavirus a los receptores de ACE2, para su entrada a la célula huésped y el comienzo de la infección viral.19,21,22,23) En la Figura 2 aparece una representación esquemática de la proteína de la espícula del coronavirus.
Unión de la proteína de la espícula a ACE2
El SARS-CoV-2 utiliza la ACE2 como medio para entrar a la célula donde la ACE2 cumple el papel de receptor para la proteína de la espícula.24,25,26) Esta proteína gracias a RBD del dominio S1, aumenta la actividad proteolítica y mejora la función enzimática de ACE2.6,26) Entonces, el camino para la invasión viral comienza con la adherencia de la proteína de la espícula al receptor de la célula al que tiene mucha afinidad, ACE2.
Después se produce la escisión proteolítica de ambos dominios funcionales, es decir, de S1 y S2 gracias a la acción de la furina y la proteasa transmembranal de serina tipo2 (TMPRSS2) y la ACE2.5,6,9) Este proceso sucede específicamente en el sitio de la proteasa S1/S2 que por motivos de ubicación se refiere a los límites entre ambos dominios. Esto permite que la proteína de la espícula esté apta para invadir la célula.15,19
Por último, se produce la fusión de las membranas del virus y de la célula diana, gracias a la previa escisión y capacitación que expone el extremo amino-terminal o N-terminal de la subunidad S1 ya que es el segmento específico de fijación a la célula huésped, así como de la subunidad S2 es el extremo carboxilo-terminal o C-terminal que se adhiere a la membrana para así permitir el paso del ARN viral.5,9,19,24
Por lo tanto, el papel de la proteína ACE2 es fundamental para que se inicie el proceso de infección y este depende fundamentalmente de la acción receptora de esta proteína que funciona como un complemento perfecto.24,25) Es decir, la ACE2 potencia la actividad infecciosa del virus cuando el dominio RBD de S1 se une a la misma y la proteína de la espícula mejora la función enzimática de ACE2.16) Esta complementariedad influye en la patogénesis de los distintos síndromes, como el síndrome de insuficiencia orgánica múltiple.9,26
Consecuencias clínicas de la unión de la proteína de la espícula a ACE2
Las consecuencias de la unión de la proteína de la espícula del SARS-CoV-2 con la ACE2 dependerán de la ubicación del receptor.5,27
En las membranas del epitelio de las células respiratorias puede producirse edema alveolar.5,12) El receptor ACE2 se expresa principalmente en las células alveolares de tipo 2 (producen el surfactante pulmonar) desencadenando el síndrome respiratorio agudo grave (SRAS).10
También se presentan manifestaciones gastrointestinales como, por ejemplo: diarreas, debido a que el tracto gastrointestinal también posee receptores de ACE2. La expresión de esta enzima suele ser mayor en los enterocitos y el SARS-CoV-2 interrumpe su funcionamiento.28
En relación a las complicaciones neurológicas se destaca la encefalitis por la presencia del SARS-CoV-2 en el líquido cefalorraquídeo, como también ictus isquémicos y crisis epilépticas.29,30,31
Las lesiones cardiacas del SARS-CoV-2 se deben a tres mecanismos básicos: daño cardiaco indirecto por la exagerada respuesta inflamatoria inmune, un daño directo por la invasión de cardiomiocitos y una hipoxia severa por un daño respiratorio agudo por una mayor demanda de oxígeno.32,33) Las complicaciones cardiacas principales son la miocarditis, la insuficiencia cardíaca descompensada, el síndrome coronario agudo, las arritmias y los eventos tromboembólicos.32,33
En pacientes hipertensos hay mayor cantidad de ACE2 y, por tanto, pueden presentar respuestas amplificadas ante la infección.32 La complicación principal es la insuficiencia respiratoria progresiva que es la causa más importante de muerte en pacientes >60 años.30
Las complicaciones hematológicas se deben a un estado de hipercoagulación. Si se presenta una coagulopatía y la trombocitopenia se incrementa la hemorragia, la trombosis venosa profunda y el tromboembolismo pulmonar.30
Finalmente, las complicaciones renales que se desencadenan principalmente por la disfunción múltiple de órganos es la insuficiencia renal aguda.30
Terapia anti-COVID-19 contra proteína de la espícula
El rol fundamental de la proteína espicular del SARS-CoV-2 en la infección viral indica que es una diana potencial para el empleo de medicamentos y vacunas contra la COVID-19.21,34,35
Entre los inhibidores desarrollados para impedir la fusión de la membrana mediada por la proteína de la espícula se encuentra EK1C4, cuya aplicación nasal protege a los ratones de las infecciones por coronavirus.17) CA1 y CB6 son anticuerpos contra RBD capaces de neutralizar in vitro la infección por SARS-CoV-2, además, CB6 reduce los títulos virales y el daño pulmonar cuando se administra a monos Rhesus.
Las vacunas desarrolladas contra la COVID-19 en el mundo son de cinco tipos. Las principales emplean la proteína de la espícula o alguno de sus componentes.36,37,38,39
Los candidatos vacunales BNT162b2 de Pfizer/BioNTech y mRNA-1273 de Moderna se basan en un ARNm estabilizado que codifica la prefusión del SARS-CoV-2 y que puede producirse después de que el ARNm se introduce y se traduce en la célula humana.3,36
La estrategia de la vacuna de Oxford (AstraZeneca®) introduce un ADN vector de un adenovirus no patógeno (chimpancé no replicable).5,22 Las otras vacunas mencionadas introducen el ARNm que codifica para la proteína de la espícula envuelto en un vehículo llamado nanopartícula.5 La idea es que las células humanas sinteticen esta proteína extraña para generar respuesta de anticuerpos y de células T para atacar al intruso.
La Ad26.COV2.S de Johnson & Johnson, se basa en el adenovirus 26 no replicable y la Gam-COVID-Vac (Sputnik V) del Instituto Gamaleya, expresan la proteína de la espícula del SARS-CoV-2.22 NVX-CoV2373 (Novavax), una proteína recombinante, es también la proteína de la espícula completa del SARS-CoV-2.
Las vacunas cubanas contienen una parte de la proteína de la espícula utilizada por el virus para unirse a las células humanas, lo que genera anticuerpos neutralizantes que bloquean este proceso de unión.37)
Soberana 02, Soberana Plus y Abdala se aprobaron como vacunas de emergencia.38 Las dos primeras se componen de subunidad proteica de RDB de la proteína de la espícula de SARS-CoV-2, conjugada con toxoide tetánico y la Abdala es una sub-unidad proteica con hidróxido de aluminio como coadyuvante.38
Para ampliar sobre el diseño y aplicación de vacunas contra la COVID-19 se encuentran los trabajos de Chung, et al.34; Dai y Gao,39; Sharma, et al.40; Jia y Gong35 y Sharma, et al.38
Este aspecto del diseño y aplicación de vacunas anti-COVID-19 se encuentra en desarrollo.
Consideraciones finales
La proteína de la espícula del SARS-CoV-2, que desempeña un papel clave en el reconocimiento del receptor y en el proceso de fusión de la membrana celular, está compuesta por dos sub-unidades, S1 y S2. La sub-unidad S1 contiene un dominio de unión al receptor que se une al receptor del huésped: la ECA2, mientras que la subunidad S2 interviene en la fusión de la membrana celular del virus.
El conocimiento de la relación entre el SARS-CoV-2 y su receptor ECA2 permite no solo conocer la fisiopatología de la COVID-19, sino el diseño de fármacos antivirales y vacunas que contribuyen a la prevención y tratamiento de esta enfermedad viral.