My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Fitosanidad
Print version ISSN 1562-3009On-line version ISSN 1818-1686
Fitosanidad vol.13 no.3 Ciudad de la Habana Jul.-Sept. 2009
Ecología
Variabilidad genética de las poblaciones de Fusarium oxysporum f. sp. cubense en bananos y plátanos de Cuba
Genetic Variability of Fusarium oxysporum f. sp. cubense populations in Cuban Plantains and Bananas
Alicia Batlle Viera y Luis Pérez Vicente
Instituto de Investigaciones de Sanidad Vegetal. Calle 110 no. 514 e/ 5.a B y 5.a F, Playa, Ciudad de La Habana, CP 11600, lperezvicente@sanidadvegetal.cu; lperezvicente@live.com
RESUMEN
El mal de Panamá _causado por Fusarium oxysporum f. sp. cubense (Foc)_ es una de las enfermedades más nocivas que atacan las musáceas. La raza 1 causó una epidemia que destruyó la industria de exportación bananera basada en el clon Gros Michel en América, así como la desaparición del cultivo comercial del clon Manzano en Cuba. Con el fin de determinar la variabilidad genética de las poblaciones en el país, se realizaron 52 aislamientos monospóricos del hongo de clones naturalmente infectados en 26 localidades de ocho provincias de Cuba. Se determinó la morfología de las colonias en PDA y medio K2, se caracterizaron los compuestos volátiles producidos en un medio a base de arroz, se generaron mutantes nit auxotróficos y se determinaron los grupos de compatibilidad vegetativa con una colección internacional de nit M pertenecientes a todos los GCV de Foc. Se determinó la relación genética entre ellos mediante análisis de AFLP. En medio K2 no se observaron colonias lacinadas en ninguno de los aislamientos pertenecientes a las razas 1 y 2; se observó la producción de compuestos volátiles con independencia de las razas a que pertenecían los aislamientos. Las poblaciones deFoc de Cuba pertenecen a los GCV 01210, 0124, 0124/0125 y 0128. Los aislamientos con mayor similitud genética estuvieron en todos los casos agrupados en el mismo GCV y los aislamientos del GCV 0124, se encontraron distribuidos en todos los grupos genéticos por lo que posiblemente de este se diferenciaron los genotipos pertenecientes los GCV 0124/0125 y 0128.
Palabras claves: Fusarium oxysporum f. sp. cubense, marchitez por Fusarium, mal de Panamá, grupos de compatibilidad vegetativa, AFLP
ABSTRACT
Panama disease caused by Fusarium oxysporum f. sp. cubense (Foc), is one of the most destructive diseases of Musa. The epidemic of race 1 was the cause of banana export industry destruction which was based on Gros Michel in América and its substitution by Cavendish cultivars as well as the commercial cultivation of Manzano cv. in Cuba. In order to determine genetic variability of fungi populations in the country, fifty two monosporic isolates from naturally infected plants from 26 sites in eight provinces of Cuba were obtained. There were determined the colonies morphology in PDA and K2 media, the volatiles compounds produced on a rice based media were identified. Auxotrofic nit mutants were generated to determine the vegetative compatibility groups of each isolate with an international Nit M collection of all VCG of Foc. The genetic relationship between isolates and VCGs were determined by AFLP analysis. In K2 media were not observed lacinated colonies in any of the isolates belonging to race 1 and 2 and volatile compounds were or not were produced in different isolates with independence of the race that they belong. In Cuba the Foc populationswere grouped in the VCGs 01210, 0124, 0124/0125 and 0128. The isolates with a higher genetic similarity were in all the cases grouped in the same VCG and in all the genetic groups were present isolates belonging to the VCG 0124 as well, indicating that it could be the original VCG from which the GCV 0124/0125 and 0128 evolved.
Key words: Fusarium oxysporum f. sp. cubense, fusarium wilt, Panamá disease, vegetative compatibility groups, AFLP
INTRODUCCIÓN
La marchitez por Fusarium o mal de Panamá causado por Fusarium oxysporum f. sp. cubense (Foc) es a nivel mundial una de las enfermedades más importantes, ampliamente distribuida y de mayor impacto de los bananos y plátanos.
El patógeno tiene su origen en el sudeste asiático, aunque la enfermedad fue reconocida por primera vez en otros lugares, y ha coevolucionado junto a las musáceas en su centro de origen [Bentley et al., 1998]. Se calculan en más de 80 000 ha de cultivo del clon Gros Michel destruidas por la raza 1 entre 1890 y mediados de la década de los cincuenta [Stover, 1962b], lo que determinó su cambio por clones del subgrupo Cavendish (AAA). Los clones Cavendish se afectaban solo en los subtrópicos, pero la aparición más recientemente de la raza 4 tropical, grupo de compatibilidad vegetativa (GCV 01213-01216) ha causado importantes pérdidas en plantaciones de Malasia e Indonesia [Masdek et al., 2003; Nasdir, 2003]. Su entrada eventual a las plantaciones de Cavendish de América tendría un gran impacto económico y social.
El primer informe oficial de la enfermedad en Cuba fue el de Smith (1910). Johnston (1915) informó que el clon Gros Michel (AAA) y el Manzano (subgrupo Silk, AAB) se encontraban severamente afectados ya en 1910, aunque hay antecedentes de incidencia de la enfermedad en el clon Manzano (subgrupo Silk, AAB) desde finales del siglo XIX. A partir de la sustitución del Gros Michel por clones del subgrupo Cavendish y el cultivo masivo de clones de plátanos AAB, la marchitez por Foc perdió su importancia económica en Cuba, y quedó confinada a las pequeñas parcelas de agricultores y jardines de viviendas, donde Foc se mantenía sobre plantas de los clones Burro Criollo (Bluggoe, ABB) y Manzano.
La aparición de la Sigatoka negra en Cuba a finales de 1990 [Vidal, 1992] tuvo un impacto marcado en los costos de producción, pero especialmente en la estructura clonal de la superficie del país plantada de musáceas [Pérez et al., 2002]. Los bananos Cavendish (AAA) se sustituyeron por los clones tetraploides híbridos de la FHIA (FHIA 23, FHIA 18, FHIA 3), que ocupan alrededor de 11 000 ha. En la actualidad se cultivan a gran escala el clon Burro CEMSA (ABB) que ocupa unas 63 000 ha, y el FHIA 3 (AABB), y se ha popularizado el cultivo del Burro Vietnamita (Pisang awak, ABB) debido a su sabor asemejante al del Manzano. En estos clones se ha incrementado la presencia de la enfermedad.
Foc está considerado un patógeno altamente complejo, y se han utilizado numerosos métodos para caracterizarlo. La variabilidad de las poblaciones, las relaciones filogenéticas y su evolución se han determinado en función de la patogenicidad [Stover y Waite, 1960; Stover, 1962b; Su et al., 1977, 1986; Stover y Buddenhagen, 1986], grupos de compatibilidad vegetativa (GCV) [Leslie, 1990, 1993; Ploetz, 1990; Pegg et al., 1993; Ploetz y Pegg, 1999], número de cromosomas y cantidad de ADN total [Boehm et al., 1993], producción de volátiles [Stover, 1962a, b; Moore et al., 1991], los cariotipos electroforéticos [Boehm et al., 1993; O'Donnell et al., 1997] y el polimorfismo de los fragmentos de restricción [Bentley y Bassam, 1996; Koenig et al., 1997; Bentley et al., 1998].
La formación de heterocariones es una vía mediante la cual hongos normalmente haploides pueden tener el beneficio de una diploidía funcional, como son la complementación y la heterosis [Leslie, 1993]. A los hongos capaces de formar tales heterocariones se les denomina vegetativamente compatibles. La compatibilidad vegetativa ha sido utilizada para identificar subpoblaciones genéticamente aisladas de especies de patógenos fungosos [Puhalla, 1985; Cove, 1976; Correll et al., 1987]. La compatibilidad vegetativa en muchos hongos requiere que los alelos de al menos 10 locus (vic) diferentes sean idénticos [Puhalla y Spieth, 1985], por lo cual los miembros de un GCV constituyen subpoblaciones clonales que están estrechamente relacionadas (nunca se ha observado compatibilidad vegetativa entre líneas de diferentes formae specialis). Al mismo tiempo, dos individuos vegetativamente compatibles deben ser idénticos para genes responsables de la patogenicidad, la adaptación ecológica y otras características que afectan su papel como patógenos del banano. Debido a esta asociación los GCV en cada localidad son indicadores fuertes del comportamiento patogénico, y una herramienta poderosa en el estudio de la biología de las poblaciones y la genética, muy especialmente en el caso de Foc-Musa, donde el análisis genético de la patogenicidad es prácticamente imposible. Se han clasificado al menos veinte GCV o complejos de GCV en Foc [Ploetz y Correll, 1988; Ploetz, 1990; Brake et al., 1990; Moore et al., 1993; Pegg et al., 1993; Bentley et al., 1998].
El AFLP (polimorfismo de los fragmentos de restricción amplificados) es una poderosa técnica de marcaje molecular basada en la detección de fragmentos de restricción del ADN por amplificación mediante PCR, la cual puede utilizarse en ADN de cualquier origen y complejidad [Vos et al., 1995]. Andel-Satar et al. (2003) utilizaron esta técnica para estudiar las relaciones genéticas dentro y entre poblaciones naturales de cinco Fusarium spp. Por su parte, O'Neill et al. (2003) utilizaron AFLP para el estudio de los niveles de variación genómica dentro de especies como Colletotrichum y Fusarium.
El objetivo del presente estudio fue determinar la variabilidad fenotípica, fisiológica, genética y patogénica de las poblaciones de Foc presentes en Cuba.
MATERIALES Y MÉTODOS
Para la colecta de los aislados se realizaron muestreos de plantas con síntomas de marchitez por Foc en diferentes localidades del país, los cuales aparecen en la Tabla 1 y en la Fig. 2. Se tomaron fragmentos de tejidos afectados del pseudotallo con síntomas, y se desinfectaron con hipoclorito de sodio al 2% durante 3 min; luego se sembraron fragmentos de 1-2 mm en aguaagar + 50 ìg/mL de sulfato de estreptomicina. El micelio obtenido se transfirió a placas de Petri con agar-papa dextrosa (PDA), y se incubó a 25°C en la oscuridad durante cinco días. Se obtuvieron cinco cultivos monospóricos de cada aislado según el procedimiento descrito por Booth (1977), y después de incubarlos de cuatro a siete días se procedió a conservarlos en papel de filtro a 4°C, de acuerdo con el método descrito por Correll et al. (1986).
Se sembraron cinco réplicas de discos de micelio de 5 mm de diámetro de cada uno de los aislados en estudio cultivados en PDA y en medio Komada modificado (K2) [Sun et al., 1978]. Se caracterizó la morfología de las colonias y la formación de pigmentos en PDA después de 14 días de incubación a 27°C, y se determinó la presencia o no de bordes laciniados de las colonias en el medio K2.
Para determinar la producción de compuestos volátiles se prepararon erlenmeyers de 250 mL con 30 mL de arroz pulido y 90 mL de agua destilada, y se esterilizaron a temperatura de 128ºC durante 1 h por dos días consecutivos. Estos erlenmeyers se inocularon con discos de 5 mm de diámetro de micelio de cada aislado en estudio, y se incubaron a 27°C durante 14 días bajo la acción de tres lámparas de 40 W de luz fluorescente a 40 cm de altura, según método descrito por Moore et al. (1991). Posteriormente se determinó la presencia de compuestos volátiles en el espacio superior del cultivo de arroz, para lo cual se tomó 1 mL de estos gases y se inyectó en un cromatógrafo gaseoso, equipado con detector de llama y columna capilar de 15 m y 0,53 mm de diámetro interno, recubierta con DB-5, a una temperatura del horno de 40°C y temperatura del inyector de 110°C. Para expresar la composición química de los gases presentes se acopló al cromatógrafo de gases un espectrómetro de masa GC-MS, TRIO 1000.
Para determinar el crecimiento de las colonias a diferentes temperaturas se seleccionaron siete aislados pertenecientes a la raza 1 (GCV 01210) y cinco aislados de la raza 2 (GCV 0124 y 0124/0125). Se sembraron discos de 5 mm de micelio en el centro de placas de Petri de 10 cm con PDA, y se incubaron en una batería de incubadoras a las temperaturas de 8, 10,
12, 15, 20, 25, 28, 30, 35 y 38°C, hasta que la placa quedara cubierta por el crecimiento radial de la colonia en la temperatura más favorable. Se utilizaron cuatro repeticiones por temperatura. Se midió el diámetro de todas las colonias y se ajustaron curvas de regresión cuadráticas del crecimiento en función de la temperatura con el paquete estadístico Statistica 5.0 para Windows. Se compararon las medias del crecimiento de las colonias pertenecientes a cada una de las razas a la temperatura de 27°C mediante el test de Neumann-Keuls.
A cada aislamiento de la colección se le determinó laraza a la que pertenecía por inoculación en los clones diferenciales descritos por Stover (1962b) mediante aplicación de una suspensión de micelio y esporas en bolsas con plantas de cultivo de tejidos sanas, o se estableció putativamente si se obtuvo en el campo en los clones diferenciales Gros Michel, Manzano o Burro Criollo (Bluggoe). Se determinó a cuál grupo de compatibilidad vegetativa pertenecía cada uno de los aislamientos obtenidos, según el procedimiento tratado por Puhalla (1985) y Correll et al. (1987), para lo cual se desarrollaron mutantes auxotróficos. Con este fin se inocularon placas Petri de 10 cm con medio mínimo más el 15% de clorato de potasio, y se incubaron a temperatura ambiente hasta el surgimiento de sectores de crecimiento micelial diferenciado, los que fueron aislados en tubos con cuñas de agar con medio mínimo. A continuación se realizaron las pruebas fisiológicas para determinar los fenotipos nit 1, nit 3 y Nit M (deficientes en las enzimas nitrato reductasa, nitrito reductasa o el cofactor molibdeno, respectivamente), de acuerdo con el procedimiento utilizado por Correll et al. (1987). Se realizaron ensayos de autocompatibilidad por el apareo de los mutantes nit 1 y Nit M obtenidos de cada aislamiento entre sí. Se descartaron los que fueron autoincompatibles, y se asumió que pertenecían a fenotipos crn que no formarían heterocariones al cruzarse con los probadores Nit M de la colección internacional. Se determinó a cuál GCV pertenecía cada aislamiento de la colección, y para ello se sometieron a prueba de compatibilidad los mutantes nit 1 obtenidos de cada aislamiento, con una colección de mutantes Nit M pertenecientes a los GCV 0120, 0121, 0122, 0123, 0124, 0124/0125, 0128, 0129 01210, 01212, 01213, 01214 y 01215 donados por los doctores Ken Pegg, del Queensland Department of Primary Industries, Australia; Suzy Bentley, del Cooperative Research Center for Tropical Plant Pathology in Queensland, Australia; y Julio Hernández, del INIA en Islas Canarias. La existencia de compatibilidad fue determinada por la formación de heterocariones puestos en evidencia por el crecimiento micelial denso en la zona de unión de las colonias enfrentadas (Fig. 1).

Para proceder a la determinación de la variabilidad genética y las relaciones filogenéticas entre los aislamientos se realizó un ensayo de AFLP. A fin de obtener suficiente micelio para las extracciones de ADN, los aislamientos de Foc se cultivaron en erlenmeyers de 250 mL con 100 mL de caldo de papa-dextrosa durante cinco días a una temperatura de 27-30°C. Transcurrido el tiempo de incubación, el contenido de cada erlenmeyer se filtró a través de papel de filtro y se lavó con agua destilada estéril para eliminar restos del medio y se conservó en placas de Petri de 10 cm en un frízer a _20°C.
Se realizó extracción de ADN de 40 aislados de Foc, para lo cual se maceraron 0,2 g de material fresco y se desarrolló el protocolo descrito por Möller y Bahnweg (1992). La concentración de ADN fue determinada mediante un espectrofotómetro con una cubeta de 5 μL.
Para la preparación de los adaptadores se utilizaron 3,58 μL de Eco adaptador superior (0,77 ìg/μL); 2,98 μL de Eco adaptador inferior (0,98 ìg/μL); 48,14 μL de Mse adaptador superior (0,54 ìg/μL); 48,4 μL de Mse adaptador inferior (0,47 ìg/μL). Se incubaron a 65°C durante 10 min y se colocaron a temperatura ambiente para que se enfriaran lentamente y garantizar la unión de los oligonucleótidos.
Para la digestión del ADN se utilizó un volumen de reacción de 50 μL y se colocaron 100 ng/μL de ADN, 5 μL de Buffer 2; 0,2 μL de BSA; 5 u de EcoR I y 5 u de Mse I. Los buffers utilizados fueron: a) buffer t4 ligasa (Tris HCl pH 7,5; 250 mM; MgCl2, 100 mM; DTT 50 mM; espermidina 5 mM); b) buffer TaqPol (Tris HCL pH 8,3; 100 mM; MgCl2 15 mM; KCl 500 mM); c) buffer de carga (formamida 98%, EDTA pH 8, 10 mM; trazas de bromofenol azul y xileno). Esta mezcla se incubó durante 2 h a 37°C. Se preparó en tubo aparte una mezcla de los adaptadores que contenía 1 μL de adaptador de EcoRI; 1 μL de adaptador de Mse I; 1 μL de buffer ligasa, con 10 mM de ATP; y 1 u de T4 ADN ligasa. Se llevó a 10 μL con agua Milli-Q y se añadió a cada tubo de la digestión. Se incubó a 16°C durante toda la noche y se realizó una dilución de 1/10 de cada tubo.
Preamplificación: Se tomaron 5 μL del ADN diluido del paso anterior y en un volumen final de 50 μL se añadió 1 μL de cebador Eco + A (100 ng/μL); 1 μL de cebador Mse + C (100 ng/μL); 2 μL de dNTP; 5 μL buffer TaqPol; 2 μL MgCl2; 1 u de TaqPol, y se utilizó un equipo termociclador para PCR con un programa de 20 ciclos de: a) 30 s a 94°C; b) 60 s a 56°C; c) 60 s a 72°C.
Amplificación: Para la amplificación se utilizaron 5 μL de ADN preamplificado, y se añadió en un volumen final de 20 μL; cebador de E-NNN 0,2 μL; cebador MNNN 4,5 μL; buffer de TaqPol 2,0 μL; MgCl2 0,8 μL; TaqPol 1 u. Las combinaciones de los cebadores utilizadas fueron: a) E-AAC/M-CAA; b) E-AAC/M-CAG; c) E-AGG/M-CTC. Se utilizó el mismo equipo de PCR. La descripción del programa utilizado para la amplificación fue: ciclo 1) 30 s a 94°C, 30 s a 65°C, 60 s a 72°C; ciclo 2_13) similar al ciclo 1, pero la temperatura de hibridación disminuyó 0,7°C en cada ciclo durante 12 ciclos; ciclo 14_36) 30 s a 94°C; 30 s a 56°C; 60 s a 72°C.
Corridas electroforéticas. Las corridas electroforéticas se realizaron en gel de poliacrilamida, para lo cual a las muestras se les añadieron 10 μL de buffer de corrida, se desnaturalizaron durante 3 min a 95°C y se colocaron rápidamente en hielo. Se aplicaron 7 μL en cada pocito del gel. Los geles de poliacrilamida para las corridas electroforéticas se prepararon según el protocolo de Promega (1998). A través de un espectrofotómetro se determinaron las densidades ópticas de las muestras, y con ellas la concentración de cada una. Se realizó la digestión del DNA con las enzimas Taq I y Ase I.
Análisis de datos. Se consideraron solamente los marcadores polimórficos, los cuales se registraron manualmente en una matriz de datos binarios (con la presencia como 1 y la ausencia como 0). A partir de las matrices de datos originales se calculó la similitud genética (SGij) entre cada par de aislados, para lo que se empleó el índice de Jaccard (1908):

donde:
a: Presencia de la banda en los genotipos i y j
b: Presencia de la banda en el genotipo i y ausencia en j
c: Ausencia de la banda en el genotipo i y presencia en j
Los agrupamientos de los genotipos se efectuaron sobre la base de los valores de disimilitud (1-SGij) entre todos los pares de aislados, con el algoritmo SAHN propuesto por Sneath y Sokal (1973) del paquete estadístico de computación NTSYS pc., versión 2.1oj, por presentar los valores cofenéticos más adecuados.
Se realizó la confirmación del análisis de conglomerados y de los grupos de diversidad mediante remuestreo (2000 permutaciones), para lo cual se utilizó el paquete WinBoot (1984-1991; UPGMA bootstrapping for binary data [Nelson, 1996].
RESULTADOS Y DISCUSIÓN
Se obtuvo un total de 52 aislamientos a partir de plantas naturalmente enfermas de diferentes localidades del país (Fig. 2 y Tabla 1).

La morfología de todas las colonias monoconidiales obtenidas fue del tipo algodonoso, descrito por Stover (1962b), de crecimiento aéreo abundante con diferencias en el tipo de pigmentación. No fueron observadas colonias pionnotales o cordadas. En la Tabla 2 aparecen siete grupos de colonias de acuerdo con el color del micelio y la pigmentación. No existió relación entre las razas a la que pertenecían los aislamientos y la morfología de las colonias. Asimismo ninguno de los aislamientos formó lacinias en medio K2, lo que concuerda con los resultados de Sun et al. (1978).
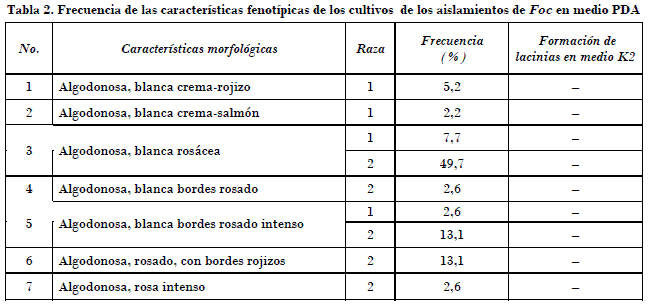
Las determinaciones cromatográficas y espectrofotométricas de los gases producidos en la superficie de los cultivos de Foc en arroz demostraron la presencia de los compuestos etenil-benceno y el biciclo 4,2,0-Octa- 1,3,5-trieno (Tabla 3 y Fig. 3), confirmados por los informes previos realizados, por lo que ya había sido ratificado previamente en los informes de Brandes (1919) y Stover (1962a), y cuya composición química coincide con la de los compuestos encontrados por Moore et al. (1991) en estudios con poblaciones de Foc del sudeste asiático y el Pacífico. Los aislamientos cubanos de las razas 1 y 2 de Foc pueden indistintamente producir o no volátiles, lo que confirman los informes previos de Stover (1962a). No se observó relación entre la producción de pigmentos, las razas a la que pertenecían los aislamientos y la presencia de compuestos volátiles, lo cual está en conflicto con lo reportado por Moore et al. (1991), que observaron la producción de estos compuestos volátiles solo en el caso de aislamientos pertenecientes a la raza 4 del patógeno en aislamientos de Asia. Sería interesante en el futuro determinar si existe correlación con la agresividad de Fusarium oxysporum mediante la comparación de aislamientos no patogénicos y patogénicos de diferentes formae specialis de esta especie.
 .
.

En la Fig. 4 aparecen las curvas ajustadas del crecimiento de las colonias de diferentes aislamientos pertenecientes a las razas 1 y 2. Todos los aislamientos se desarrollaron en el rango de temperatura entre 8 y 38°C. Se observa una meseta de crecimiento en todas las curvas en el rango de temperaturas entre 23 y 29°C. En la Tabla 4 aparece el resultado del análisis de varianza del crecimiento de los aislamientos, estimado de acuerdo con ecuaciones de ajuste de crecimiento de las colonias de un grupo de cinco aislamientos agrupados por razas, en relación con la temperatura de 27°C, que resultó la óptima para el crecimiento de las colonias según todas las ecuaciones de ajuste obtenidas. Los aislamientos pertenecientes a la raza 2 presentaron una leve tendencia a un crecimiento más rápido a la temperatura de 27°C.

La mayoría de los aislamientos (Tabla 5) pertenecieron a los GCV 0124 y 0124/125 (más del 50% de las accesiones de la colección). Estos GCV son de una amplia distribución mundial [Ploetz, 1990 y Pegg et al., 1996]. La mayoría de los aislamientos pertenecientes a estos dos grupos con dos excepciones pertenecieron a la raza 2.

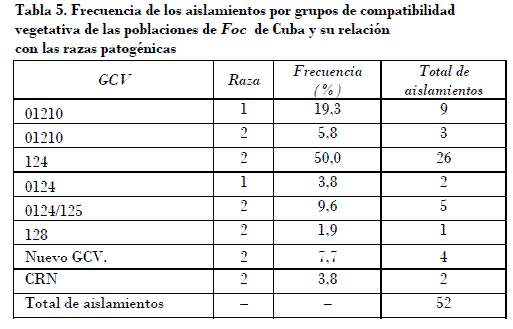
El grupo 0128 presenta una limitada distribución en Cuba (1,9% de los aislamientos). Hay informes de su presencia únicamente en Australia [Ploetz, 1990]. El único aislado encontrado en el presente estudio perteneció a la raza 2. Bentley y Bassam (1996) y Bentleyet al. (1998) informaron que los aislamientos de este GCV que analizaron se encontraban genéticamente muy cercanos a los grupos 0124 y 0125, y lo incluyeron en el mismo linaje a que pertenece el grupo 0124.Los resultados de los análisis moleculares realizados en este estudio confirman estos informes previos, y puede considerarse que evolucionó a partir del GCV 0124.
El grupo 01210 se encontró con una frecuencia relativamente amplia (21,1% de los aislamientos) y con vasta presencia entre los aislamientos de la raza 1. Está informado además solo en el condado de Dade, en la Florida en 1986, y en las Islas Caimán [Ploetz y Pegg, 1999], aunque no se han publicado estudios exhaustivos en el resto de los países del Caribe, incluido las Antillas. La totalidad de los aislamientos obtenidos del clon Manzano (Silk, AAB) en el presente estudio pertenecen al grupo 01210 en correspondencia con los resultados de Ploetz (1990), en la Florida. La enfermedad fue descrita en Cuba desde principios del siglo pasado [Smith, 1910], y dado que el mal de Panamá no se había informado hasta hace relativamente poco tiempo en el condado de Dade, en la Florida [Ploetz y Shepard, 1989], hay posibilidades de que la presencia de este GCV en la Florida pudiera tener su origen en plantas del clon Manzano llevadas desde Cuba. Los GCV 01210 y 0124 fueron clasificados en linajes dife rentes debido a la considerable distancia genética en los estudios con una colección mundial de Foc, en la que no se incluyeron aislamientos cubanos. Bentley et al. (1998) y Ploetz y Pegg (1999) han informado que comparte del 89 al 92% de sus alelos con el GCV 0120, de amplia distribución mundial (Australia, África del Sur, Costa Rica, Guadalupe, Honduras, Islas Canarias, Malasia y Taiwán) [Ploetz, 1990], en el cual se incluyen aislamientos pertenecientes a la raza 4, la que no se ha encontrado en América.
En los estudios incluidos en el presente informe el VCG 01210 resultó agrupado en el mismo grupo donde se encuentra el GCV 0124, aunque no se pudieron incluir aislamientos del GCV 0120 por no encontrarse en Cuba. Por otro lado, todo indica que la marchitez por Fusarium antecede a la llegada del Gros Michel a América, y que puede tener una íntima relación con la distribución del clon Manzano [Stover, 1962b]. O'Donnel et al. (1997) sugirieron la posibilidad de un origen polifilético de las poblaciones y de la evolución independiente de los diferentes linajes encontrados de Foc a partir de otrasformae specialis de Fusarium oxysporum. Hay evidencias que sugieren que unas razas patogénicas pueden derivarse de otras [Gordon y Martín, 1997], aunque no se han aportado pruebas irrefutables de esto.
En la Tabla 6 aparecen los aislamientos de la colección que se incluyeron en el análisis molecular del polimorfismo de los fragmentos de restricción mediante AFLP, la raza a la que pertenecen y el rendimiento de ADN obtenido en la extracción.

En las Figs. 5A, 5B y 5C se muestran los resultados de la electroforesis en gel de poliacrilamida al procesarse las muestras con la combinación de cebadores A (E-AAC/ M-CAA), B (E-AAC/M-CAG) y C (E-AGG/M-CTC).
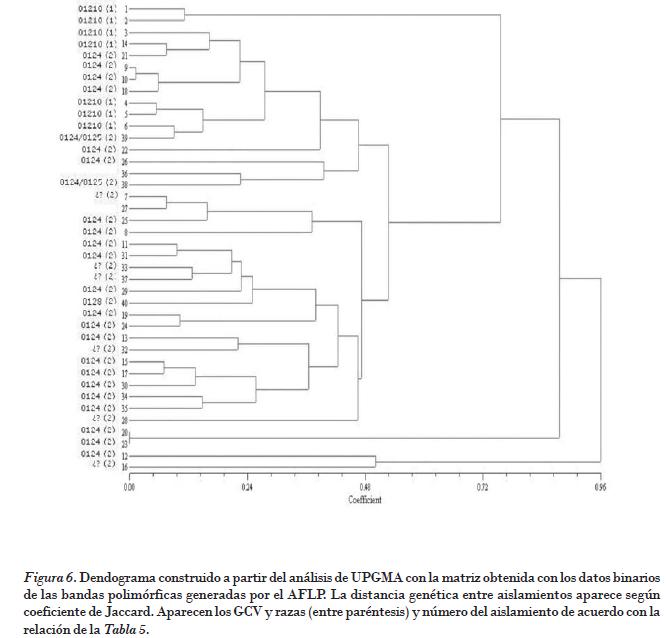
Por cuanto no se encontraron diferencias entre el análisis de la distancia genética con el empleo del coeficiente de Dice o de Jaccard, en la Fig. 6 se muestra el dendograma donde aparecen los grupos según la distancia genética de acuerdo con los coeficientes de Jaccard.
Los resultados de los análisis se encuentran de alguna manera influidos por el limitado número de combinaciones de cebadores disponibles para el estudio y por la alta proporción de aislamientos pertenecientes al GCV 0124 y la raza 2, en relación con los otros GCV representados en la colección con una menor frecuencia. De acuerdo con el análisis de UPGMA de los datos de la matriz binaria construida con el conjunto de los patrones polimórficos generados en el análisis de AFLP a partir del ADN total extraído de los aislamientos y según los coeficientes de similitud genética de Jaccard, se pudieron separar dos grandes grupos genéticos: uno que agrupa al aislamiento 12 _obtenido de Burro Criollo, GCV 0124_ y el aislamiento16 _logrado de Burro CEMSA y sin GCV dentro de Foc, el cual posiblemente es un aislamiento no patogénico no perteneciente a esta formae specialis_ y el otro que agrupa al resto de los aislamientos incluidos en el estudio y cuatro subgrupos con un nivel menor de similitud.
Los aislamientos pertenecientes al grupo 0124 se encontraron distribuidos en todos los subgrupos. Los correspondientes al GCV 01210, procedentes del clon Manzano (AAB), se agruparon, con muy poca similitud genética, junto a algunos aislamientos del GCV 0124 en unos de los subgrupos diferenciados, lo que indica que pudieran haber alelos compartidos en el genoma de estos dos subgrupos, y que si se hubiese dispuesto de más cebadores habrían podido agruparse en grupos independientes con un nivel mayor de disimilitud al encontrado.
Los dos aislamientos pertenecientes al grupo 0124/0125 y el único obtenido del GCV 0128 se agruparon dentro del grupo general correspondiente al GCV 0124.
La confirmación por remuestreo _2000 permutaciones, UPGMA bootstrapping_ del análisis de conglomerados y de los grupos de diversidad, permitió confirmar que el aislamiento 16 no pertenece a Foc, a pesar de estar presente en el sistema radical afectado de la planta. Asimismo accedió verificar que hay una considerable distancia genética entre los aislamientos de diferentes GCV, y que los miembros de un mismo GCV formaron subgrupos entre sí y tienen un alto nivel de similitud genética. Los aislamientos pertenecientes al GCV 0124 fueron nueve obtenidos de Burro Cemsa, 10 del Burro CEMSA y 18 obtenidos del Pisang awak (con un nivel de similitud del 99,8%); 7, 21 y 27 todos de Burro Criollo (con un nivel del 96,1%); 24 de Burro Criollo y 19 de Burro CEMSA (con el 98,9%); 17 de Pisang awak y 15 de Burro Criollo (con el 86,4%); los aislamientos 13, 15 y 30 de Burro Criollo, 34 y 35 de Burro CEMSA, y 32 de Pisang awak (con el 89%).
Los aislamientos 14 (único aislamiento obtenido del clon Manzano perteneciente al GCV 0124) y 21 (Burro Criollo GCV 0124) mostraron una alta identidad genética (95,3%).
Los aislamientos 38 y 39 obtenidos de Burro Criollo y pertenecientes al GCV 0124/0125 mostraron en el análisis de remuestreo una alta similitud con los aislamientos 26, 22 de Burro Criollo correspondientes al GCV 0124, pero con un nivel más moderado de soporte (79%), lo que indica que comparte alelos con él y puede considerarse que se derivó de este grupo de compatibilidad, como se ha sugerido previamente.
El aislamiento 40, perteneciente al GCV 0128, quedó agrupado en un clúster junto con aislamientos del GCV 0124 (aislamientos procedentes de los clones Burro CEMSA y Burro Criollo), pero con un nivel menor de similitud (66,7%), lo que argumenta a favor de que este GCV comparte alelos comunes con el 0124, aunque se distancia genéticamente de este en alguna medida.
Los aislamientos pertenecientes al GCV 01210 _aislamientos 4, 5, 6, todos obtenidos del clon Manzano de diferentes regiones del país_ presentaron una alta identidad genética entre sí, con un nivel del 95,8%. Estos aislamientos, al mismo tiempo, presentan muy baja similitud genética con los aislamientos del GCV 0124, y recibieron muy bajo nivel de soporte en el análisis de remuestreo (< del 44%).
Como puede apreciarse, los aislamientos con mayor similitud genética estuvieron en todos los casos agrupados en el mismo GCV, lo que permite argumentar a favor de esta vía de clasificación genética de las poblaciones. Los aislamientos pertenecientes al GCV 0124 se encontraron distribuidos en todos los grupos genéticos, para considerar, dada además su amplia distribución geográfica, que comparte sus alelos con la mayoría de los aislamientos y GCV, y que por tanto es un GCV original, del cual posiblemente se diferenciaron los genotipos pertenecientes a los GCV 0124/0125 y 0128. Esto confirma informes previos de los estudios con colecciones mundiales de la variabilidad de las poblaciones de Foc [Ploetz, 1990; Pegg et al., 1996; Koenig, et al., 1997].
En estudios similares donde se incluyeron aislamientos pertenecientes a todos los GCV conocidos de Foc, el GCV 01210 se agrupó en un linaje independiente [Koenig et al., 1997] o en un linaje en el que se encuentra el GCV 0120 [Bentley et al., 1998], también de gran distribución mundial, el cual no fue encontrado en Cuba, y por tanto no se incluyó en el análisis. En el presente estudio los aislamientos del GCV 01210 presentaron una gran identidad genética entre sí y se agruparon con algunos aislamientos del GCV 0124, lo que indica que comparte algunos alelos comunes en su genoma; pero en el remuestreo se encontró poco nivel de soporte (< 40%), de ahí que ambos GCV sean genéticamente distantes, lo que apoya los informes previos mencionados. No obstante, el hecho de que el GCV 01210 se ha encontrado solo a nivel mundial _en Cuba, donde hay referencias desde el siglo XIX de la presencia del hongo en el clon Manzano [Pérez, 2004] y la Florida, en que fue primeramente informado por Ploetz y Sheperd (1989), sitios donde no se encuentra el GCV 0120, así como la falta de similitud genética encontrada con el GCV 0124 en el presente estudio y en los informes previos_ apoya el enfoque de un origen polifilético de las poblaciones de Foc, como han sugerido O'Donnell et al. (1997).
No obstante, se detectaron suficientes divergencias entre los aislamientos individuales como para considerar oportuno realizar un estudio posterior, en regiones más conservadas del ADN de Foc. Asimismo, el análisis de las bandas polimórficas comunes obtenidas entre los aislamientos del GCV 01210, no presentes en las de GCV 0124 0124/0125 y 0128, permite recomendar para el futuro su secuenciación, y ver la posible generación de cebadores para detectar y diferenciar por PCR los diferentes GCV del país, lo que agilizaría, haría más sensible y abarataría el diagnóstico del patógeno.
Los resultados permiten aseverar que los aislamientos del Burro Criollo (Bluggoe ABB, raza 2) son genéticamente muy similares a los obtenidos del clon Pisang awak (Burro Vietnamita, ABB), el cual ha sido atacado en la Florida por aislamientos pertenecientes a la raza 1 [Ploetz, 1990], y confirman los resultados de las inoculaciones artificiales realizadas por Pérez et al. (2004), en que ambos clones presentaron reacciones similares frente a dos aislamientos de la raza 2 a los GCV 0124 y 0128 obtenidos del clon Burro Criollo.
Se ha discutido la falta de estudios de la variabilidad de las poblaciones del patógeno en el nuevo mundo [Pérez, 2004]. Sería deseable completar un estudio de la composición de las poblaciones de Foc a nivel del Caribe y América del Sur que permitiera reconocer las poblaciones existentes en esta región y obtener un conocimiento más completo de la variabilidad real de las poblaciones de Foc en esta zona.



CONCLUSIONES
• La morfología de las colonias de los aislamientos de Foc en PDA perteneció en todos los casos al tipo algodonoso blanco con pigmentaciones. No se observó producción de lacinias en medio K2 en ninguno de los aislados de la colección.
• Los aislados estudiados pueden dividirse en dos grupos de acuerdo con la producción (odoratum) o no (inodoratum) de compuestos volátiles. Este carácter no está relacionado ni con las razas patogénicas ni con la producción de pigmentos.
• Las poblaciones cubanas de Foc determinadas hasta el presente pertenecen a los GCV 01210, 0124, 0124/ 0125 y 128. Existen poblaciones correspondientes a GCV vegetativos diferentes a los grupos 0120, 0121, 0122, 0123, 0124, 0124/0125, 0128, 0129 01210, 01212, 01213, 01214 y 01215 que no pudieron ser clasificados en ningún GCV. Su frecuencia es muy baja y requieren de una recomprobación de su patogenicidad en Musa.
• Las poblaciones de Foc encontradas en el clon Manzano (subgrupo Silk) pertenecieron en casi todos los casos al GCV 01210. Las poblaciones encontradas sobre el clon Burro Criollo y Burro CEMSA (subgrupo Bluggoe, ABB) pertenecieron casi exclusivamente a los GCV 0124/0125, 0124 y 0128. Los GCV tienen por tanto a nivel local una alta correlación con las razas y los clones de donde se obtuvieron los aislamientos.
• Los aislamientos dentro de cada GCV tienen una alta identidad genética entre sí y son genéticamente distantes de los pertenecientes a los de otros GCV. El GCV 01210 es genéticamente diferente del 0124, aunque comparten alelos comunes, por lo que se consideran sus orígenes independientes. Se comprobó que los aislamientos de los GCV 0128 y 0124/0125 se agruparon con el GCV 0124, de ahí que se pueda asumirque comparten alelos con el 0124 y que pudieran, dada su más baja distribución y frecuencia, haberse derivado de este grupo de amplia distribución mundial y nacional.
• La compatibilidad vegetativa ha mostrado ser una potente herramienta para el diagnóstico de poblaciones y la cuarentena. Los resultados del presente estudio se deben tener en cuenta para la exclusión cuarentenaria de otros GCV no presentes en el país en el germoplasma que se importa con diferentes fines. · El análisis de las bandas polimórficas comunes obtenidas entre los aislados del GCV 01210 no presentes en las de GCV 0124 0124/0125 y 0128 permite recomendar para el futuro su secuenciación, y ver la posible generación de cebadores para detectar y diferenciar por PCR los diferentes GCV del país, lo que agilizaría, haría más sensible y abarataría el diagnóstico del patógeno.
REFERENCIAS
Andel-Satar, M.; M. S. Khalil; I. N. Mohmed: «Molecular Phylogeny of Fusarium Species by AFLP Fingerprinting», African Journal of Biotechnology 2 (3):51-55, Nigeria, 2003.
Bentley, S.; B. J. Bassam: «A Robust DNA Amplification Fingerprinting System Applied to Analysis of Genetic Variation within Fusarium oxysporum f. sp. cubense», J. Phytopathology 144:207-213, EE.UU., 1996.
Bentley, S.; K. G. Pegg; N. Y. Moore; D. R. Davis; I. W. Buddenhagen: «Genetic Variation Among Vegetative Compatibility Groups of Fusarium oxysporum f. sp. cubense Analyzed by DNA Fingerprinting », Phytopathology 88:1283-1293, EE.UU., 1998.
Boehm, E. W. A.; R. C. Ploetz; H. C. Kistler: «Statistical Analysis of Electrophoretic Karyotype Variations Among Vegetative Compatibility Groups of Fusarium oxysporum f. sp. cubense», Molecular Plant- Microbe Interactions 7:196- 207, EE.UU., 1993. Booth, C.; Fusarium Laboratory Guide to the Identification of the Major Species, CMI, Kew, Surrey, Inglaterra, 1977. Brandes, E. W.: «Banana Wilt», Phytopathology 9:339-389, EE.UU., 1919. Brake, V. M.; K. G. Pegg; J. A. G Irwin; P. W. Langdon: «Vegetative Compatibility Groups within Australian Populations of F. oxysporum f. sp. cubense the Cause of Fusarium wilt of Banana», Aust. J. Agric. Res. 41:863-870, 1990. Correll, J. C.; J. E. Puhalla; R. W. Schneider: «Identification of Fusarium oxysporum f. sp. apii on the Basis of Colony Rise Virulence and Vegetative Compatibility», Phytopathology 76:396-400, EE.UU., 1986. Correll, J. C.; C. J. R. Klittich; J. F. Leslie: «Nitrate Non-Utilizing Mutants of Fusarium oxysporum and Their Use in Vegetative Compatibility Tests», Phytopathology 77:1640-1646, EE.UU., 1987. Cove, D. J.: «Chlorate Toxicity in Aspergillus nidulans: the Selection and Characterization of Chlorate Resistant Mutants», Heredity 36:191-203, EE.UU., 1976.
Gordon, T. R.; R. D. Martyn; «The Evolutionary Biology of Fusarium oxysporum», Annual Review of Phytopathology 35:111-128, EE.UU., 1997. Jaccard, P.: «Nouvelles recherches sur la distribution florale»,Bull. Soc. Vaud. Sci. Nat. 44:223-270, Francia, 1908. Johnston, J. R.: «La enfermedad del plátano en Cuba», Circ. 47, 13, Estación Experimental Agronómica de Santiago de las Vegas, La Habana, 1915. Koenig, R. L.; R. C. Ploetz; H. C. Kistler: «Fusarium oxysporum f. sp. cubense Consist of a Small Number of Divergent and Globally Distributed Clonal Lineages», Phytopathology 87:915-923, EE.UU., 1997. Leslie, J. F.: Genetic Exchange within Sexual and Asexual Populations in the Genus Fusarium. Fusarium wilt of Banana, APS Press St. Paul, EE.UU., 1990, pp. 37-48. ____: «Vegetative Compatibility in Fungi», Ann. Rev. of Phytopath. 31:127- 151, EE.UU., 1993.
Masdek, N.; M. Mahmood; A. Molina; S. C. Hwang; A. Dimyati; R. Tangaveli; I. Omar: «Global Significance of Fusarium Wilt: Asia», Abstracts of Papers 2nd. International Symposium on Fusarium wilt on Banana, Promusa-INIBAP/EMBrapa, Salvador de Bahía, Brasil, 22-26 sept. 2003.
Möller, E. M.; G. Bahnweg: «A Simple and Efficient Protocol for Isolation of High Molecular Weight DNA from Filamentous Fungi, Fruit Bodies and Infected Plant Tissues», Nucleic Acids Research 20:6115-6116, EE.UU., 1992.
Moore, N. Y.; P. Heargraves; K. E. Pegg; J. A. G. Irvin: «Characterization of Strains of Fusarium oxysporum f. sp. cubense by Production of Volatiles», Aust. J. of Bot. 39:161-166, Australia, 1991.
Moore, N. Y.; K. Pegg; A. R. Allen; J. A. G. Irvin: «Vegetative Compatibility and Distribution of Fusarium oxysporum f. sp. cubense in Australia», Aust. J. Exp. Agric. 33:797-802, Australia, 1993.
Nasdir, N.: «Fusarium wilt Race 4 in Indonesia», Research Institute for Fruits West Sumatra, Indonesia, Abstracts of Papers 2nd. International Symposium on Fusarium wilt on Banana, Promusa-INIBAP/EMBRAPA, Salvador de Bahía, Brasil, 22- 26 septiembre del 2003.
Nelson, R. J.: «WinBoot Yap IV: a program for Performing Bootstrap Analysis ~of Binary Data to Determine the Confidence Limits of UPGMA-Based Dendrograms», IRRI, Manila Philippines, http://www.irri.org/science/software/winboot.asp. 1996.
O'Donnell, K.; H.C. Kistler; E. Cigelnick; R. C. Ploetz: «Multiple Evolutionary Origins of the Fungus Causing Panama Disease of Banana: Concordant Evidence from Nuclear and Mitochondrial Gene Genealogies», Proc. Natl. Acad. Sci. 95:2044-2049, EE.UU., 1997.
O´Neill, N. R.; B. A. Bailey; P. B. Berkum: «AFLP, a Novel PCR-Based DNA Analysis Technique, Reveals Levels of Genomic Variation within Species of Colletotrichum, Fusarium and Dendryphion», http://www.fgsc.net/fgn38/duteau.html. 2003 (consultado en noviembre del 2004).
Pegg, K. G.; N. Y. Moore; S. Sorensen: «Fusarium Wilt in the Asian Pacific Region», Proc. Int. Symp. Recent Developments in Banana Cultivation Techn., International Network of Banana and Plantain/ ASNET, Los Baños, Laguna, Filipinas, 1993, pp. 225-269.
Pegg, K. G.; N. Y. Moore; S. Bentley: «Fusarium Wilt of Banana in Australia: a Review», Aust. J. of Agric. Res. 47:637-650, Australia, 1996.
Pérez, L.; J. M. Álvarez; M. Pérez: «Economic Impact and Management of Black Leaf Streak Disease in Cuba», Mycosphaerella Leaf Spot Diseases of Bananas: Present Status and Outlook, Proceedings of the International Workshop on Mycosphaerella Leaf Spot Diseases, San José, Costa Rica, 2002, pp. 71-83.
Pérez, L.: «Fusarium wilt (Panama Disease) of Bananas: an Updating Review of the Current Knowledge on the Disease and Its Causal Agent», Memorias del XV ACORBAT, Oaxaca, México, oct. 2004, pp 1-14.
Pérez, L.; A. Batlle; J. Fonseca; V. Montenegro: «Reaction of Cultivars of Bananas and Plantains to Fusarium oxysporum f. sp. cubense», Resúmenes del V Seminario Científico Internacional de Sanidad Vegetal, Basílica Menor de San Francisco de Asís, 24-28 de mayo del 2004, La Habana.
Ploetz, R. C.; J. C. Correll: «Vegetative Compatibility Among Races of Fusarium oxysporum f. sp. cubense», Plant Disease 72:325-328, EE.UU., 1988. Ploetz, R. C.; E. S. Shepard: «Fusarium Wilt of Banana in Florida», Mycol. Research 93:242-245, EE.UU., 1989.
Ploetz, R. C.: Population Biology of Fusarium oxysporum f. sp. cubense. Fusarium Wilt of Banana, APS Press St. Paul, EE.UU., 1990, pp. 63-76.
Ploetz, R. C.; K. G. Pegg: Fusarium Wilt. Diseases of Banana, Abaca and Enset, CABI Publishing,. Colombia, 1999, pp. 143-159.
Promega: «Preparación de geles de poliacrilamida», GenePrint STR.Systems (Silver Stain Detection), Technical Manual, Part # TMD004, Revised 5/1998.
Puhalla, J. E.: «Classification of Strains of Fusarium oxysporum on the Basis of Vegetative Compatibility», Canadian Journal of Botany 63:179-183, Canadá, 1985.
Puhalla, J. E.; P. T. Spieth: «A Comparison of Heterokaryosis and Vegetative Compatibility Among Varieties of Gibberella ujikuroi (Fusarium moniliforme)», Exptl. Mycol. 9:39-47, EE.UU., 1985.
Smith, E. F.: «A Cuban Banana Disease» (Abstr.), Science 31:754-755, EE.UU., 1910.
Sneath, P. H. A.; R. R. Sokal: Numerical Taxonomy, Freeman, San Francisco, 2000 by Applied Biostatistics, 1973.
Stover, R. H.; B. H. Waite: «Studies on Fusarium Wilt of banana. V. Pathogenic and Distribution of F. oxysporum f. sp. cubense Races 1 and 2», Can. J. Botany 38:51-61, Canadá, 1960.
Stover, R. H.: «Studies on Fusarium Wilt of Bananas. VIII. Differentiation of Clones by Cultural Interaction and Volatile Substances», Can. Journal of Bot. 40:1467-1471, Canadá, 1962a.
____: «Fusarium Wilt (Panama Disease) of Bananas and Other Musa species», Phytopathological Paper 4, CMI, Kew, Surrey, Inglaterra, 1962b.
Stover, R. H.; I. W. Buddenhagen: «Banana Breeding, Polyploidy, Disease Resistance and Productivity», Fruits 41:175-191, EE.UU., 1986.
Su, H. J.; T. J. Chuang; W. S. Kong: «Physiological Race of Fusarial Wilt Fungus Attacking Cavendish Banana of Taiwan», Taiwan Banana Research Institute, Spec. Publ. 2, 1977.
Su, H. J.; S. C. Hwang; W. H. Ko: «Fusarium Wilt of Bananas in Taiwan», Plant Disease 70:813-818, EE.UU., 1986.
Sun, E. J.; H. J. Su; W. H. Ko: «Identification of Fusarium oxysporum f. sp. cubense Race 4 from Soil or Host Tissue by Cultural Characters», Phytopathology 68:1672-1673, EE.UU., 1978.
Vidal, A.: «Sigatoka negra en Cuba. Nuevos Focos de plagas y enfermedades », Boletín Fitosanitario de la FAO 40:(1-2), Italia, 1992.
Vos, P.; R. Hogers; M. Bleeker; M. Reijans; T. Van de Lee; M. Hornes; A. Fritjers; J. Peleman; M. Kuiper; M. Zabeau: «AFLP: a New Technique for DNA Fingerprinting», Nucleic Acid Research, 23:4407-4414, EE.UU., 1995.














