Meu SciELO
Serviços Personalizados
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Cubana de Informática Médica
versão On-line ISSN 1684-1859
RCIM vol.5 no.2 Ciudad de la Habana jul.-dez. 2013
ARTÍCULO ORIGINAL
Funciones de transferencia en el perceptrón multicapa: efecto de su combinación en entrenamiento local y distribuido
Transfer functions in the multilayer perceptron: effects of its combination on local and distributed training
MsC. Yuleidys Mejías César,I Dr. Ramón Carrasco Velar,II Ing. Isbel Ochoa Izquierdo,III Ing. Edel Moreno LemusIII
IMáster en Bioinformática. Profesor Asistente. Universidad de las Ciencias Informáticas. La Habana, Cuba. E-mail: ymejias@uci.cu
IIDoctor en Ciencias Químicas. Profesor Auxiliar. Investigador Titular. Universidad de las Ciencias Informáticas. La Habana, Cuba. E-mail: rcarrasco@uci.cu
IIIIngeniero. Profesor Instructor. Universidad de las Ciencias Informáticas. La Habana, Cuba.
El perceptrón multicapa (PMC) figura dentro de los tipos de redes neuronales artificiales (RNA) con resultados útiles en los estudios de relación estructura-actividad. Dado que los volúmenes de datos en proyectos de Bioinformática son eventualmente grandes, se propuso evaluar algoritmos para acortar el tiempo de entrenamiento de la red sin afectar su eficiencia. Se desarrolló un algoritmo para el entrenamiento local y distribuido del PMC con la posibilidad de variar las funciones de transferencias para lo cual se utilizaron el Weka y la Plataforma de Tareas Distribuidas Tarenal para distribuir el entrenamiento del perceptrón multicapa. Se demostró que en dependencia de la muestra de entrenamiento, la variación de las funciones de transferencia pueden reportar resultados mucho más eficientes que los obtenidos con la clásica función Sigmoidal, con incremento de la g-media entre el 4.5 y el 17 %. Se encontró además que en los entrenamientos distribuidos es posible alcanzar eventualmente mejores resultados que los logrados en ambiente local.
Palabras clave: funciones de transferencia, perceptrón multicapa, redes neuronales.
ABSTRACT
The multilayer perceptron (PMC) ranks among the types of artificial neural networks (ANN), which has provided better results in studies of structure-activity relationship. As the data volumes in Bioinformatics' projects are eventually big, it was proposed to evaluate algorithms to shorten the training time of the network without affecting its efficiency. There were evaluated different tools that work with ANN and were selected Weka algorithm for extracting the network and the Platform for Distributed Task Tarenal to distribute the training of multilayer perceptron. Finally, it was developed a training algorithm for local and distributed the MLP with the possibility of varying transfer functions. It was shown that depending on the training sample, the change of transfer functions can yield results much more efficient than those obtained with the classic sigmoid function with increased g-media between 4.5 and 17 %. Moreover, it was found that with distributed training can be achieved eventually, better results than those achieved in the local environment.
Key words: transfer functions, multi-layer perceptron, neural networks.
INTRODUCCIÓN
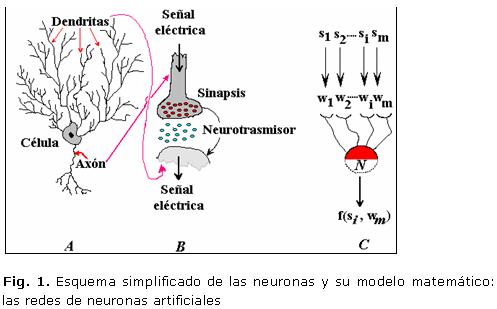
Las redes de neuronas artificiales (RNA) son una aproximación matemática al funcionamiento del cerebro que pueden representarse esquemáticamente para su mejor comprensión (Fig. 1).
Su utilización se ha extendido a campos tan disímiles como la cromatografía,1,2 para la selección de hiperparámetros en Least Squares Support Vector Machine (LS-SVM) approximators with Gaussian Kernels,3 en estudios cuantitativos de relación estructura-actividad4 y estructura-propiedad,5 para la selección de aspectos,6 entre otras diferentes y múltiples aplicaciones.
Se conocen diferentes compuestos químicos en el cerebro y otras partes del SNC que actúan como neurotrasmisores a diferentes niveles, los cuales trasmiten señales entre células nerviosas que contienen información. A estos trasmisores de señales se les puede catalogar como pequeños, aminas, aminoácidos y otros según su estructura química o como Inhibidores o excitadores por la función. Independientemente de los criterios de clasificación, es una realidad que el cumplimiento de su función bioquímica está íntimamente ligado a su estructura química y que la respuesta o efecto dependerá de un cierto valor de potencial que puede ser representado mediante una determinada función matemática.
Las redes de neuronas artificiales, como modelo matemático, pretenden asemejar el comportamiento de dichos trasmisores de información pero, según se infiere de lo anteriormente expresado, estas funciones no pueden ser iguales para cada molécula orgánica que están actuando sistemáticamente para mantener en equilibrio las distintas funciones orgánicas.
Esa es la razón fundamental para iniciar este tipo de trabajo en las RNA con vistas evaluar el comportamiento de los modelos en situaciones particulares, variando las funciones de transferencia de la red, como una aproximación adicional al comportamiento del cerebro, sin pretender vincular cada una de dichas funciones matemáticas a un neurotransmisor específico.
Por otra parte, para describir la estructura química se puede partir de enfoques diferentes entre los que se encuentra la representación molecular como un grafo conexo y la división de la molécula o grafo molecular en fragmentos o subgrafos, que pueden o no estar ponderados en las aristas (enlaces) o los vértices (átomos).7 Atendiendo a la naturaleza del problema y las capacidades de las RNA de extraer información no-lineal de un conjunto de datos, se decidió ampliar las investigaciones en este campo para incrementar las potencialidades de la Plataforma en la búsqueda de modelos eficientes de relación estructura-actividad biológica.
Al enfrentar inicialmente el estudio de las RNA, se identificaron dos interrogantes fundamentales. Una de ellas estaba relacionada con el tiempo que requiere el entrenamiento de las redes y la capacidad de cómputo que consumen, máxime cuando se cuenta con grandes volúmenes de información, como es el caso de los proyectos de Bioinformática. La otra interrogante estaba asociada a las funciones de transferencia de las redes y cómo su combinación podría afectar la calidad de la respuesta durante el entrenamiento de la red. La función de transferencia, o de activación, como también se le conoce, es uno de los elementos principales del funcionamiento de las RNA. La función de activación de una neurona artificial simula la respuesta de una neurona biológica ante un cierto estímulo. Se conoce que el cerebro posee un comportamiento diferente ante cada estímulo, sin embargo, en la mayoría de los casos las RNA son entrenadas y utilizadas haciendo uso de una única función de transferencia.
Por todo ello se trazó como objetivo determinar el efecto de la variación de las funciones de transferencia en la calidad de la respuesta del entrenamiento de un PMC, en ambiente local y distribuido.
El éxito de esta propuesta permitirá agilizar la etapa de desarrollo de nuevos fármacos, ya que a partir de la descripción de la estructura de una molécula, la red neuronal entrenada podrá predecir, con un porciento de error especificado por el usuario, la actividad biológica asociada a una molécula nueva a partir de sus descriptores.
METODOLOGÍA
Herramientas utilizadas
Teniendo en cuenta las características del trabajo a desarrollar, se escogió Weka (Waikato Environment for Knowledge Analysis) para la extracción del algoritmo de las RNA, ya que es un software de código abierto que posee una colección de algoritmos de aprendizaje automático para la extracción de datos.8 Se escogió la Plataforma de Tareas Distribuidas (Tarenal) para realizar la distribución del entrenamiento ya que ofrece una alternativa de cómputo que aglutina en un solo conglomerado un conjunto de estaciones de trabajo.9 Se seleccionó Java como lenguaje de programación para realizar los cambios necesarios a los algoritmos presentes en el Weka y en la plataforma Tarenal y así ajustarlos al problema presente en esta investigación, puesto que es un lenguaje potente y con grandes capacidades de interconexión TCP/IP, por lo cual se puede acceder a la información disponible en red con mucha facilidad y seguridad.
Por otra parte se escogió Eclipse como herramienta IDE por ser un entorno de desarrollo integrado distribuido con el código abierto y multiplataforma. Se seleccionó el SPSS (Statistical Package for the Social Sciences),10 software estadístico informático muy usado por las funcionalidades que brinda; el módulo utilizado en la presente investigación fue el de Pruebas no Paramétricas, el cual permite realizar distintas pruebas estadísticas especializadas en distribuciones no normales.11 El SPSS fue empleado con el objetivo de comparar los resultados de los 2 algoritmos utilizados, o sea, el algoritmo para el entrenamiento local de un perceptrón multicapa y el algoritmo distribuido, y de esta forma conocer si son significativas las diferencias entre las respuestas dadas por dichos algoritmos. Para ello se realizaron pruebas no paramétricas utilizando la medida de evaluación (g-media) de los algoritmos para realizar las comparaciones.
Se realizaron diferentes estudios sobre el funcionamiento y rendimiento del perceptrón multicapa, tanto en ambientes locales como distribuidos. Todos las pruebas o experimentos que se llevaron a cabo fueron realizados en computadores personales con un procesador Pentium 4 a 1.86 GHz de frecuencia y con 1Gb de RAM.
Arquitectura del perceptrón multicapa
La arquitectura del perceptrón multicapa se compone de un número de neuronas en la capa de entrada, el cual depende de la cantidad de componentes del vector de entrada. Depende también de la cantidad de capas ocultas y del número de neuronas de cada una de ellas así como del número de neuronas en la capa de la salida, el cual depende a su vez del número de componentes del vector de salida o patrón objetivo.
Tanto el vector de entrada como el de salida están definidos por el problema a resolver, por lo que la cantidad de neuronas en la capa de entrada es la cantidad de descriptores de la muestra escogida. En todos los casos, la cantidad de neuronas en la capa de salida será 1, ya que la función objetivo tendrá como salida una única respuesta (componentes activos o inactivos para las 2 primeras muestras y componentes de clase 1 o clase 2 para la última muestra).
Respecto al criterio a tener en cuenta para la selección de las neuronas de las capas ocultas surge una interrogante, este número en general debe ser lo suficientemente grande como para que se forme una región compleja que pueda resolver el problema, sin embargo no debe ser muy grande pues la estimación de los pesos puede ser no confiable para el conjunto de los patrones de entrada disponibles.
La mayor eficiencia del perceptrón multicapa en cuanto a su arquitectura se logra principalmente con una mayor experiencia de su diseñador, aunque también existen varios criterios para realizar esta selección.
A partir de una serie de criterios consultados en la bibliografía, se decidió trabajar con una sola capa de neuronas ocultas, ya que de esta manera se pueden representar todas las funciones matemáticas conocidas hasta el momento.12
Para determinar la cantidad de neuronas en las capas ocultas existen varios criterios, de ellos se seleccionó el que plantea que la cantidad de neuronas ocultas debe ser las dos terceras partes de la cantidad de neuronas de entrada más la cantidad de neuronas de salida.12
Entrenamiento del perceptrón multicapa
Inicialmente se fijaron algunos valores necesarios en el entrenamiento de la red como es el caso del momentum, con 0.001 y la velocidad de aprendizaje, con 0.003.
Luego de seleccionados los valores con los que se van a entrenar, y de definir la arquitectura de la red, la velocidad de aprendizaje y el momentum, además de la cantidad de patrones de entrada con que se va a realizar el entrenamiento, la aplicación genera aleatoriamente los pesos sinápticos.
Cada patrón de entrada se hace pasar a través de la estructura activando cada neurona y generando salidas en estas, dichas salidas son multiplicadas por los pesos sinápticos y constituyen la entrada de las neuronas de la capa siguiente, así sucesivamente hasta llegar a la capa de salida donde el resultado final se compara con el resultado esperado, generando un error el cual es propagado por toda la red (backpropagation) hasta llegar al origen, corrigiendo los valores sinápticos. Así sucede con cada patrón hasta que todos hayan pasado a través de la red, esto constituye una iteración o epoch.
El criterio para establecer la condición de parada para el entrenamiento estuvo dado por la cantidad de iteraciones o epochs, o sea, el investigador define la cantidad de iteraciones que considere necesaria para obtener una red neuronal lo suficientemente entrenada para procesar sus datos de manera satisfactoria.
Muestras utilizadas durante la investigación. Características fundamentales
Para realizar los estudios se utilizaron 3 muestras, dos de ellas de naturaleza bioinformática, que se utilizaron fundamentalmente para analizar el efecto que podría ocasionar la variación de las funciones de transferencia en la eficiencia de la respuesta de la red. Para realizar el entrenamiento estas muestras se dividieron en 2 partes, el 80 % para realizar el entrenamiento del perceptrón y el 20 % restante para realizar las pruebas de la red entrenada. Las muestras se relacionan a continuación:
- Muestra 1: Cefalosporinas
Las cefalosporinas son potentes compuestos antibacteriales pertenecientes a la familia de los ß-lactámicos que poseen bajan toxicidad. Estos antibióticos pueden utilizarse para el tratamiento incluso de niños con fallos renales o hepáticos.13
La muestra utilizada contiene 179 descriptores y 104 instancias (o patrones de entrenamiento). Se conoce que todos los compuestos de la muestra son activos, es por ello que la investigación va enfocada a determinar los aspectos estructurales descritos por las variables independientes, en la actividad. Para ello se tuvo que adaptar la variable dependiente (actividad biológica) para que pudiera ser tratada como un problema de clasificación. El procedimiento seguido fue el de determinar el promedio de los valores de la actividad biológica (AB) reportada para cada compuesto de la muestra y considerar como muy activos aquellos cuya AB fuera mayor o igual que el promedio, y como poco activos los que no cumplieran con este criterio. De esta manera la muestra quedó dividida en 52 elementos muy activos y 52 poco activos.
- Muestra 2: Inhibidores del Factor 1 del receptor esteroidogénico (SF-1)
La segunda muestra corresponde a los Inhibidores del Factor 1 del receptor nuclear esteroidogénico (SF-1). Este es un ensayo de dosis-respuesta basado en células para la inhibición del receptor huérfano. La muestra utilizada contiene 311 descriptores, 315 instancias (o patrones de entrenamiento) y la actividad biológica a predecir va a estar dividida en 175 elementos activos y 140 no activos.
- Muestra 3: Reconocimiento facial
La tercera muestra utilizada es de reconocimiento facial. Está destinada a la identificación automática de una persona en una imagen digital, mediante la comparación de determinadas características faciales a partir de una imagen digital o un fotograma de un video. A pesar de no ser de la rama de bioinformática se decidió trabajar con esta muestra por la cantidad de patrones que contiene. La muestra contiene 70 mil patrones de entrenamiento con 376 descriptores, de los cuales 63175 pertenecen a la clase 1 y 6825 a la clase 2.
Reducción del número de variables
Para realizar la reducción de variables se emplearon como métodos de búsqueda los Algoritmos Genéticos, el Enfriamiento Simulado y un Algoritmo Híbrido (compuesto por el Greddy y Algoritmos Genéticos). También se tuvieron en cuenta las medidas de evaluación CFS y Consistencia. La combinación de los métodos de búsqueda con las medidas de evaluación permitió obtener varias posibles muestras de entrenamiento, las cuales fueron utilizadas para seleccionar las de entrenamiento.
RESULTADOS Y DISCUSIÓN
A continuación se muestran los resultados experimentales obtenidos, tanto en las pruebas realizadas en una computadora personal como en la plataforma de tareas distribuidas Tarenal. Se realiza una comparación de los resultados obtenidos en la calidad del modelo medido por la g-media, y el tiempo de ejecución del entrenamiento. También se comparan los valores de la g-media al entrenar las redes con diferentes funciones de transferencia.
Resultados experimentales del entrenamiento del perceptrón para la muestra de cefalosporinas
De los 104 patrones de entrenamiento se utilizaron 83 para realizar el entrenamiento y los 21 restantes para desarrollar la validación. Se mantuvieron constantes los siguientes parámetros:
Cantidad de neuronas en la capa de entrada: 47
Cantidad de neuronas en la capa oculta: 32
Cantidad de neuronas en la capa de salida: 1
Velocidad de aprendizaje: 0.003
Momentum: 0.001
Algoritmo de Búsqueda: Algoritmo Genético con 0,6 de probabilidad de cruzamiento y CFS como Medida de Evaluación.14
Los parámetros que se variaron fueron: cantidad de iteraciones y las funciones de transferencia utilizadas en la capa oculta y en la capa de salida. En la tabla 1 se muestran los resultados obtenidos.
Como se puede apreciar en la tabla 1, el aumento del número de las iteraciones es directamente proporcional al aumento del tiempo (lo cual es bastante predecible), no siendo así con el valor de la G-Media, ya que en algunos casos este valor puede aumentar (aumenta la eficiencia de la respuesta del algoritmo, la red aprende más), puede disminuir (la red no aprende) o simplemente mantenerse constante.
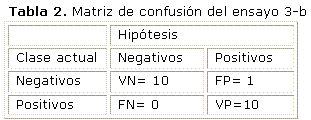
De estos resultados se puede inferir que los mejores resultados de la red para la muestra analizada no se obtienen precisamente con la función de transferencia Sigmoidal en todas las capas, sino con la combinación de funciones Tangente-Sigmoidal en la capa oculta y Sigmoidal en la capa de salida, para la que se obtuvo un valor de G-Media igual a 0.9534. Se puede apreciar en la matriz de confusión asociada al ensayo 3-b (tabla 2) que se solo se clasificó incorrectamente un valor del conjunto de validación.
Otro aspecto a resaltar es que para la muestra en cuestión no es conveniente utilizar la combinación de funciones Lineal (en la capa oculta) y Sigmoidal (en la capa de salida), ya que la red "no aprende". El valor de la G-Media igual a cero indica que la red no clasificó correctamente los patrones muy activos (+1) o ninguno de los pocos activos (-1) presentes en la muestra de validación. Se puede apreciar en la matriz de confusión asociada a este ensayo (tabla 3) que a pesar de que se clasificaron bien 7 de los 10 elementos muy activos, no se clasificó correctamente ningún valor de los elementos poco activos.
Es importante destacar también el resultado obtenido con la combinación de funciones Sigmoidal y Lineal, ya que con solo 1500 iteraciones se obtuvo el segundo mejor valor de G-Media de las pruebas realizadas, al igual que con la combinación de funciones (b) del experimento 2.
Otro aspecto a observar es que en muchos casos no es necesario realizar muchas iteraciones de la red, ya que el mejor valor de la G-Media se logra con pocas iteraciones, como es el caso del ensayo 1-c.
Resultados experimentales del entrenamiento del perceptrón para la muestra de SF-1
Inicialmente se hizo un agrupamiento (clusterización) de la muestra debido a la diversidad estructural que poseía. De los 4 clúster resultantes se seleccionaron aleatoriamente los clústeres 0 y 3 para realizar las pruebas. A estos dos clústeres se les aplicaron los algoritmos de búsqueda y las medidas de evaluación explicadas anteriormente para realizar la reducción del espacio muestral. En este caso se seleccionó el clúster 0 para realizar las pruebas a la red. De los 56 patrones de entrenamiento resultantes de la selección de variables, se utilizaron 45 para realizar el entrenamiento y el 20 % restante se utilizó para desarrollar la validación.
Para la realización de los experimentos se mantuvieron constantes las siguientes variables:
Cantidad de neuronas en la capa de entrada: 20
Cantidad de neuronas en la capa oculta: 14
Cantidad de neuronas en la capa de salida: 1
Velocidad de aprendizaje: 0,03
Momentum: 0,05
Algoritmo de Búsqueda: Enfriamiento Simulado con un valor de alfa igual a 0,7 y Consistencia como Medida de Evaluación.
Los parámetros que se variaron fueron: cantidad de iteraciones y las funciones de transferencias utilizadas en la capa oculta y en la capa de salida. En la tabla 4 se muestran los resultados obtenidos.
Los resultados obtenidos con la muestra SF-1 no superan los de la muestra de cefalosporinas. Sin embargo, se aprecia nuevamente cómo los mejores resultados de la G-Media se obtienen con combinaciones de funciones de transferencia distintas, que en este caso, a diferencia de los resultados de la muestra de cefalosporinas, se logra colocando la función Sigmoidal en la capa oculta y Lineal en la capa de salida. El mejor valor de la G-Media obtenido fue de 0.8660 en el ensayo 1-e, al clasificar el 100 % de los valores activos y el 60 % de los valores inactivos (tabla 5). El peor resultado se le atribuye a la combinación de Sigmoidea-Tangente Sigmoidea para el cual se obtuvo un valor de G-Media de 0.5270; en este caso se clasifican correctamente casi todos los valores activos, no siendo así con los valores inactivos (tabla 6). Esta diferencia se le atribuye al desbalance entre las clases presentes en la muestra (2 activos por cada inactivo).
Por otro lado, se aprecia que con todas las combinaciones de funciones de transferencia utilizadas la red aprende, no hay ningún caso en que el valor de la G-Media resultante sea cero.
Resultados experimentales del entrenamiento del perceptrón para la muestra de reconocimiento facial
De los 70 000 patrones de entrenamiento se utilizaron 60 000 para realizar el entrenamiento y los 10 000 restantes para la validación. Se mantuvieron constantes los siguientes parámetros:
Cantidad de neuronas en la capa de entrada: 20
Cantidad de neuronas en la capa oculta: 14
Cantidad de neuronas en la capa de salida: 1
Velocidad de aprendizaje: 0.003
Momentum: 0.001
Los parámetros que se variaron fueron: cantidad de iteraciones y las funciones de transferencia utilizadas en la capa oculta y de salida. En la tabla 7 se muestran los resultados obtenidos.
De la tabla 7 se puede apreciar que el valor de la G-Media en un entorno local se mantiene relativamente estable; inicialmente aumenta su valor y luego disminuye en cada iteración realizada, lo que sugiere que en este caso no es conveniente aumentar el número de iteraciones, pues la red no aprende más. El mejor valor de la G-Media que se obtiene es de 0.7151 y se logra en un entorno local.
Para conocer la eficiencia de la distribución del algoritmo para entrenamiento local se hicieron pruebas a los ensayos 3, 5 y 6 y al 4, 7 y 8. La comparación de los valores de G-media del algoritmo local y el distribuido, se obtuvo un valor de 0,180 para el primer grupo de ensayos (3,5,6) el cual revela que no es significativa la diferencia entre las respuestas dadas por los algoritmos. No obstante, no se recomienda hacer la distribución en 2 PC, sino en 4, ya que como se aprecia en el ensayo 5 la calidad de la respuesta para 2 PC es peor que la obtenida por el algoritmo en un entorno local, no siendo así en el caso de la distribución en 4 PC.
Por otra parte, en las pruebas a los ensayos 4, 7 y 8 el valor obtenido con el test estadístico fue 0.655, el cual revela que tampoco hay diferencias significativas entre los resultados obtenidos. A pesar de ello no se recomienda hacer la distribución en más de 2 computadoras, ya que como se puede apreciar en el ensayo 8, el valor de la G-Media disminuyó al aumentar el número de iteraciones.
Los resultados de la tabla 8 muestran que el aumento del número de iteraciones en los entrenamientos realizados en un entorno local, no se corresponde con una mejoría en el valor de la G-Media, ya que mejor resultado se logra con 2000 iteraciones, con un valor de G-Media de 0.6998.
Los resultados alcanzados sugieren que, para esta combinación de funciones, no es aconsejable hacer la distribución en más de 2 computadoras, ya que como se puede apreciar de esa misma tabla, el valor de la G-Media disminuyó (ensayo 6). Además, debe valorarse cuidadosamente la distribución en 2 PC, ya que aunque el valor de la G-Media obtenido no difiere considerablemente con el obtenido en un entorno local, la pérdida de dos unidades de precisión, puede resultar relevante para el resultado que se desea obtener.
Un resultado similar se obtiene al realizar las pruebas a los ensayos 4, 7 y 8 en que el valor obtenido con el test de Wilcoxon fue también de 0.180. A pesar de ello, tampoco se recomienda hacer la distribución en más de 2 computadoras, ya que como se puede apreciar, el valor de la G-Media disminuyó (ensayo 8) al pasar de 2 a 4 computadoras.
Resulta interesante la diferencia entre los resultados obtenidos al aplicar diferentes combinaciones de funciones de transferencia a esta muestra. Solo con variar las combinaciones durante el entrenamiento de la red, se mejoraron los resultados en muchos casos, con lo que se demostró nuevamente el peso fundamental que tienen dichas funciones y sus combinaciones en la eficiencia de la respuesta de la red. En la actualidad no se cuenta con una respuesta definitiva de la razón por la cual algunas combinaciones de funciones reportan mejores resultados que otras, lo que será objeto de estudio en futuras investigaciones.
CONCLUSIONES
Se reportan resultados diferentes con la variación de funciones de transferencia en el perceptrón multicapa, medibles por el valor de la G-Media. La sustitución de la combinación clásica Sigmoidea-Sigmoidea por la combinación de funciones Sigmoidea-Lineal o Lineal-Sigmoidea mejora los resultados en el orden del 4.5 al 17 %.
Se desarrollaron dos procedimientos que permiten realizar el entrenamiento del perceptrón multicapa en ambiente local y distribuido respectivamente, variando las funciones de transferencia para realizar el entrenamiento.
Se disminuyó el tiempo de entrenamiento aproximadamente entre un 30-60 % al entrenar la red en un ambiente distribuido. El test estadístico aplicado sugirió que la calidad de los resultados en ambiente distribuido no se afecta de manera apreciable en comparación con los resultados en un entorno local.
REFERENCIAS BIBLIOGRÁFICAS
1. Sremac S, Skrbk B, Onjia A. Artificial neural network prediction of quantitative structure - retention relationships of polycyclic aromatic hydocarbons in gas chromatography. J Serb Chem Soc. 2005;70(11):1291-300.
2. Bolanca T, Cerjan-Stefanovic S, Ukic S, Rogosic M, Lusa M. Application of different training methodologies for the development of a back propagation artificial neural network retention model in ion chromatography. J Chemometrics. 2008;22:106-13.
3. Lendasse A, Ji Y, Reyhani N, Verleysen M. LS-SVM Hyperparameter Selection with a Nonparametric Noise Estimator. In: Artificial Neural Networks: Formal Models and Their Applications-ICANN 2005. Springer Berlin Heidelberg; 2005:625-30.
4. Sutherland JJ, O'Brien LA, Weaver DF. A Comparison of Methods for Modeling Quantitative Structure-Activity Relationships. J Med Chem. 2004;47(22):5541-54.
5. Lucic B, Nadramija D, Basic I, Trinajstic N. Toward Generating Simpler QSAR Models: Nonlinear Multivariate Regression versus Several Neural Network Ensembles and Some Related Methods. J Chem Inf Comput Sci. 2003;43:1094-102.
6. Huang L, Lu H-M, Dai Y. Feature Selection of Support Vector Regression for Quantitative Structure-Activity Relationships (QSAR). The International Conference on Mathematics and Engineering Techniques in Medicine and Biological Sciences (METMBS'03) (Paper ID # : 269ME); 2003.
7. Escalona-Arranz JC, Carrasco-Velar R, Padrón-García JA. Introducción al diseño racional de fármacos. La Habana: Editorial Universitaria, Ministerio de Educación Superior; 2008.
8. Hall M, Frank E, Holmes G, Pfahringer B, Reutemann P, Witten IH. The WEKA Data Mining Software: An Update. ACM SIGKDD Explorations Newsletter. 2009;11(1):10-18.
9. Colectivo de Autores. Tarenal, Plataforma de Tareas Distribuidas, Proyecto Grid. Manual del desarrollador. La Habana: Universidad de las Ciencias Informáticas; 2009.
10. SPSS Inc. SPSS 13 for Windows. 13.0 ed: Copyrigth (c) SPSS Inc. 1989-2004. All rigths reserved; 2004.
11. Universidad Complutense de Madrid. Departamento de Sociología. Introducción al análisis de datos [citado el 15 Marzo 2013] Disponible en: http://www.ucm.es/info/socivmyt/paginas/D_departamento/materiales/analisis_datosyMultivariable/19nparam_SPSS.pdf
12. Heaton J. Introduction to Neural Networks for Java. 2nd ed. St. Louis, Missouri: Heaton Research, Inc.; 2008.
13. Carrasco-Velar R. Nuevos descriptores atómicos y moleculares para estudios de estructura-actividad: Aplicaciones [PhD. Thesis]. La Habana: Universidad de La Habana; 2008.
14. Hernández-Díaz Y. Desarrollo de modelos de clasificación de actividad biológica empleando Máquinas de Soporte Vectorial [Master]. La Habana: Universidad de las Ciencias Informáticas; 2010.
Recibido: 14 de octubre de 2013.
Aprobado: 16 de noviembre de 2013.