My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
MediSur
On-line version ISSN 1727-897X
Medisur vol.15 no.1 Cienfuegos Jan.-Feb. 2017
ARTÍCULO ORIGINAL
Detección del polimorfismo T991T en familias cubanas con diagnóstico clínico de la enfermedad de Wilson
Detection of T991T Polymorphism in Cuban Families with Clinical Diagnosis of Wilson’s Disease
Yulia Clark FeoktistovaI , Caridad Ruenes DomechII , Elsa F. García BacallaoII , Teresa Collazo MesaIII , Hilda Roblejo BalbuenaIII , Estela Morales PeraltaIV , Iliana Clark FeoktistovaIII , Zoe Robaina JiménezIII
I Universidad de Guantánamo, Guantánamo, Guantánamo, Cuba
II Instituto de Gastroenterología, La Habana, La Habana, Cuba
III Centro Nacional de Genética Médica, La Habana, La Habana, Cuba, CP: 90100
IV Facultad de Ciencias Médicas de 10 de Octubre, La Habana, La Habana, Cuba
RESUMEN
Fundamento: la enfermedad de Wilson se caracteriza por la acumulación de cobre, fundamentalmente en el hígado y cerebro. Se transmite con un patrón de herencia autosómico recesivo. La causa molecular que la provoca son las mutaciones en el gen atp7b, que se encuentra en el cromosoma 13. Se han informado en la literatura más de 139 polimorfismos en el gen atp7b, tanto en los exones como en los intrones.
Objetivo: identificar el polimorfismo T991T en pacientes cubanos con diagnóstico clínico de enfermedad de Wilson.
Métodos: se realizó un estudio descriptivo, en el Centro Nacional de Genética Médica, durante el período 2011-2013, que incluyó 35 pacientes con diagnóstico clínico de enfermedad de Wilson. Para la amplificación del fragmento de interés, se utilizó la técnica de Reacción en Cadena de la Polimerasa y para identificar los cambios conformacionales, la técnica de Polimorfismo Conformacional de Simple Cadena, en el exón 13 del gen atp7b. El polimorfismo T991T fue identificado por secuenciación del fragmento analizado y la mutación L708P por digestión enzimática.
Resultados: la frecuencia alélica del polimorfismo T991T en los 35 pacientes cubanos estudiados fue de 10 %. Las manifestaciones hepáticas fueron las más frecuentes (57,1 %). En el 71,4 % de los pacientes que presentaron el polimorfismo T991T se identificó además el polimorfismo K832R. En un paciente se identificó la mutación L708P.
Conclusión: la identificación del polimorfismo T991T permitirá en un futuro inmediato el diagnóstico molecular por métodos indirectos y su correlación con las manifestaciones clínicas presentes en los pacientes con enfermedad de Wilson.
Palabras clave: genes, degeneración hepatolenticular, polimorfismo genético, cuba.
ABSTRACT
Foundation: Wilson’s disease is characterized by the accumulation of copper, mainly in the liver and the brain. It is transmitted with an autosomal recessive inheritance pattern. The mutations of atp7b gene are the cause which produces it, which is found in chromosome 13. In literature it has been informed more than 139 polymorphisms in atp7b gene, both in exons and in introns.
Objective: To identify T991T polymorphism in Cuban patients with clinical diagnosis of Wilson’s disease.
Methods: A descriptive study was done, in the Center of Medical Genetic, during the period of 2011-2013, which included 35 patients with clinical diagnosis of Wilson’s disease. For the amplification of fragment of interest, it was used the Polymerase Chain Reaction technique and to identify conformational changes, the Simple Chain Conformational Polymorphism technique, in exon 13 of the atp7bgene. T991T polymorphism was identified by the sequence of the fragment analyzed and the L708P mutation by enzymatic digestion.
Results: T991T polymorphism allele frequency in the 35 Cuban studied patients was of 10 %. Hepatic manifestations were the most frequent (57.1 %). In the 71.4 % of the patients who presented T991T polymorphism it was identified K832R polymorphism as well. In one patient it was identified mutation L708P.
Conclusion: the identification of T991T polymorphism will allow in the immediate future the molecular diagnosis by indirect methods and its correlations with clinical manifestations present in patients with Wilson’s disease.
Key words: genes, hepatolenticular degeneration, polymorphism, genetic, cuba.
INTRODUCCIÓN
La enfermedad de Wilson (EW, MIM 27790) es un trastorno hereditario que presenta un patrón de herencia autosómico recesivo. Se caracteriza por la acumulación de cobre fundamentalmente en el hígado, cerebro y córnea. El diagnóstico clínico de esta enfermedad resulta complejo.1 Sus manifestaciones hepáticas varían desde la alteración de los niveles séricos de transaminasas hasta una cirrosis descompensada. Las afectaciones a nivel cerebral pueden manifestarse con temblores y llegar a desencadenar enfermedad de Parkinson en algunos casos. Es una enfermedad genética tratable; sin embargo, algunas de las alteraciones que provoca son irreversibles y pueden llevar a la muerte de no atenderse de forma adecuada. La causa molecular que la induce son las mutaciones en el gen atp7b (MIM 606882), que presenta 21 exones y del cual se han reportado más de 500 mutaciones hasta la actualidad.1-2 Además, se han identificado más de 139 polimorfismos que están distribuidos en todo el gen atp7b y en los intrones; los exones más polimórficos reportados son 2, 8 y 16, aunque en el exón 13 se han identificado varios polimorfismos.3 El polimorfismo T991T se encuentra localizado en el exón 13 del gen atp7b. Ha sido identificado en diversas poblaciones, en países como la India, Canadá, Reino Unido e Irán. Se describen pocos estudios moleculares en el gen atp7b en América. En un estudio en el que se analizan pacientes con diagnóstico clínico de la Enfermedad de Wilson en Estados Unidos y Puerto Rico, se identifica el polimorfismoT991T, aunque no se reporta la frecuencia alélica.4 Para la determinación del espectro mutacional y la identificación de polimorfismos en el gen atp7b, se requiere de una adecuada tecnología de cribaje. Una de las técnicas más utilizadas para este propósito es el polimorfismo conformacional de simple cadena, SSCP (del inglés single-strand conformation polymorphism).5 Considerando que en Cuba no existe el diagnóstico molecular de la EW, es objetivo de este estudio identificar los cambios conformacionales en el exón 13 y detectar el polimorfismo T991T en el gen atp7b de pacientes cubanos con diagnóstico clínico de esta enfermedad. La identificación del SNP (polimorfismo de un solo nucléotido) va a constituir una herramienta molecular para el asesoramiento genético adecuado para el individuo afectado y la familia.
MÉTODOS
Se realizó un estudio descriptivo, en el Centro Nacional de Genética Médica, durante el período 2011-2013, que incluyó seis familias cubanas que sumaron 24 pacientes y 11 pacientes independientes, para un total de 35 individuos (14 mujeres y 21 hombres) con diagnóstico clínico de EW, los cuales asistían a la consulta del Instituto Nacional de Gastroenterología. Estos pacientes dieron su consentimiento para participar en la investigación, de acuerdo con los principios éticos de la declaración de Helsinki. Este estudio fue aprobado por el consejo científico y el comité de ética del Centro Nacional de Genética Médica. Las variables analizadas fueron: edad de inicio de las manifestaciones clínicas de EW, provincia de procedencia, frecuencia alélica del polimorfismo T991T (cambio conformacional a para la variante normal y cambio conformacional b para la presencia del polimorfismo T991T en estado heterocigótico), manifestaciones clínicas (hepáticas, neurológicas, asintomáticos), concentración de ceruloplasmina en sangre, resultado de biopsia de tejido hepático, presencia de los polimorfismos T991T y K832R, presencia de la mutación L708P. Todas estas variables fueron registradas en una base de datos en el Centro Nacional de Genética Médica. La evaluación de las manifestaciones clínicas fue realizada por un equipo multidisciplinario (gastroenterólogos, genetistas, neurólogos, bioquímicos), siguiendo los criterios de diagnóstico de la enfermedad. La concentración de ceruloplasmina en sangre se determinó en el Instituto Nacional de Gastroenterología por el método colorimétrico descrito por Ravin.6 Las determinaciones de cobre en tejido hepático se realizaron por espectrofotometría de absorción atómica, en el espectrofotómetro Perkin Elmer, modelo 2380, según Osheim y Ross.7 Los estudios moleculares se realizaron en Centro Nacional de Genética Médica, en el laboratorio de Biología Molecular. Para la detección de cambios conformacionales y la identificación del polimorfismo T991T, se seleccionó el exón 13 del gen atp7b. A todos los pacientes se les tomó una muestra de ADN, mediante el método de precipitación salina8 a partir de 10 ml de sangre periférica con ácido etildiaminotetraacético (EDTA, del inglés Ethylene DiamineTetra Acetic Acid) (56 mg/ml). Las condiciones para la amplificación del exón 13 mediante la técnica de Reacción en Cadena de la Polimerasa (PCR, del inglés Reverse Transcription Polymerase Chain Reaction), fueron: 100ng de ADN, pmoles/ml de cada oligonucléotido del exón 13: (F) 5'-AGT CGC CAT GTA AGT GAT AA-3' y (R) 5'-CTG AGG GAA CAT GAA ACA A-3', 1mM de dNTPs (Boehringer), 10X tampón PCR, 15mM de MgCl2, 1u de Taq polimerasa (Amplicen), en un volumen de 25 µl.9 Posteriormente se realizó la electroforesis SSCP. Se mezcló 3,5µl con una solución de parada de bromofenol azul (0,05 % BFA, 10mM NaOH, 95 % formamida, 20mM EDTA) y 1 µl del producto amplificado, en un volumen final de 7µl. Se aplicó en un gel de acrilamida comercial (GeneGel Excel 12,5/24 Kit). La visualización del ADN se realizó por el método de tinción con plata, siguiendo las instrucciones del juego comercial: kit PlusOne DNA Silver Staining (Amersham Biosciences, 2007). Se procedió a la secuenciación del patrón alterado por SSCP. El secuenciador utilizado fue ALF-Express. Un paso crítico y decisivo fue la purificación del producto de PCR, el cual se realizó con el juego comercial de la Quigen (QIAquick, PCR Purification Kit). Esta investigación es uno de los resultados del proyecto ramal Estudio molecular de familias cubanas con la EW.
RESULTADOS
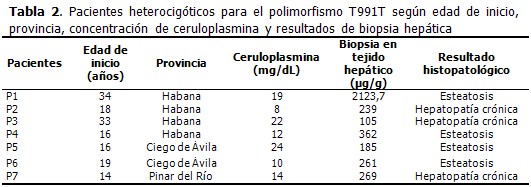
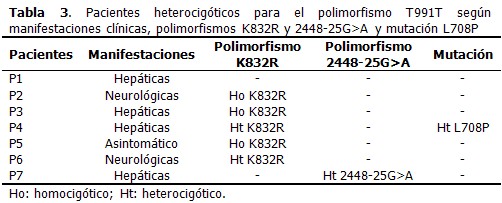
La frecuencia alélica en los 35 pacientes estudiados fue de 10 %. (Tabla 1). El polimorfismo T991T en estado heterocigótico fue identificado en el 20 % de los pacientes. La media de la edad de inicio de EW de los pacientes que resultaron heterocigóticos para el polimorfismo T991T fue 21,4 años. En el 71,4 % de los pacientes los niveles de concentración de ceruloplasmina fueron menores que 20mg/mL (criterio diagnóstico de EW). El 57,1 % de los pacientes presentó una concentración de cobre en tejido hepático mayor que 250µg/g de tejido seco (criterio diagnóstico de EW). En cuanto al resultado histopatológico, la hepatopatía crónica mostró un ligero predominio respecto a la esteatosis. (Tabla 2). Entre las principales manifestaciones clínicas en los pacientes que presentaron el polimorfismo T991T, fueron más frecuentes las hepáticas (57,1 %), seguidas de las neurológicas, identificadas en el 28,6 %. Solo un paciente se mostró asintomático. También se encontraron otros polimorfismos y mutaciones del gen atp7b en estos pacientes. (Tabla 3).
DISCUSIÓN
En Cuba se comienza a realizar la detección de polimorfismos en el gen atp7b en el año 2008. Un paso previo a la búsqueda de mutaciones y polimorfismos en este gen, es la detección de cambios conformacionales. Este polimorfismo no afecta la función de la proteína y se ha identificado en diversas poblaciones con una frecuencia mayor que 1 %. La frecuencia alélica del polimorfismo T991T obtenida en este estudio, es más alta que la reportada por otros países (Tabla 1).8-12 Una posible explicación de este resultado puede ser el origen genético, pero habría que estudiar una población mayor para llegar a juicios más concluyentes. El polimorfismo T991T es consecuencia de un cambio de citosina por guanina, lo cual no provoca el cambio del aminoácido Treonina en la posición 991 de la proteína atp7b en el sexto segmento de transmenbrana. Se obtuvo que en el 71,4 % de los pacientes que presentaron el polimorfismo T991T, se identificó además el polimorfismo K832R, ubicado en el exón 10 del gen atp7b; asimismo, se identificó en un solo paciente el polimorfismo 2448-25G>A. Se esperaba encontrar ambos polimorfismos, pues en estudios anteriores se obtuvo para ellos una frecuencia alélica de 35 % y 8,5 % respectivamente.18,19 También se identificó, en un paciente heterocigótico compuesto para los polimorfismos T991T y K832R, la mutación L708P, localizada en el exón 8 y frecuente en Islas Canarias. Esta mutación ha sido identificada en otros pacientes cubanos con la enfermedad de Wilson.20 Los polimorfismos K832R, T991T y 2448-25G>A pueden ser utilizados en las familias cubanas con algún individuo enfermo, para la realización de los haplotipos y realizar el diagnóstico por el método indirecto. Además, se pueden establecer relaciones entre los haplotipos y las mutaciones, similar a lo realizado por Paradisi y colaboradores en Venezuela.16 Debido al pequeño tamaño de la muestra no es factible establecer una relación entre la presencia del polimorfismo T991T con la edad de inicio, las manifestaciones clínicas y los resultados de la biopsia hepática. Sin embargo, la identificación del polimorfismo T991T, disponible en la Red Nacional de Genética Médica y el Instituto Nacional de Gastroenterología, permitirá en un futuro inmediato el diagnóstico molecular por métodos indirectos y su correlación con las manifestaciones clínicas presentes en los pacientes con EW, por lo que será una herramienta molecular para el asesoramiento genético.
REFERENCIAS BIBLIOGRÁFICAS
1. Kumar SS, Kurian G, Eapen CE, Roberts EA. Genetics of Wilson’s disease: a clinical perspective. Indian J Gastroenterol. 2012;31(6):285-93
2. Kenney SM, Cox DW. Sequence Variation Database for the Wilson Disease Copper Transporter, ATP7B. Hum Mutat. 2007;28(12):1171-77
3. Badenas Orquin C. Avances en el diagnóstico molecular de la enfermedad de Wilson. Gastroenterol Hepatol. 2011;34(6):428-33
4. Shah A, Chernov I, Zhang H, Ross B, Das K, Lutsenko S, et al. Identification and Analysis of Mutations in the Wilson Disease Gene (ATP7B): Population Frequencies, Genotype-Phenotype Correlation, and Functional Analyses. Am J Hum Genet. 1997;61(2):317-28
5. Li XH, Lu Y, Ling Y, Fu QC, Xu J, Zang GQ, et al. Clinical and molecular characterization of Wilson’s disease in China: identification of 14 novel mutations. BMC Med Genet. 2011;12:6
6. Ravin H. An improved colorimetric enzymatic assay of ceruloplasmin. J Lab Clin Med. 1961;58:161-8
7. Osheim DL, Ross PF. Atomic absorption spectrophotometric determination of liver copper: collaborative study. J Assoc Off Anal Chem. 1985;68(1):44-5
8. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res. 1988;16(3):1215
9. Margarit E, Bach V, Gómez D, Bruguera M, Jara P, Queralt R, Ballesta F. Mutation analysis of Wilson disease in the Spanish population identification of a prevalent substitution and eight novel mutations in the ATP7B gene. Clin Genet. 2005;68(1):61-8
10. Cox D, Prat L, Walshe J, Heathcote J, Gaffney D. Twenty-four Novel Mutations in Wilson Disease Patients of Predominantly European Ancestry. Hum Mutat. 2005;26(3):280
11. Zali N, Mohebbi SR, Esteghamat S, Chiani M, Haghighi MM, Hosseini-Asl SM. Prevalence of ATP7B gene mutations in Iranian patients with Wilson disease. Hepat Mon. 2011;11(11):890-4
12. Simsek Papur O, Akman SA, Cakmur R, Terzioglu O. Mutation analysis of ATP7B gene in Turkish Wilson disease patients: Identification of five novel mutations. Eur J Med Genet. 2013;56(4):175-9
13. ?im?ek Papur Ö, A??k Akman S, Terzio?lu O. Clinical and genetic analysis of pediatric patients with Wilson disease. Turk J Gastroenterol. 2015;26(5):397-404
14. Curtis D, Durkie M, Balac P, Sheard D, Goodeve A. Peake I, Quarrell O, Tanner S. A study of Wilson diseasemutations in Britain. Hum Mutat. 1999;14:304-311
15. Ljubić H, Kalauz M, Telarović S, Ferenci P, Ostojić R, Noli MC, et al. ATP7B gene mutations in Croatians patients with Wilson Disease. Genet Test Mol Biomarkers. 2016;20(3):112-7
16. Paradisi I, De Freitas L, Arias S. Most frequent mutation c.3402delC (p.Ala1135GlnfsX13) among Wilson disease patients in Venezuela has a wide distribution and two old origins. Eur J Med Genet. 2015;58(2):59-65
17. Aggarwal A, Chandhok G, Todorov T, Parekh S, Tilve S, Zibert A, et al. Wilson disease mutation pattern with genotype-phenotype correlations from Western India: confirmation of p.C271 as a common Indian mutation and identification of 14 novel mutations. Ann Hum Genet. 2013;77(4):299-307
18. Clark Y, Ruenes C, García E, Collazo T, Robaina Z, Castañeda C, et al. Identificación del polimorfismo K832R en pacientes con diagnóstico clínico de la enfermedad de Wilson. Revista Habanera de Ciencias Médicas [revista en Internet]. 2013 [citado 12 Feb 2015];12(2):[aprox. 6p]. Disponible en: http://www.bvs.sld.cu/revistas/rhab/vol_12_2_13/rhcm06213.htm
19. Clark Y, Ruenes C, García E, Collazo T, Robaina Z, Roblejo H. Identificación del polimorfismo c.2448-25G>A en pacientes con diagnóstico clínico de la enfermedad de Wilson. Medisur [revista en Internet]. 2015 [citado 23 Nov 2016];13(5):[aprox. 9p]. Disponible en: http://scielo.sld.cu/scielo.php?script=sci_arttext&pid=S1727-897X2015000500008
20. Clark Y, Collazo T, Ruenes C, García E, Robaina Z, Fragoso T, et al. Detection of conformational shifts and mutations in exon 8 from the atp7b gene in Cuban Wilson’s disease patients. Biotecnol Apl [revista en Internet]. 2011 [citado 23 Nov 2016];28(2):[aprox. 12p]. Disponible en: http://scielo.sld.cu/scielo.php?script=sci_arttext&pid=S1027-28522011000200004&lng=es
Recibido: 05 de febrero de 2016.
Aprobado: 09 de enero de 2017.
Yulia Clark Feoktistova. Máster en Ciencias en Bioquímica. Mención Biología Molecular. Investigador Agregado. Profesor Asistente Correo electrónico: yuliacf@cug.co.cu