Introducción
El cáncer es la principal barrera para incrementar la esperanza de vida en todo el mundo y una de las causas principales de muerte en personas del rango de edad menor de 70 años en 112 naciones y clasifica de tercero en más de 23 países [1]. El cáncer de pulmón es el segundo que más casos nuevos aporta, siendo el 18% de todas las muertes por cáncer.
Los nódulos pulmonares son masas de tejido pequeñas, usualmente de 5 a 30 milímetros [2], con forma aproximadamente esférica. Son potencialmente una manifestación inicial de cáncer de pulmón y su temprana detección facilita el tratamiento, mejorando las probabilidades de supervivencia del paciente [3].
Las imágenes médicas son una herramienta fundamental para el diagnóstico y el tratamiento de diversas patologías, ya que permiten visualizar el interior del cuerpo humano de forma no invasiva y con gran precisión. Entre las diferentes técnicas de imagen, la radiografía digital (Chest X-Ray, CXR) se destaca por su rapidez, su bajo costo y su amplia disponibilidad [4]. La CXR consiste en la obtención de imágenes bidimensionales del tórax mediante el uso de rayos x, que atraviesan los tejidos y se proyectan sobre un detector digital. La CXR puede detectar lesiones pulmonares.
Sin embargo, la interpretación de las imágenes de CXR no es una tarea sencilla, ya que requiere de un alto nivel de conocimiento y experiencia por parte del profesional médico. Además, la subjetividad, la variabilidad y la fatiga pueden afectar la precisión y la consistencia del diagnóstico, debido a que la región del tórax es muy compleja al superponerse tejidos y estructuras con un nivel de atenuación de los rayos x muy diverso. Esto conduce con frecuencia a diagnósticos erróneos con presencia de falsos positivos y negativos. Por este motivo, se han desarrollado los sistemas CAD (Computer-Aided Diagnosis), que son programas informáticos que analizan las imágenes de CXR mediante algoritmos matemáticos y proporcionan información adicional al médico, como la detección, la localización, la segmentación y la clasificación de las lesiones [5].
En un comienzo los CAD consistían en reglas bien definidas con varias fases de procesamiento de píxeles de bajo nivel y modelado matemático [6], [7]. Luego se fueron incorporando de forma gradual algoritmos de aprendizaje automático (Machine Learning, ML) como la máquina de soporte vectorial (Support Vector Machine, SVM) [8], con importantes resultados en cuanto a efectividad.
En los últimos años, gracias a la creciente popularidad de los sistemas que incorporan inteligencia artificial y el aumento de los recursos de cómputo, la capacidad de almacenamiento y la disponibilidad de datos, han hecho posible la incorporación del aprendizaje profundo (Deep Learning, DL) a través de las redes neuronales de convolución [9]. Los algoritmos de redes neuronales usualmente envuelven un gran número de parámetros, valores de activación y de gradiente. Cada uno de ellos debe ser actualizado en cada paso del proceso de entrenamiento de la red. Para esto se requiere de suficiente cantidad de información, lo cual suele exceder las capacidades de una computadora de escritorio tradicional. Para ello se desarrollaron técnicas avanzadas de procesamiento por lotes, en arquitecturas paralelas y distribuidas, implementadas sobre unidades de procesamiento gráfico (Graphics Processing Units, GPU) y unidades especializadas en procesamiento de tensores (Tensor Processing Units, TPU) [10]. Por esta vía, puede reducirse el consumo de recursos al disminuir la cantidad de datos de entrenamiento. Sin embargo, entrenar durante más épocas (cantidad de iteraciones completas sobre el conjunto de entrenamiento), sobre una cantidad de datos reducida, determina que la red solo memorice los datos y no los generalice. Esto se conoce como sobreajuste del modelo.
Los sistemas CAD no pretenden reemplazar al médico, sino asistirlo en su labor, facilitando su trabajo y mejorando su rendimiento. Pueden aumentar la sensibilidad y la especificidad del diagnóstico, reducir el tiempo y el costo del proceso, disminuir los errores humanos y mejorar la calidad de la atención al paciente. Además, pueden contribuir a la formación y la educación de los médicos, así como a la investigación y el desarrollo de nuevas técnicas de imagen.
Existen numerosas arquitecturas, métodos y enfoques que han demostrado ser especialmente efectivos en determinadas tareas y para extraer las características de algunos tipos de datos específicos. Entre estos se destaca la red YOLO (You Only Look Once) [11]. Este método incorpora una etapa de detección de objetos en tiempo real, que utiliza una red neuronal convolucional para dividir la imagen en regiones y predecir las coordenadas y probabilidades de existencia de los objetos en cada región. YOLO tiene la ventaja de ser rápido, preciso y robusto frente a diferentes condiciones de iluminación, tamaño y forma de los objetos.
Varios estudios han aplicado YOLO en tareas de detección a partir de imágenes médicas: como la mamografía [12], el estudio del melanoma [13], enfermedades dentales [14] y para nódulos pulmonares [15] [18], alcanzándose precisiones y sensibilidades por encima del 90 % en simulaciones de laboratorio y estudios con pacientes, para la data interna de validación. Estos resultados pueden ser un indicador de la eficacia del método en la detección de objetos anormales en tareas biomédicas, así como su potencial para convertirse en un enfoque novedoso, capaz de realizar detección y clasificación de enfermedades con buen desempeño en rutina clínica, lo que podría tener implicaciones relevantes también para el diagnóstico precoz del cáncer pulmonar y la reducción de biopsias innecesarias. No obstante, se requiere de múltiples estudios, en condiciones de adquisición muy diversas, con datas externas al entrenamiento del modelo, para demostrar la eficacia del método y su posible poder de generalización. [16], [17]
El objetivo de este trabajo ha sido estudiar el potencial para la tarea de un sistema de diagnóstico automatizado, con un diseño basado en el método YOLO, para detectar nódulos pulmonares a partir de rayos x de tórax. La contribución científica radica en que se proporciona una metodología que incluye la construcción de una arquitectura para procesar y segmentar las imágenes 2D de rayos x de tórax y detectar nódulos pulmonares sobre estas, a partir del empleo de redes neuronales convolucionales, así como un correcto método de evaluación del modelo, que permite detectar los sesgos de entrenamiento, su lejanía del poder de generalización y las posibles causas de las clasificaciones erróneas.
Metodología
Software, hardware e implementación general
Se utilizó el lenguaje de programación Python y los marcos de trabajo de aprendizaje automático TensorFlow y PyTorch. Además, se recurrió al uso de las herramientas que disponen los paquetes de OpenCXR, Scikit-Image y Albumentations. La aplicación de la arquitectura YOLO estuvo apoyada en el proyecto de Ultralytics en Github, YOLOv5 [18], una implementación que mantiene la idea original detrás de la arquitectura YOLO e incorpora los últimos avances en los campos del aprendizaje automático y el aumento de datos. Para el desarrollo del proyecto se utilizó una computadora de escritorio con un procesador Intel® Core™ i7-8700 3,2 GHz, una tarjeta gráfica NVIDIA GeForce® RTX 3070 con 8 GB GDDR6, 32 GB (2 x 16 GB) DDR4 a 3200MHz de RAM, una placa base MSI B365M PRO-VH y almacenamiento SSD de 128GB M.2 SATA + HDD 1TB (7200RPM).
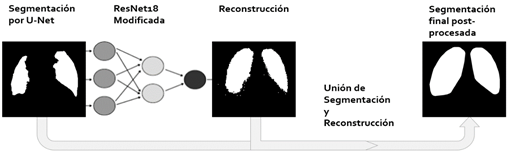
La metodología propuesta en esta investigación consta de tres etapas fundamentales: segmentación del área pulmonar, reconstrucción de dicha área y detección de los nódulos pulmonares. En cada etapa está involucrada una red neuronal diferente, nombrada igual que la respectiva etapa.
La red de segmentación emplea el modelo pre-entrenado contenido en la librería OpenCXR [19], que es una colección de algoritmos y utilidades de rayos x de tórax, de código abierto, mantenida por el Grupo de Análisis de Imágenes Diagnósticas del Centro Médico de la Universidad Radboud, Nijmegen, en los Países Bajos. La red de reconstrucción utilizada fue ResNet18 [20], modificada en sus últimas capas, para tener como salida imágenes. Esta utilizó como entrada el resultado de la red de segmentación y como referencia imágenes correctamente segmentadas previamente por expertos, de esta forma es capaz de corregir los errores de la etapa previa. Por último, la red de detección se basa en la arquitectura YOLO. Fue entrenada con las imágenes reconstruidas y la localización anotada de los nódulos que traen las bases de datos.
Conjuntos de datos utilizados
En la tarea del entrenamiento de la red de detección con YOLO se emplearon dos conjuntos de radiografías anotadas, uno para la fase de entrenamiento y otro, de diferente origen, para evaluar su capacidad de generalización. El conjunto de datos de entrenamiento consistió en la combinación de las bases de datos (BD) ChestX-ray14 [21], PadChest [22], Openi [23] y VinBigData ChestX-ray [24]. Para realizar la prueba externa se utilizó la BD JSRT (Japón) [25]. También se agregaron radiografías de pacientes sanos a ambos conjuntos, tomadas de la BD VinBigData ChestX-ray [24], con el objetivo de reducir los falsos negativos en los resultados. En la Tabla 1 se recogen las características generales de todas las BD utilizadas.
Tabla 1 Bases de datos utilizadas
| Base de datos | Total de imágenes sin nódulos | Total de imágenes con nódulos | Uso |
|---|---|---|---|
| VinBigData | 14174 | 826 | Entrenamiento y validación interna |
| ChestX-ray14 | 2348 | 617 | |
| PadChest | 1196 | 314 | |
| Openi | 205 | 54 | |
| Total | 17923 | 1811 | |
| JSRT | 93 | 153 | Prueba externa |
La BDs VinBigData, PadChest y Openi están conformadas por imágenes de diferentes resoluciones, siempre mayores que 1280 x 1280 píxeles y niveles de gris entre 12 y 16 bits, mientras que la BD ChestX-ray14 posee imágenes de 1024 x 1024 píxeles de 8 bits. En cuanto a JSRT, las imágenes digitalizadas tenían una matriz de 2048 × 2048 píxeles y niveles de gris de 12 bits de profundidad. Adicionalmente, se utilizó la BD Lung Segmentation Data Kit de CSIRO[26], que cuenta con 770 radiografías y sus máscaras de segmentación, para entrenar la red de reconstrucción.
Preparación de las imágenes para el entrenamiento con YOLOV5
Primero se extrajeron de todas las BD las anotaciones de las imágenes completas. En segundo lugar, las imágenes fueron pre-procesadas y segmentadas con la red U-Net [27]. Luego se entrenó una red de reconstrucción, que se utilizó para reconstruir las máscaras de segmentación originalmente obtenidas, extrayendo así la región pulmonar, que sería únicamente la analizada por la red de detección de nódulos, para disminuir los posibles sesgos de aprendizaje [28].
Las imágenes completas fueron extraídas de las BD, redimensionadas a 1280x1280 píxeles, buscando conservar la mayor cantidad de detalles posibles, y ajustadas a 256 niveles de grises (8 bits de profundidad). Seguidamente, las anotaciones de las imágenes con nódulos fueron re-escritas al formato YOLO [11, 12]. Esto implica que los valores de sus coordenadas fueron recalculados para la resolución escogida y colocados en la siguiente estructura: número de clase, coordenada x del centro, coordenada y del centro, ancho y largo.
En la segunda etapa, debido a características deficientes, como el bajo brillo de muchas imágenes, se procedió a normalizar las mismas. Para ello se utilizó el algoritmo de estandarización de intensidad, a partir de las bandas de energía de la librería OpenCXR [19]. La Figura 1 muestra estos pasos.
Para segmentar el área pulmonar se recurrió nuevamente a la librería OpenCXR, específicamente a su red neuronal convolucional (Convolutional Neural Network, CNN) pre-entrenada basada en la arquitectura U-Net [27]. Las imágenes de entrada a la red tuvieron en este caso una resolución de 512 x 512 píxeles, que es la admitida por dicha red.
A partir de las máscaras de segmentación obtenidas a la salida de U-NET se pasó a la fase de reconstrucción de imagen. El primer paso consistió en tratar con el problema de los falsos positivos a partir de operaciones morfológicas. Las técnicas aplicadas consistieron en erosión, dilatación, un filtro de área que mantiene solamente las mayores áreas y la operación de cerrado morfológico. Los elementos estructurantes utilizados en las operaciones morfológicas fueron: una cruz de tamaño 11x11 píxeles en la erosión, un disco 10x10 píxeles en la dilatación y un disco 9x9 píxeles en el cerrado. Estos parámetros lograron los mejores resultados entre el conjunto de parámetros probados. La Figura 2, muestra el resultados de estos pasos.
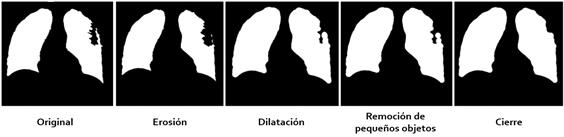
Figura 2 Etapa de reconstrucción de imágenes a partir de la segmentación inicial. De izquierda a derecha se aprecia máscara de segmentación original, erosión, dilatación, remoción de pequeños objetos y cierre.
El siguiente paso está dirigido a paliar la problemática de los falsos negativos causados por regiones severamente afectadas por opacidades o consolidaciones. Cómo solución para estos casos se procedió a entrenar una segunda CNN, para realizar una reconstrucción sobre las regiones afectadas. El entrenamiento de dicha red utilizó como datos de entrada las máscaras inicialmente segmentadas y post-procesadas (Figura 1), contra máscaras de segmentación reales construidas por expertos de la BD Lung Segmentation Data Kit de CSIRO.
La arquitectura de la CNN de reconstrucción consistió en una modificación de la ResNet18 [29] para generar una máscara reconstruida a partir de la segmentación inicial obtenida en el paso anterior. Debido a las características de esta red, las imágenes de entrada fueron redimensionadas a 128x128 píxeles. La salida de la red está dada luego del último bloque de ResNet18, por una capa totalmente conectada de 65 536 neuronas, cuya salida es reordenada a 256x256 píxeles y pasada por una operación de agrupación máxima de 2x2 píxeles con stride de 2 píxeles, que reduce a la mitad la resolución.
La segmentación final consistió en la unión de la segmentación inicial y la reconstrucción generada, pasada nuevamente por un proceso de post-procesado similar al anterior, en este caso para pulir las irregularidades añadidas en el proceso reconstrucción, pero utilizando como parámetros discos de 15x15 píxeles para la erosión, 25x25 píxeles para la dilatación y se añadió un paso adicional, que consistió en rodear las áreas resultantes con el menor polígono convexo posible. La Figura 3 muestra el resultado de estos pasos.
Finalmente, se descartaron los nódulos que se localizaban fuera de la máscara resultante, al igual que las imágenes cuyas máscaras no alcanzaron la calidad requerida. Utilizando las máscaras válidas se conformaron dos conjuntos de datos, con las imágenes médicas originales y las normalizadas.
Antes de pasar a la etapa de entrenamiento de la red de detección con YOLOv5 para la detección de nódulos pulmonar, se emplearon diversas técnicas de aumento de datos, con el fin de disminuir sesgos de aprendizaje y evitar el sobreajuste del modelo [28]. Estas fueron, la variación de HSV (del inglés Hue, Saturation, Value - matiz, saturación, brillo), transformaciones geométricas, mosaico y mezclado. En cuanto a las variaciones en el HSV, se escogieron valores de forma aleatoria dentro de un margen comprendido desde 0 hasta un máximo determinado, fracciones de 0,015 para el matiz, 0,7 para la saturación y 0,4 para el brillo. En cuanto a las transformaciones geométricas, también se realizaron aleatoriamente dentro de márgenes específicos, estos fueron 45 grados para la rotación, 10 grados de distorsión del eje para simular ángulos de percepción y fracciones del tamaño de la imagen de 0,2 para la traslación y el escalado. Estos valores, tanto en variaciones HSV como en transformaciones geométricas, fueron escogidos por prueba y error. En la mitad de los casos se aplicó la técnica de mosaico incorporada en YOLOv5, que combina 4 imágenes de entrenamiento en una sola. Esto permite que el modelo aprenda a identificar objetos a una escala más pequeña de lo normal. Por último, se aplicó el mezclado con una probabilidad del 10%, técnica de aumento de datos que genera combinaciones ponderadas de pares de imágenes aleatorias. La Figura 4 muestra un resumen de todos los pasos seguidos hasta aquí para procesar las imágenes antes de comenzar el entrenamiento con el modelo del método YOLOv5.
Figura 4 Últimas fases de la preparación de las imágenes para los conjuntos de datos de entrenamiento y validación.
Entrenamiento, validación y prueba de los modelos
Para obtener el mejor modelo se realizaron varios ensayos poniendo a prueba los dos conjuntos conformados, ejecutando el entrenamiento desde cero y utilizando pesos pre-entrenados. En todos los casos, el conjunto de entrenamiento se dividió en dos grupos, el 80 % para entrenar los modelos y el 20 % para validarlos. Se utilizaron tres estrategias de entrenamiento: en la primera se emplearon imágenes segmentadas y normalizadas, en la segunda solo segmentadas y en la tercera segmentadas, pero incluyendo el área del mediastino.
El entrenamiento previo se realizó durante 300 épocas con los pesos originales de YOLOv5. El uso de pesos pre-entrenados permitiría que la CNN aprenda solamente las características de alto nivel (correspondientes a estructuras anatómicas) en tanto se aprovecha del modelo pre-entrenado el aprendizaje de estructuras más sencillas como líneas, bordes, entre otras. Por tanto, estos pesos solo fueron cargados en las primeras capas de la arquitectura.
A la hora de aplicar la transferencia de aprendizaje, los modelos fueron entrenados durante algunas épocas manteniendo los pesos pre-entrenados congelados, luego se descongelaron las capas reutilizadas y se redujo la tasa de aprendizaje para evitar dañar los pesos reutilizados y se continuó entrenando para realizar un ajuste fino. Para las pruebas donde se realizó el entrenamiento, desde cero, fue necesario reproducirlo durante 600 épocas para lograr apreciar el sobreajuste.
Otra especificación utilizada en la implementación, fue el empleo del optimizador de descenso de gradientes estocástico (Stochastic Gradient Descent, SGD) con momentum, debido a su calidad de convergencia [30]. Las funciones de pérdidas de YOLOv5 consisten en la suma de tres partes, las pérdidas de clase y objetividad, que emplean la función de pérdidas de entropía cruzada binaria (Binary Cross Entropy Loss) y las pérdidas de localización, donde se utilizó la función de pérdidas de intercepción sobre unión.
Para determinar los valores óptimos para todos los hiperparámetros: tasa de aprendizaje inicial y final, y el momentum, se utilizó la herramienta Hyperparameter Evolution. En este caso se utilizó la mutación con un 80% de probabilidad y varianza de 0,04. Debido a que es imposible albergar todas las imágenes en la memoria de la tarjeta gráfica, el entrenamiento se realizó por lotes. Se utilizó un tamaño de lotes de 16 para todos los entrenamientos [30].
Se utilizó el método Grad-CAM (Gradient-weighted Class Activation Mapping) para visualizar las regiones de las imágenes donde los modelos prestaron mayor atención para detectar los nódulos pulmonares [31]. Tras cada época, el modelo fue validado. Las métricas utilizadas fueron precisión, sensibilidad, el valor F1, AP@0,5 y AP@0,5:0,95, donde AP@0,5 y AP@0,5:0,95 son la precisión promedio (Average Precision, AP). Esto no es más que encontrar el área bajo la curva de precisión contra sensibilidad. Por tanto, AP@0,5 es el área para valores de Intersección sobre Unión (IoU) de 0,5, mientras que AP@0,5:0,95 es la media de las áreas para varios valores de IoU desde 0,5 a 0,95 [30].
Una vez completado el entrenamiento, se comprobó la capacidad de generalización del mejor modelo obtenido, mediante una prueba externa. En este caso, se ejecutaron las mismas métricas descritas sobre el conjunto externo de prueba, nunca antes visto por la red.
Resultados y discusión
Resultados de cada estrategia de entrenamiento y validación de los modelos
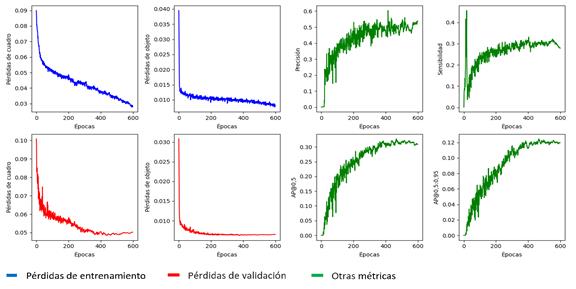
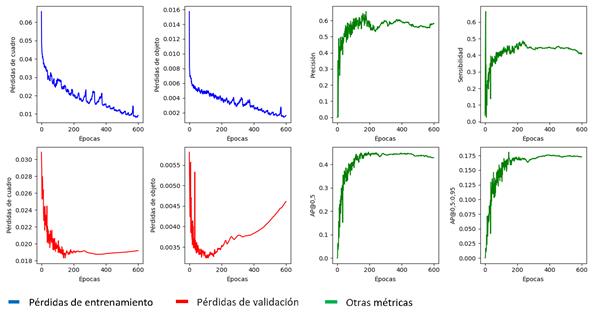
En un primer experimento, se entrenó la red de detección a partir de la segmentación de las imágenes normalizadas. Esto obliga a la red neuronal a emitir una clasificación basándose solo en la región pulmonar normalizada. La Figura 5 muestra el desarrollo del entrenamiento, donde el color azul representa las pérdidas de entrenamiento, en rojo las pérdidas de validación y en verde las métricas. Puede observarse que, aunque el comportamiento de las pérdidas en el entrenamiento continúa disminuyendo, en la validación se observa una tendencia a estancarse o empeorar, lo cual es evidencia de sobre-ajuste [28]. Esto se refleja directamente en las métricas sobre el conjunto de validación, que no muestran buenos resultados. El mejor modelo fue seleccionado a partir de la AP@0,5:0,95 en un 90% y el AP@0,5 en un 10% en la etapa de validación. La selección se efectuó en la época 467 y tras su validación alcanzó una sensibilidad máxima del 68% y AP@0,5 de 0,328.
También se apreció, cómo la taza de falsos positivos tiende a aumentar al aumentar la sensibilidad, mientras que la tasa de verdaderos negativos tiende a disminuir. Una vez que la sensibilidad máxima es superada, el número de fallos cometidos por el sistema se dispara.
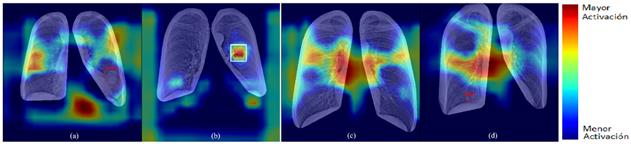
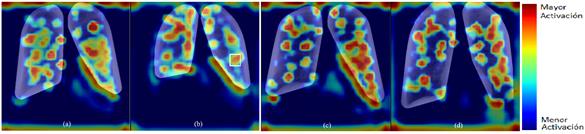
Con el fin de valorar la calidad de la red al desempeñar la tarea, se aplicó el método de Grad-CAM, para verificar qué regiones fueron consideradas más importantes para determinar el resultado. En la Figura 6 se muestra la aplicación de Grad-CAM, donde los colores cálidos indican las regiones de mayor peso en la clasificación.
Se puede apreciar en la primera imagen (a) que la red detecta un nódulo, lo cual es cierto (VP). Sin embargo, la red destaca sitios donde en realidad no hay lesión, y que además están fuera de la región pulmonar. Este resultado evidencia que existen sesgos aprendidos por el modelo [28], [32]. En la segunda imagen (b) existe realmente un nódulo en ella, pero no ha sido detectado por la red, lo cual constituye un falso negativo (FN). A pesar de que el área del nódulo sí presenta colores cálidos, otras áreas ajenas a este, también son consideradas importantes, aunque realmente solo añaden confusión a la decisión final de la red.
En la tercera imagen (c) encontramos pulmones sanos. Esta imagen es un verdadero negativo (VN). La mayor parte de las zonas activas se encuentran fuera del área pulmonar, lo que podría justificar la decisión de la red, aunque también existe una porción del pulmón con colores cálidos, lo que indica que persiste la conducta sesgada de la red [32]. En cambio, en la cuarta imagen (d), la red ha predicho un nódulo, cuando en realidad no existe, siendo este entonces un falso positivo (FP). Los colores cálidos abarcan la región central y parte del pulmón que contiene la lesión, y realmente pocas áreas externas a los pulmones están destacadas. En conclusión, el experimento actual, donde el conjunto de entrenamiento está conformado por radiografías normalizadas y segmentadas, no aportó resultados satisfactorios.
Debido al aprendizaje por atajos, evidenciado en el experimento anterior, se evitó la normalización de las imágenes. La Figura 7 muestra el desarrollo completo del entrenamiento para igual escala de colores que en el experimento anterior. Puede observarse que, aunque el comportamiento de las pérdidas en el entrenamiento continúa disminuyendo, en la validación tampoco se obtienen buenos resultados, aunque son superiores al experimento anterior. No obstante, se aprecian pérdidas de objetos, prueba de la presencia de sobre-ajuste [28]. Esto se refleja además directamente sobre las métricas, para el conjunto de validación, que no muestran buenos resultados. El mejor modelo fue seleccionado a partir de la AP@0,5:0,95 en un 90% y el AP@0.5 en un 10% en la etapa de validación. El mejor resultado para estas métricas se logró en la época 147 y tras su validación alcanza una sensibilidad máxima del 74% y AP@0,5 de 0,45.
Se observa un comportamiento similar al caso anterior, pero con una clara mejoría. Una vez más, al superar la sensibilidad máxima, el número de fallos cometidos por el sistema se dispara.
En la Figura 8 se muestra la aplicación de Grad-CAM, donde los colores cálidos indican regiones de mayor peso. Se puede apreciar en la primera imagen (a) de la izquierda que la red detecta un nódulo (VP), aunque también se resaltan sitios donde no hay lesión y que están fuera de la región pulmonar. En cuanto a la segunda imagen (b) existe realmente un nódulo en ella, pero no ha sido detectado por la red, por lo que constituye un falso negativo (FN). A pesar de que el área del nódulo presenta colores cálidos, estos tienen menor intensidad que otras áreas ajenas a este. En la tercera imagen (c) se presentan pulmones sanos (VN), pues no fue detectada ninguna lesión. En cambio, en la cuarta imagen (d), la red ha predicho un nódulo, cuando en realidad no existe. En todos los casos, los mapas obtenidos con Grad-CAM son más concretos y comprensibles que en el experimento anterior, lo que justifica la mejora en los resultados. Aun así, se observan sesgos, siendo los más claros, los localizados en el área del mediastino, el hemi-diafragma y el corazón, entre ambos pulmones. Al realizar una búsqueda detallada en esta zona, se identificó el posible problema como una pérdida de información de los nódulos del área durante el proceso de segmentación.
Debido a los sesgos identificados en el experimento anterior y la determinación de su causa posible, se decidió entrenar el modelo con imágenes segmentadas, incluyendo dicha área.
La Figura 9 muestra el desarrollo completo del entrenamiento. Las pérdidas en el entrenamiento continúan disminuyendo, mientras que en la validación persiste sobre-ajuste [28]. Esto se refleja directamente en las métricas, que muestran caídas abruptas, más notables en la curva de sensibilidad. El mejor modelo fue seleccionado a partir de la AP@0,5:0,95 en un 90% y el AP@0,5 en un 10% en la etapa de validación. El mejor resultado para estas métricas se logró en la época 169 y tras su validación final alcanza una sensibilidad máxima del 87% y AP@0,5 de 0,654. Este fue el modelo de mejor desempeño de todos los probados.
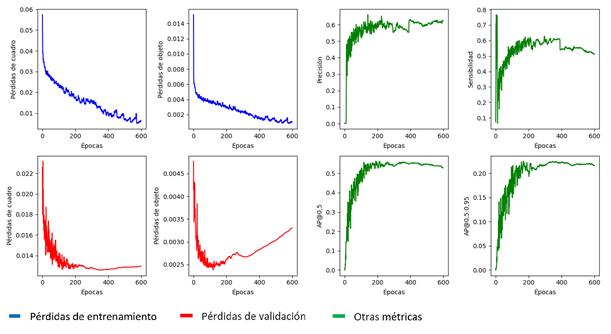
Figura 9 Comportamiento del entrenamiento del modelo para imágenes segmentadas incluyendo el área del mediastino.
En este caso, los resultados para la tasa de falsos positivos y la tasa de verdaderos negativos son superiores a los experimentos precedentes. Debido a que el último punto graficado se encuentra a una sensibilidad del 80% y la sensibilidad máxima del modelo es 87%, no se manifiesta el aumento de falsos positivos tan evidentemente como en el resto de experimentos.
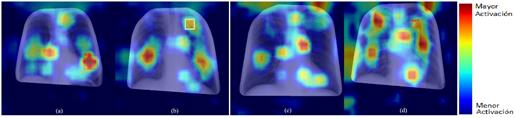
Con el fin de valorar la calidad de la red al desempeñar la tarea, se aplicó el método de Grad-CAM. En la Figura 10 se muestra un ejemplo de su aplicación de Grad-CAM.
En la primera imagen (a) la red detecta correctamente la lesión presente (VP), el motivo queda claro tras ver la intensidad del área de la lesión en relación con otras áreas levemente destacadas. En cuanto a la segunda imagen (b), sucede lo contrario, la red no identifica el área del nódulo como tal (FP), aunque dicha área está resaltada, es relativamente mucho menos intensa que otras regiones. En la tercera imagen (c) encontramos un verdadero negativo (VN), pues no fue detectada ninguna lesión. En cambio, en la cuarta imagen (d), la red ha predicho un nódulo, cuando en realidad no existe, siendo este entonces un falso positivo (FP). En ambos casos, se destacan varias zonas, fuera de la región pulmonar.
Luego de analizar los resultados de la aplicación del algoritmo de Grad-CAM, se evidencia que el modelo resultante de este experimento posee una mejor comprensión de las características de los nódulos, lo que le permite alcanzar mejores resultados en sus métricas. Habiendo llegado a este punto, se pueden realizar algunas comparaciones de los presentes resultados con estudios previos de la literatura científica donde se utiliza otro tipo de red convolucional para igual propósito, así como con respecto a la evaluación visual que puede realizar el ojo humano experto en esta tarea. Las Tablas 2 y 3 muestran estos resultados para un estudio que emplea un conjunto de bases de datos muy similar al nuestro, este artículo comparte la base de datos VinBigData que constituye casi el 50% de las imágenes con presencia de nódulos.
Tabla 2 Comparación de resultados entre el estudio [34] y el presente trabajo, en cuanto al área bajo la curva de precisión vs. sensibilidad
| Arquitectura | Prueba | AUPRC-val |
|---|---|---|
| ResNet-34 | 16 parches | 0,669 |
| ResNet-34 | 6 parches | 0,720 |
| YOLOv5 | Región pulmonar segmentada incluyendo el área del mediastino | 0,654 |
Tabla 3 Comparación entre el rendimiento humano según [34], el estudio [33] y el presente trabajo en cuanto a sensibilidad.
| Método | Sensibilidad-val (%) |
|---|---|
| Tres radiólogos con 30, 15 y 10 años de experiencia | 65 |
| ResNet-34 | 81 |
| YOLOv5 | 87 |
Resultados de la prueba con data externa
El mejor modelo obtenido (con la estrategia de segmentación que incluye el área del mediastino) fue sometido a una prueba externa, donde se utilizó una data nunca vista por el modelo. Se obtuvo que para una confianza mayor de 76,3% el modelo posee precisión del 100%, valor de F1 de 42% para una confianza de 24%, sensibilidad máxima de 68% y un AP@0,5 de 0,347. Estos resultados, por debajo de los obtenidos en la etapa de validación, sugieren que la cantidad de datos de las BD empleadas para el entrenamiento no evidencian ser suficientes para eliminar completamente el sobre-ajuste del modelo [28].
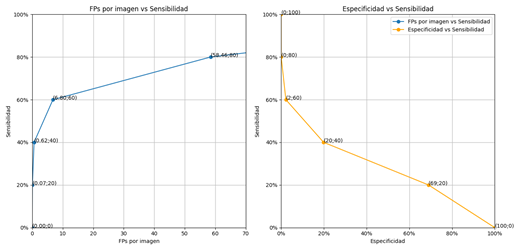
El deterioro de las métricas también es apreciable en el aumento de la tasa de falsos positivos y la disminución de verdaderos negativos como muestra la figura, Figura 11.
Discusión general
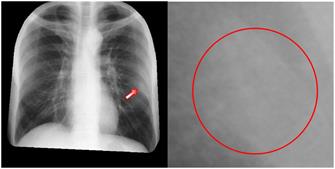
La sensibilidad de los radiólogos sobre imágenes radiográficas digitales suele estar entre el 45% y el 68% [35]. En el caso en que los nódulos sean menores de 10 mm, la sensibilidad es del 29%. Esto no solo está determinado por el tamaño del nódulo, sino también por su bajo contraste, la superposición de estructuras anatómicas, su localización, el cansancio visual y los límites propios del sistema visual humano [35]. En ello reside la importancia del empleo de sistemas automatizados como una segunda opinión o auxilio del radiólogo.
La Figura 12 muestra un nódulo detectado por el mejor modelo obtenido en la presente investigación, que es extremadamente difícil de visualizar por el ojo humano entrenado, a causa de su pequeño tamaño, bajo contraste y la superposición con una costilla. Al observar el zoom de la derecha, queda claro que la diferencia de contraste es prácticamente indistinguible. Este ejemplo ilustra las potencialidades del modelo.
Las dos primeras estrategias de aprendizaje no fueron exitosas. Los modelos aprenden por atajos [28], [37] y el Grad-CAM evidencia sesgos de aprendizaje, al marcar como positivas o negativas zonas que están fuera de la región de interés seleccionada. Se concluye que estos sesgos se deben: en el caso de la primera estrategia a que, aunque la normalización mejora la calidad general de la imagen, puede haber provocado la pérdida de características particulares de los nódulos, que son de gran importancia para el correcto aprendizaje de la red [33]. En el caso de la segunda estrategia, donde las métricas mejoran respecto a la primera estrategia, pero donde se marcan zonas principalmente en la región mediastinal, no incluida en esta segmentación, se toma como una justificación de la necesidad de la tercera estrategia de aprendizaje. La tercera estrategia permitió obtener un modelo de mejores resultados, aunque requiere de mayor entrenamiento, porque el Grad-CAM detecta la tendencia del modelo a generar falsos positivos.
En esta investigación, se trabajó para atenuar los posibles sesgos de entrenamiento descritos en [28]. No obstante, el modelo durante la prueba externa solo fue capaz de igualar la máxima sensibilidad del ojo humano, por lo que es importante continuar su ajuste para incrementar su exactitud en la tarea de detectar nódulos de pulmón a partir de radiografías de tórax y alcanzar poder de generalización. Las vías para hacerlo, que se deducen de la investigación realizada, son: Acudir a una arquitectura más profunda [38], utilizar un mayor número de datos de entrada para el entrenamiento [21] y/o entrenar durante un mayor número de épocas el modelo actual. En [38] por ejemplo, se entrenaron 4 algoritmos de DL (uno de ellos con enfoque eDenseYOLOv2) y se compararon contra el desempeño de dos radiólogos expertos. Los datos de validación interna de eDenseYOLOv2 muestran una sensibilidad de 0.89, utilizando solo 944 imágenes para el entrenamiento.
Por lo general, este tipo de arquitecturas de capas convolusionales necesitan grandes volúmenes de datos. En la presente investigación se utilizaron solo 3613 imágenes, ya que, en orden de mantener el balance entre ambos conjuntos, solo se tomó la misma cantidad de imágenes sin nódulos que imágenes con nódulos y nueve imágenes fueron descartadas tras los malos resultados del proceso de segmentación (1811 x 2 = 3622 - 9 = 3613). Un dataset usualmente muy utilizado para transfer learning, como por ejemplo [21], consta de 12500 imágenes por clase y en varios retos ha demostrado su suficiencia para la tarea de separar dos clases. Sin embargo, en Medicina por lo general no se dispone de tantos datos por clase, por lo que a la tarea de incrementar el número de datos de entrenamiento se le deben dedicar esfuerzos en el futuro.
Por otro lado, el modelo obtenido, haciendo uso del valor de confianza que siempre reporta, es capaz de ordenar los datos según la probabilidad de que estén presentes los nódulos pulmonares. Esto proporciona a los especialistas un orden para manejar los datos médicos masivos. Como resultado, el flujo de trabajo puede reducir la posibilidad de retraso en el diagnóstico de cáncer de pulmón.
El costo computacional del entrenamiento para 600 épocas fue de 7 minutos por época aproximadamente, para las prestaciones computacionales empleadas. El modelo una vez entrenado, posee un tiempo de inferencia por imagen de 31 milisegundos, considerándose por tanto adecuado para una futura rutina clínica.
Para ubicar el modelo obtenido en algún punto respecto al estándar a nivel mundial, se escogió un similar de la literatura científica como punto de referencia. El sistema en cuestión [36] fue evaluado en condiciones reales en 4 hospitales. La Tabla 4 muestra el valor de la sensibilidad para la detección de nódulos pulmonares.
Tabla 4 Resultados según la sensibilidad del sistema publicado en la fuente [36]
| Hospital | Sensibilidad (%) |
|---|---|
| Seoul National University Hospital | 69,9 |
| Boramae Hospital | 82,0 |
| National Cancer Center | 69,6 |
| University of California San Francisco Medical Center | 75,0 |
Aunque el sistema mostrado en la tabla exhibe un desempeño mayor que el propuesto en esta investigación, cuya sensibilidad en la prueba externa fue de 68%, se considera que se encuentra en el entorno de lo logrado a nivel internacional en la actualidad para radiografías de tórax analizadas con inteligencia artificial.
En otro trabajo [39], se utilizaron 1048 imágenes para entrenar un algoritmo de DL. Aquí sí se realizó prueba externa y la máxima sensibilidad lograda en esta fue del 73 %. Comparar estos resultados con los presentados en el presente artículo no es del todo válido porque las BD utilizadas son diferentes, pero sí constituye una referencia de lo que en la actualidad se está logrando con CRX en la detección de nódulos pulmonares.
También, estos resultados son superados por sistemas desarrollados para imágenes en 3D con rayos x de tomografía computarizada [3], lo cual, en el contexto de Cuba, sería una fase posterior en el esquema de detección del cáncer de pulmón. Por todo lo anterior, se considera que el modelo obtenido tiene potencial, para una vez logrado su poder de generalización, implementar su algoritmo con YOLOv5 en uso clínico de rutina.
Conclusiones
La primera estrategia de entrenamiento y validación probada, entrenar el sistema con imágenes de radiografías segmentadas y normalizadas, no fue exitosa. Fue evidente el aprendizaje por atajos, causado por la pérdida de características puntuales de los nódulos durante el proceso de normalización. Cuando se retira el proceso de normalización de las imágenes segmentadas, durante la segunda estrategia de entrenamiento, los resultados mejoran, pero persisten algunos sesgos. La causa de estos sesgos es identificada a través del uso del algoritmo de Grad-CAM y corregida para la última estrategia de entrenamiento, imágenes segmentadas incluyendo el área del mediastino. En este último experimento la mejora fue apreciable, tanto en los resultados como en el análisis por medio de Grad-CAM, donde es notable la reducción del aprendizaje por atajos. Un factor decisivo es el tratamiento de las BD. Es necesario cerciorarse de que no existe ningún factor que empobrezca la calidad de las imágenes o que propicie la pérdida de información de las lesiones. Como se aprecia, el algoritmo YOLO muestra potencial para la tarea propuesta, pero aún precisa de ajustes en sus parámetros para alcanzar poder de generalización, así como su entrenamiento en un volumen de datos mucho mayor que el empleado.