INTRODUCCIÓN
El aguaymanto (Physalis peruviana L.) es una especie nativa de la región Andina, principalmente de los valles andinos de Perú, Colombia y Ecuador (Antunez-Ocampo et al., 2016). Pertenece a la familia de las Solanáceas, se caracteriza por poseer un fruto con alto valor nutricional, que contiene vitamina C y las del complejo B, minerales, polifenoles y ácidos grasos (Corrales-Bernal et al., 2015). A esta especie se le atribuyen propiedades anticancerígenas, antiinflamatorias y antioxidantes y es también conocida por su potencial rol en el control de enfermedades como la diabetes, hipertensión (Ramadan, 2011; Hassan et al., 2017) e hipercolesterolemia (Reyes-Beltrán et al., 2015).
En los últimos años se ha presentado un aumento en su demanda lo que ha generado que se promueva el desarrollo comercial del cultivo, debido justamente a las características nutricionales y propiedades medicinales que posee el fruto. Por ello, es considerado como una alternativa de producción para la economía de muchos países (Fischer et al., 2014). Su producción se ha convertido en una alternativa económica viable para los sistemas familiares campesinos (Moreno-Miranda et al., 2018) y debido a sus cualidades nutraceúticas así como por ser un producto de exportación, es de gran interés para la agroindustria (Falcón et al., 2019).
A nivel mundial, Colombia es el primer productor de aguaymanto y ha logrado posicionarse en el mercado europeo como el primer proveedor de esta fruta en estado fresco, con un valor de las exportaciones que ascendió a US$ 27.8 millones en el 2017 (Agroforum, 2018). Esto a su vez va de la mano con que en ese país el sector empresarial ha avanzado en investigación y en mecanismos financieros para el desarrollo de recursos con potencialidad en el biocomercio (Jiménez et al., 2017). En Perú, no obstante ser considerado como un lugar de origen, el cultivo en forma comercial es reciente y no masificado. A pesar de esto, las exportaciones de este recurso han estado en constante aumento. El portal de la (Agencia Agraria de Noticias (2020) refiere que en el año 2019 el volumen de exportación de aguaymanto subió en un 11.175% con respecto al año anterior. Las principales zonas de producción de aguaymanto son los departamentos de Cajamarca, Cuzco, Huancayo, La Libertad, Ayacucho y Ancash (PDRS/GIZ, 2011).
En Perú se presentan varios ecotipos o selecciones (Schreiber, 2012), lo que evidenciaría que hay una gran diversidad y potencial de explotación del recurso. Para que sea sostenible es necesario mejorar sustancialmente la calidad del producto, mediante la obtención de un estándar de aguaymanto que presente características de interés, y explotar los ecotipos que están presentes en el país. Entre los primeros pasos para lograr un estándar de aguaymanto se encuentra la realización de estudios de diversidad genética que permitirán evaluar y caracterizar estos ecotipos y así dar sustento genético a este estándar.
Los marcadores microsatélites (del inglés: Simple Sequence Repeats o SSR) son considerados como una de las herramientas más eficientes y de amplio uso en plantas (Carneiro et al., 2016). Se trata de secuencias cortas (1-6 pb) repetidas en tándem en donde el número de repeticiones revela diferencias genéticas entre individuos (Martínez, 2018). Presentan ventajas como requerir poca cantidad de ADN, herencia codominante, altamente informativos, de naturaleza multialélica, pero sobre todo, ser altamente reproducibles a nivel experimental y transferibles dentro de especies relacionadas (Souza, 2015). Por lo anterior, siguen siendo utilizados como una herramienta muy útil para evaluar diversidad genética (Roa et al., 2014; Sharafi et al., 2017; Guerra et al., 2018; León-Brito et al., 2018). Gracias al uso de estos marcadores moleculares es posible analizar una gran información genética de las poblaciones en estudio, mediante el cálculo y análisis de ciertos parámetros como las frecuencias alélicas, niveles de heterocigosidad así como los índices de diferenciación genética Fst, Fis, Fit y el análisis de varianza molecular (AMOVA) (Chacón et al., 2016).
Dentro de los cultivos andinos estudiados con microsatélites se encuentran Solanum, Rubus y Passiflora (Ortiz et al., 2012; Bushakra et al., 2015; Juyó et al., 2015). En el caso de Physalis sp., es una solanácea que se ha constituido en un grupo económicamente importante por lo que es necesario realizar estudios de diversidad genética utilizando marcadores moleculares (Gonzáles-Chavira et al., 2019).
Simbaqueba et al. (2011) fueron los primeros en informar un conjunto de 30 marcadores microsatélites polimórficos para P. peruviana y especies relacionadas. Más adelante, Garzón-Martínez et al. (2015) estudiaron la diversidad genética y la estructura poblacional de 47 accesiones de P. peruviana procedentes de la colección in vitro de la Corporación Colombiana de Investigación Agrícola (CORPOICA), y analizaron 642 marcadores SNP y 24 loci InDels (inserción o eliminación) y concluyeron que la diversidad genética estaba relacionada con el estado de cultivo (silvestre o cultivado) y no con el origen geográfico. Sin embargo, Chacón et al. (2016) estudiaron un conjunto de 85 accesiones de P. peruviana entre cultivadas y no cultivadas de tres regiones geográficas diferentes de los andes colombianos, usando 15 loci SSR, y llegaron a la conclusión que las regiones geográficas jugaron un papel importante en la estructuración de la diversidad genética en comparación con el estado biológico de la muestra (cultivado o silvestre). Por otro lado, Berdugo et al. (2015) analizaron la variabilidad molecular de P. peruviana provenientes de cinco accesiones de CORPOICA, utilizando marcadores COSII e IRGs y no encontraron polimorfismo a nivel intraespecífico. Igualmente, Morillo-Coronado et al. (2018) utilizaron marcadores RAM (del inglés: Random Amplified Microsatellite) para determinar la variabilidad genética de ejemplares procedentes de Boyacá (Colombia), y concluyeron que la variabilidad encontrada estaba influenciada por el sitio geográfico y era a nivel intraespecífico. Así mismo, Delgado-Bastidas et al. (2019) analizaron la variación genética de muestras colombianas procedentes de la colección de la Universidad de Nariño y del Centro de Investigación Tibaitata AGROSAVIA, utilizando seis marcadores SSR y evidenciaron una baja diversidad genética sin estructura poblacional.
Estos resultados algo diferentes, implican que aún se deben realizar más estudios de diversidad genética en la especie. En Perú, los estudios de aguaymanto se han centrado mayormente en evaluar la capacidad antioxidante (Jurado et al., 2016; Chau et al., 2019) o medicinal (Campos et al., 2011; Chasquibol y Yacono, 2015; Churampi et al., 2017). En cuanto a variabilidad genética, Bonilla et al. (2019) refirieron análisis de diversidad genética pero a partir de proteínas de reserva seminal. No se cuenta con informes publicados de análisis de diversidad genética utilizando marcadores moleculares para poblaciones de aguaymanto ubicadas en el territorio peruano, lo que constituye un vacío de información que debe ser cubierto para que pueda servir en posteriores programas de mejoramiento genético, y así potenciar una mayor ventaja competitiva en el mercado internacional.
Debido a ello, el objetivo del presente estudio fue determinar la diversidad genética de Physalis peruviana L. usando marcadores microsatélites (SSR) en poblaciones cultivadas de aguaymanto procedentes de tres regiones del Perú: Ayacucho, Cajamarca y La Libertad.
MATERIALES Y MÉTODOS
Material vegetal y recolección de la muestra
La recolección de los individuos de P. peruviana de poblaciones cultivadas se realizó al azar y se llevó a cabo en tres provincias diferentes del Perú. Las provincias de recolección fueron: Huamanga en el departamento de Ayacucho, San Pablo en el departamento de Cajamarca y Santiago de Chuco en el departamento de La Libertad (Figura 1). Se escogieron al azar 10 individuos de cada provincia y de cada individuo se escogieron al azar 10 hojas frescas de 2 cm de longitud. Cada conjunto de 10 hojas por individuo se trabajó por separado. Primero, las hojas fueron lavadas con alcohol 70% (v/v) y agua destilada. Posteriormente, se cortaron en tiras y se colocaron en empaques herméticos rotulados, que contenían sílica gel con la finalidad de desecar las hojas, y trasladarlos al laboratorio de Genética de la Facultad de Ciencias Biológicas de la Universidad Nacional Mayor de San Marcos (UNMSM).
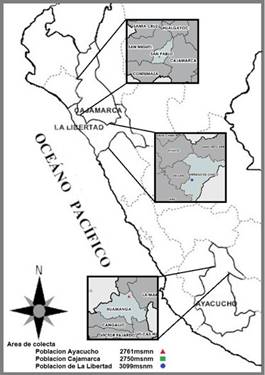
Figura 1. Mapa que muestra los sitios de recolecta de las tres poblaciones de Physalis peruviana de tres zonas diferentes del Perú: San Pablo (Cajamarca), Santiago de Chuco (La Libertad), Huamanga (Ayacucho).
Extracción de ADN
La extracción de ADN se realizó según Doyle y Doyle (1990) con algunas modificaciones: 0.6 g de hojas desecadas fueron pulverizadas dentro de un tubo de 1.5 ml, se añadieron 700 µl de solución amortiguadora de extracción (CTAB 2%, NaCl 1,4 M, 2-mercaptoetanol 0.2%, EDTA 20 mM, Tris-HCl 100 mM, pH 8.0) y se incubaron a 65 °C por 45 minutos. Se homogeneizó por inversión cada 15 minutos, después se adicionaron 700 µl de solución cloroformo:alcohol isoamílico (24:1) y se centrifugó a 14 000 g por 5 minutos. El sobrenadante se transfirió a otro tubo de 1.5 ml y se añadieron 50 µl de CTAB al 10%, se agregaron 700 µl de solución amortiguadora de extracción y se incubó a 65 °C por 45 minutos. Posteriormente, se añadieron 700 µl de solución cloroformo:alcohol isoamílico (24:1) y se centrifugó a 14 000 g por 5 minutos. El sobrenadante se transfirió a otro tubo de 1.5 ml. Se adicionaron 500 µl de isopropanol frío y se mezcló por inversión suavemente hasta observarse la fibra de ADN. Se dejó reposar a -20 °C durante 30 minutos, luego se centrifugó a 14 000 g durante 20 minutos. Se eliminó el sobrenadante y se dejó secar con el tubo invertido durante 5 minutos a temperatura ambiente. Luego el pellet se lavó con etanol al 70% durante 3 minutos, se centrifugó a 14 000 g por 30 minutos y se eliminó el sobrenadante. El tubo se dejó secando en posición invertida durante 12 horas, después se resuspendió en 100 µl de agua grado molecular y se agregaron 2 µl de RNAsa (100 mg l-1). Se incubó a 37 °C durante una hora. Finalmente, se cuantificó la cantidad de ADN (NanoDroop Lite, Thermo Scientific) y se almacenó a -20°C hasta su posterior uso.
Condiciones de PCR y electroforesis en poliacrilamida
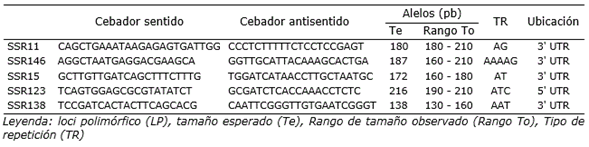
Se usaron cinco marcadores microsatélites (Tabla 1) con las secuencias de cebadores obtenidos por el estudio de Simbaqueba et al. (2011). Estas secuencias de pares de cebadores pertenecen al grupo que obtuvo mayor porcentaje de éxito (84%) en la amplificación de los productos de PCR, y además está relacionado al hecho que estos loci son específicos para P. peruviana (Simbaqueba et al., 2011). Las reacciones de PCR se realizaron de acuerdo con las siguientes condiciones: solución amortiguadora de PCR 1X, MgCl2 2.0 mM, dNTPs 0.2 µM, 0.2 µM de cada cebador, 1 U de Taq polimerasa y 100 ng de ADN genómico, en un volumen de reacción de 20 µl. El programa de PCR fue el siguiente: 95 °C durante 9 minutos, seguido de 30 ciclos de 95 °C durante 30 segundos, 53 °C durante 30 segundos y de 72 °C durante 60 segundos, seguido por una extensión final de 72 °C durante 4 minutos. Los productos de PCR fueron sometidos a una electroforesis vertical en geles de poliacrilamida (PAGE) al 12%. Los geles se tiñeron con GelREd, fueron visualizados en un transiluminador y fotografiados con una cámara Nikon D7500. Posteriormente, se determinó el tamaño aproximado de los alelos mediante una curva de regresión obtenida por el marcador de peso molecular de ADN, al medir la distancia de migración de las bandas con la ayuda del programa ImageJ.
Tabla 1. Lista de loci SSR utilizados para determinar la diversidad genética de tres poblaciones de Physalis peruviana L. de tres zonas diferentes del Perú, los cuales fueron obtenidos del estudio de Simbaqueba et al. (2011).
Análisis de la diversidad genética y estructura genética
Para el análisis de la diversidad genética se utilizó el programa GenAlEX v. 6.5 (Peakall y Smouse, 2012). Se calcularon los índices de variación en los loci de microsatélites en las tres poblaciones, el número de alelos por locus (Na), número de alelos efectivos (Ne), heterocigosidad observada (Ho), heterocigosidad esperada (He) y los índices de diferenciación genética Fst (Coeficiente de diferenciación genética entre poblaciones), Fis (Coeficiente de endogamia dentro de poblaciones), Fit (Coeficiente de endogamia total). Así mismo, con el programa GenAlEX v. 6.5 se determinó la estructura genética mediante un análisis de varianza molecular (AMOVA) (Excoffier et al., 1992). Para el AMOVA se consideró el número de alelos diferentes como una medida de la variación genética y el programa implementó 1 000 permutaciones para evaluar la significancia estadística. Por otro lado, se usó el programa iMEC (Amiryousefi et al., 2018) para calcular el contenido de información polimórfica (PIC) para cada marcador SSR.
RESULTADOS Y DISCUSIÓN
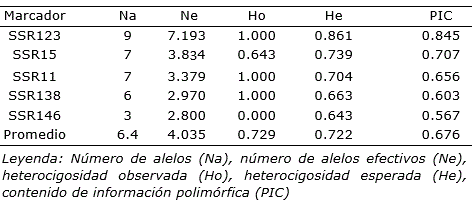
Los cinco loci microsatélites analizados dieron un total de 32 alelos, donde el marcador SSR138 presentó seis alelos, SSR11 siete, SSR146 tres, SSR15 siete y SSR123 nueve alelos. El número de alelos (Na), número de alelos efectivos (Ne), heterocigosidad observada (Ho), heterocigosidad esperada (He) y el contenido de información polimórfica (PIC) por locus se presentan en la tabla 2. Se observó que el número de alelos fue mayor en el marcador SSR123 y el que presentó menor valor fue el marcador SSR 146. La heterocigosidad observada osciló entre 0.00 (SSR146), 0.643 (SSR15) y 1.00 (SSR138, SSR11, SSR123), mientras que la heterocigosidad esperada varió desde 0.643 hasta 0.861, siendo el de menor valor el SSR146 y el de mayor SRR123. También se determinó un valor promedio de He= 0.722. El valor PIC fue de 0.567 para SSR146 y entre 0.603-0.845 para los otros marcadores (SSR138, SSR15, SSR11, SSR123). Los valores PIC brindan un estimado del poder discriminante de un locus, teniendo en cuenta tanto el número de alelos expresados como la frecuencia relativa que en ellos aparecen (Oladasu et al., 2015). Según Arias et al. (2018) estos valores pueden oscilar entre 0 (para marcadores monomórficos) y 1 (para marcadores altamente discriminantes). De los resultados, cuatro de los cinco marcadores (SSR138, SSR15, SSR11, SSR123) son considerados altamente polimórficos (PIC >0.6) y el marcador SSR146 mostró un polimorfismo considerado como moderado (0.567) (Hildebrand et al., 1992; Chacón et al., 2016).
Tabla 2. Valores de diversidad genética(por locus) de la población en estudio de Physalis peruviana L. de tres zonas de Perú.
Los índices de diversidad genética promedio de los SSR se diferenciaron por población (Tabla 3). La población de Ayacucho presentó el mayor número de alelos promedio, pero el menor valor de heterocigosidad esperada promedio. Por su parte, la población de La Libertad presentó el menor número de alelos promedio, pero el mayor valor de heterocigosidad esperada promedio. Este resultado está acorde con la fórmula del cálculo matemático de He (Liu, 1998). En cuanto al promedio de número de alelos por locus, el valor encontrado (Na=3.8) fue mayor que el referido por Delgado-Bastidas et al. (2019) quienes señalaron un promedio de 1.22 en material genético de P. peruviana utilizando seis marcadores SSR. Sin embargo, se aproxima a otros estudios, como el de Chacón et al. (2016) quienes refieren un valor promedio de Na de 3.05 para accesiones colombianas de P. peruviana utilizando marcadores microsatélites (SSR).
Tabla 3. Valores de diversidad genética de tres poblaciones de Physalis peruviana L. basados en el promedio de las estadísticas de marcadores microsatélites (SSR) en cada población.

Por otra parte, los rangos en pares de bases (pb) estimados experimentalmente para cada marcador en este trabajo fueron SSR11 de 160-174, SSR146 de 197-237, SSR15 de 169-227, SSR123 de 208-235 y SSR138 de 131-161 (Figura 2). Sin embargo, difirieron con los rangos esperados propuestos por Simbaqueba et al. (2011) (Tabla 1), a excepción del SSR138 que presentó el valor más cercano.
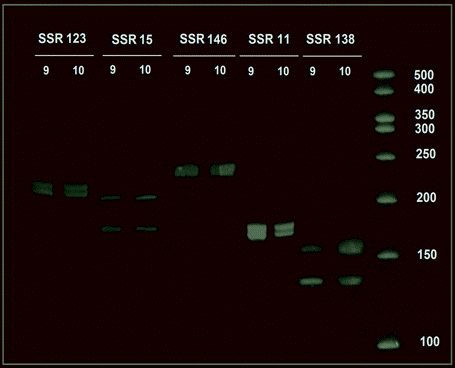
Figura 2. Patrón electroforético combinado de dos individuos de Physalis peruviana L. de la población de Cajamarca usando cinco marcadores SSR. Electroforesis vertical en geles de poliacrilamida (PAGE) al 12%, tinción con GelRed. A la derecha marcador de peso molecular.
Al igual que en el presente estudio, autores como Chacón et al. (2016) analizaron la diversidad genética del aguaymanto en accesiones colombianas, con los cebadores para marcadores SSR diseñados por Simbaqueba et al. (2011), y obtuvieron bajos niveles de diversidad genética (He=0.223).
Existen otros estudios donde se muestran valores diferentes de He. Por ejemplo, Garzón-Martínez et al. (2015) utilizaron marcadores InDel en 47 accesiones colombianas de aguaymanto y mostraron valores de He= 0.3. Igualmente, Bonilla et al. (2008) señalaron un valor de He= 0.255 usando marcadores RAMs en 43 accesiones silvestres y Osorio-Guarín et al. (2016) obtuvieron un He= 0.647 al utilizar 5000 marcadores SNP en 100 accesiones colombianas, entre cultivadas y silvestres. Más recientemente, Delgado-Bastidas et al. (2019) utilizaron seis marcadores SSR, y señalaron un He promedio de 0.09. Estas diferencias en los valores de estimación de diversidad genética pueden depender de muchos factores como el tamaño de la muestra, las accesiones de origen y los marcadores moleculares que se utilizan (Chacón et al., 2016). Sin embargo, el alto valor de PIC>0.6 que presentaron los marcadores usados en este estudio, a excepción del SSR 146, sugiere que estos son muy informativos en el análisis de la diversidad genética de estas poblaciones de aguaymanto y además, tal como indicaron tanto Chacón et al. (2016) y Tomas et al. (2017), aquellos con altos valores de PIC serían mucho más útiles para propósitos de identificación y selección de germoplasma.
Autores como Chen et al. (2017) han sugerido que los valores de PIC para los marcadores de SNP bialélicos varían de 0 a 0.5 mientras que para los SSR multialélicos, el valor de PIC puede ser tan alto como 0.5 - 1.0. Con respecto a esto, el valor de PIC=0.567 del marcador SSR 146, el cual es el menor de todos, se encontraría dentro del rango. Además, de acuerdo con Botstein et al. (1980) para valores de PIC>0.5 el marcador presentaría un alto valor informativo. Sin embargo, se han sugerido valores PIC > 0.6 para poder tener mayor resolución a la hora de identificar el germoplasma de distintas variedades (Chacón et al., 2016).
Por otro lado el análisis de AMOVA mostró que la mayoría de la variación genética (74.5%) se encontraba dentro de los individuos, con un Fit de 0.104 significativo, y solo el 25.5% de variación se encontraba entre las tres poblaciones analizadas, esto supone un coeficiente de Fst de 0.307, que fue significativo y alto (Tabla 4).
Tabla 4. Análisis de Varianza Molecular (AMOVA) entre tres poblaciones de Physalis peruviana L. de Perú, usando cinco marcadores microsatélites (SSR).
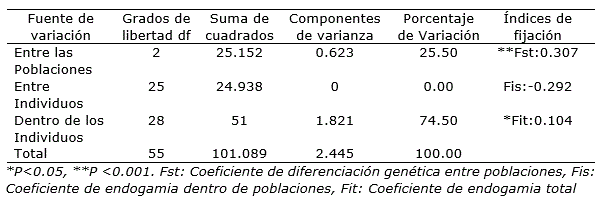
Estos resultados indicarían que no hay estructuración genética en estas poblaciones lo cual puede deberse a que las tres poblaciones pudieron derivarse de una sola variedad que se ha sometido a un proceso de domesticación temprana, como los programas de selección artificial, que le lleva a perder su diversidad genética (Rauf et al., 2010). También estaría en correspondencia con lo que menciona Aguilar (2018), quien propone que la fruta de aguaymanto que se comercializa proviene de semilla introducida de Colombia y es la que se siembra en los departamentos de Ancash, Ayacucho, Cajamarca y Cuzco.
Las comparaciones por pares revelaron que el Fst (Coeficiente de diferenciación genética entre poblaciones) osciló entre 0.243 y 0.325 (Tabla 5). Estos valores indicarían a su vez una diferenciación genética entre las poblaciones (Balloux y Lugon-Moulin, 2002), lo cual llevaría a pensar que si bien estas tres poblaciones pueden derivarse de una sola variedad, otros factores como el modo de cultivo, flujo genético por agentes polinizadores o intercambio de semillas pudieran estar contribuyendo a esta diferenciación genética por pares (Chacón et al., 2016). Adicionalmente se puede interpretar que las poblaciones con menos diferenciación serían Cajamarca y La Libertad, mientras las que presentan más diferencias entre ellas son Ayacucho y La Libertad.
Autores como Hake y Ross-Ibarra (2015) e Isbell et al. (2017) refirieron que niveles altos de diversidad genética entre las poblaciones de especies de plantas cultivadas pueden otorgar beneficios potenciales evolutivos con el fin de hacer frente al cambio ambiental, la selección artificial y la infección por agentes patógenos, lo que aumentaría los rendimientos y reduciría las pérdidas económicas. Los resultados obtenidos en el presente estudio se enmarcarían dentro de dicho planteamiento.
Estudios como el de Arthofer et al. (2018) han demostrado que el uso de pocos marcadores microsatélites puede aún revelar la estructura poblacional si estos son altamente polimórficos. De acuerdo con lo mencionado, a pesar de que en este estudio se utilizan cinco marcadores SSR, el alto grado de polimorfismo evidenciado en los valores PIC, brindarían un buen panorama de las relaciones genéticas entre estas poblaciones. Además, según Hodel et al. (2016), la alta variación alélica de los microsatélites resulta óptima para la identificación de marcadores presentes en pequeñas subpoblaciones.
Los resultados que aquí se muestran, brindan información comparativa acerca de la variabilidad genética del aguaymanto procedente de Ayacucho, Cajamarca y La Libertad. Según Ramos y Matos (2016) las variables en el manejo del aguaymanto dependen de factores genéticos, climáticos y agroculturales, los que finalmente influyen en las características fisicoquímicas y sensoriales del fruto. Entonces, es recomendable que la información genética obtenida sea complementada con estudios bioquímicos, nutricionales, geográficos y agroculturales del cultivo de aguaymanto para tener un panorama global de mejoramiento de la calidad en la cadena productiva de este fitorecurso.
CONCLUSIONES
Poblaciones de Physalis peruviana de tres regiones de Perú (Cajamarca, La Libertad y Ayacucho) presentan valores altos de diferenciación genética y bajo porcentaje de variación entre las poblaciones lo cual evidenció una misma unidad genética, no estructurada. Cuatro, de los cinco, marcadores genéticos microsatélites (SSR123, SSR 11, SSR 15, SSR138) muestran altos valores de diversidad genética, lo que indica un alto grado de polimorfismo para las poblaciones muestreadas. Dicha información podrá ser utilizada para iniciar futuros estudios de mejoramiento y caracterización genética entre las poblaciones de aguaymanto peruano.
















