Meu SciELO
Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Cubana de Ciencias Informáticas
versão On-line ISSN 2227-1899
Rev cuba cienc informat vol.7 no.4 La Habana oct.-dez. 2013
ARTÍCULO ORIGINAL
Adaptación para múltiples GPU de un simulador de actividad eléctrica en el corazón
Multi-GPU adaptation of a simulator of heart electric activity
Víctor M. García*, Antonio M. Vidal, Alejandro Liberos, Andreu M. Climent
Departamento de Sistemas Informáticos y Computación. Universitat Politècnica de València, Camino de Vera, s/n 46022, Valencia, Spain.
* Autor para la correspondencia: vmgarcia@dsic.upv.es
RESUMEN
La simulación de la actividad eléctrica del corazón se calcula mediante la resolución de un gran sistema de ecuaciones diferenciales ordinarias, que necesita una enorme cantidad de tiempo de computación. Sin embargo, en los últimos años se están introduciendo, en el ámbito de la computación de alto rendimiento, las unidades de procesamiento gráfico (GPU). Estos potentes dispositivos han atraído a grupos de investigación que requieren simular la actividad eléctrica del corazón. El grupo de investigación que firma este artículo ha desarrollado un simulador de actividad eléctrica cardíaca que se ejecuta en una sola GPU. En este artículo se describe la adaptación y modificaciones de dicho simulador para su ejecución en múltiples GPU. Los resultados confirman que la técnica empleada permite reducir sensiblemente los tiempos de ejecución respecto a los que se obtienen con una sola GPU, además de permitir afrontar problemas mucho más grandes.
Palabras clave: Ecuaciones diferenciales ordinarias, programación GPU, simulación electrocardíaca.
ABSTRACT
The simulation of the electrical activity of the heart is calculated by solving a large system of ordinary differential equations; this takes an enormous amount of computation time. In recent years graphics processing unit (GPU) are being introduced in the field of high performance computing. These powerful computing devices have attracted research groups requiring simulate the electrical activity of the heart. The research group signing this paper has developed a simulator of cardiac electrical activity that runs on a single GPU. This article describes the adaptation and modification of the simulator to run on multiple GPU. The results confirm that the technique significantly reduces the execution time compared to those obtained with a single GPU, and allows the solution of larger problems.
Key words: Electrocardiac simulation, GPU programming, ordinary differential equations.
INTRODUCCIÓN
Simular la actividad eléctrica de las células cardíacas es de crucial importancia para una correcta simulación global del corazón (Almendral, 2006; Reumann, 2007). La actividad eléctrica del corazón se obtiene de la resolución de un sistema de ecuaciones diferenciales ordinarias (EDOs) de valores iniciales. Cada célula se modela con 15-40 ecuaciones diferenciales. Una simulación 3D precisa puede requerir miles o incluso millones de células, por lo que la potencia de cálculo requerida es enorme. Es por ello que la simulación de unos segundos de la actividad cardíaca puede requerir de horas o incluso días de computación.
Una posibilidad a la hora de acortar estos tiempos de simulación consiste en llevar a cabo los cálculos sobre unidades de procesamiento gráfico (GPU). Estos dispositivos se suelen encargar de la interfaz gráfica de usuario y actualmente cuentan con una potencia de cálculo superior a una CPU (unidad central de procesos) estándar. La GPGPU es una nueva generación de GPU conocida como de propósito general que ha sido diseñada específicamente para cómputos de alto rendimiento. El principal inconveniente para el uso de estos dispositivos es que su programación para tareas generales es bastante compleja. Este problema se ha aliviado en cierta medida con la arquitectura CUDA desarrollada por NVIDIA.
Los firmantes de este artículo han desarrollado un simulador basado en CUDA para la actividad eléctrica cardíaca (García, 2011). En este artículo se describen las técnicas específicas que se han utilizado para extender el simulador de forma que pueda ejecutarse en varias GPUs simultáneamente. Una ventaja evidente de utilizar varias GPUs es que se dispone de mucha más memoria y, por lo tanto, se pueden afrontar problemas con muchas más células. Por otro lado, mediante el uso de múltiples GPUs se pretende también disminuir el tiempo total de cómputo.
El resto del artículo está organizado de la siguiente forma: en primer lugar describiremos el problema afrontado y las técnicas básicas para la versión inicial del simulador. Después describiremos las técnicas específicas para la versión multigpu y la reordenación necesaria de las células del modelo, realizada para minimizar las comunicaciones entre las diferentes GPUs.
Por último daremos algunos resultados.
METODOLOGÍA COMPUTACIONAL
Modelo cardíaco y métodos computacionales
Modelo matemático cardíaco
El trabajo realizado se ha basado en el modelo de potencial de membrana auricular de Courtemanche (1998). Este modelo se basa en experimentos sobre células auriculares humanas. Se han realizado experimentos con diferentes tipos de estructuras; algunas sencillas, (1 y 2-dimensionales) con el objetivo de ayudar al desarrollo y comprobación del software, y otras más complejas, tridimensionales, para realizar simulaciones realistas. Las células vecinas se acoplaron con una conductancia g, que se ha fijado a un valor constante ajustado para obtener una velocidad de propagación realista.
El tejido cardíaco se modeló utilizando la siguiente ecuación diferencial ordinaria:
![]() (1)
(1)
siendo ![]() el potencial transmembrana de la célula i-ésima, Itotal el resultado neto de la contribución de todas las corrientes transmembrana descritas por el modelo matemático celular,
el potencial transmembrana de la célula i-ésima, Itotal el resultado neto de la contribución de todas las corrientes transmembrana descritas por el modelo matemático celular, ![]() la capacidad transmembrana, y
la capacidad transmembrana, y ![]() la conductancia entre las células
la conductancia entre las células ![]() y
y ![]() .
.
El modelo de Courtemanche utiliza como variable principal el potencial transmembrana ![]() y como ecuación principal la ecuación (1). Sin embargo, dicho modelo comprende también otras 20 variables y 20 otras EDOs. Escribimos el vector de las 21 variables asociadas a la célula k-ésima como
y como ecuación principal la ecuación (1). Sin embargo, dicho modelo comprende también otras 20 variables y 20 otras EDOs. Escribimos el vector de las 21 variables asociadas a la célula k-ésima como ![]() y escribimos el sistema de 21 EDOs para la célula k-ésima como:
y escribimos el sistema de 21 EDOs para la célula k-ésima como:
![]() (2)
(2)
Si el sistema considerado tiene ![]() células, la dimensión total del sistema de EDOs es
células, la dimensión total del sistema de EDOs es ![]() . Agrupando todas las variables dependientes de todas las células
. Agrupando todas las variables dependientes de todas las células ![]() en el vector
en el vector ![]() , y agrupando todas las funciones
, y agrupando todas las funciones ![]() en la función vectorial
en la función vectorial ![]() , dicho sistema se puede escribir en la forma estándar:
, dicho sistema se puede escribir en la forma estándar:
![]() (3)
(3)
con condiciones iniciales ![]() . Obsérvese que evaluar la función
. Obsérvese que evaluar la función ![]() equivale a evaluar
equivale a evaluar ![]() para todas las células.
para todas las células.
Método Numérico utilizado
Existen numerosos métodos para resolver sistemas de ecuaciones diferenciales ordinarias: Runge-Kutta, Burlisch-Stoer, Predictor-Corrector, etc (Ascher, 1998; Press 1992; Shampine, 1997). También existe la posibilidad de usar métodos con paso adaptativo o con paso fijo. Los métodos adaptativos han sido explorados para este problema por los autores en (Garcia, 2011).
Para la versión inicial se ha utilizado el método de Euler explícito. Este es el método más simple para resolver sistemas de EDOs como (2).Tras la selección de un paso temporal ![]() , y dada la solución inicial
, y dada la solución inicial ![]() en el instante
en el instante ![]() , se puede obtener una solución aproximada en el instante
, se puede obtener una solución aproximada en el instante ![]() aplicando el algoritmo (1)
aplicando el algoritmo (1)

Es bien sabido que este método no es muy apropiado para sistemas de EDOs en general, porque no es muy preciso y el máximo paso de tiempo posible no es muy grande. Sin embargo, para el problema de simulación de células, existe una versión del método de Euler, llamado método de Rush-Larsen (Rush, 1978) y estudiado en diferentes artículos (Maclachlan 2007, Marsh 2012, Spiteri 2008, Perego 2009, Sundnes 2009), que permite obtener una precisión razonable con pasos de tiempo bastante largos. El método de Rush-Larsen utiliza la forma específica de ciertas ecuaciones del modelo celular (ecuaciones de puerta o "gating") para obtener una solución seudo-analítica de dichas ecuaciones. Dicha modificación hace que el método resultante sea mucho más eficiente, cuando se aplica a simulaciones celulares. El método de Rush-Larsen se ha aplicado en el simulador; sin embargo, y para evitar explicaciones demasiado largas, supondremos que el método aplicado es el de Euler explícito.
Tal como está descrito, no hay paralelismo en el algoritmo 1. Sin embargo, si reescribimos el algoritmo 1 indicando las operaciones para cada célula:

entonces queda claro que el bucle interno se puede paralelizar sin mayor problema, dado que la evaluación de la función ![]() es independiente para cada célula.
es independiente para cada célula.
Unidades de procesamiento gráfico (GPU)
Un programa CUDA se compone de un programa principal que se ejecuta en la CPU, y unos subprogramas especiales, llamados kernels, que se ejecutan en la GPU. Cada kernel se invoca desde el programa principal y asigna una cierta tarea a la GPU. Cuando un kernel se envía a la GPU, muchas instancias de dicho kernel se ejecutan en paralelo en la GPU. Cada una de estas instancias es ejecutada por un thread, cada thread se ejecuta en uno de los muchos microprocesadores o "cores" de la GPU. Los threads se organizan en bloques de threads, que se ejecutan conjuntamente. En la llamada al kernel, el programador debe especificar como parámetros de kernel, cuantos bloques (NumBlocks) y cuantos threads por bloque (NumThreadsperBlock) deben utilizarse para ejecutar este kernel.
Nuestros solvers CUDA se han estructurado de forma que se obtiene paralelismo realizando los cálculos para muchas células simultáneamente. Los cálculos para una cierta célula los llevará a cabo un solo thread CUDA. Si, como es habitual, el número de celdas es mayor que el número de threads, cada thread se encarga de varias células: Supongamos que hay NumCells células y NumThreads threads (NumThreads = NumBlocks · NumThreadsperBlock), con NumCells>NumThreads. Cada thread es identificado por un número entero Tid, 0 <=Tid < NumThreads. Entonces, en un cierto paso,el thread Tid empezará procesando la célula Tid. Cuando la acabe, empezará con la célula NumThreads + Tid, luego procesará la célula 2 · NumThreads + Tid y así sucesivamente, hasta que todas las células hayan sido procesadas.
La estructura del bucle principal, que se ejecuta en la CPU, sería la que se muestra en el Algoritmo 3, y, salvo por algunos detalles, cada kernel será como el algoritmo 4.

Adaptación para multiplesGPUs
La adaptación para múltiples GPUs resulta conceptualmente bastante sencilla. La idea básica consiste en repartir las células (o celdas) entre las diferentes GPUs, y programar las comunicaciones necesarias tras cada paso temporal de forma que cada célula que tenga alguna célula vecina residiendo en otra GPU, pueda acceder al valor actualizado del voltaje de dicha célula.

Para obtener buen rendimiento, es necesario que el bucle interno se ejecute en paralelo, y que el intercambio de datos sea lo más eficiente posible. Esto se consigue (al menos parcialmente) reduciendo la cantidad de datos que es necesario enviar.
Para la programación del algoritmo 5 se han utilizado las técnicas propuestas en el documento (Cuda, 2013), en el apartado de programación multiGPU, y en (Farber, 2012): fundamentalmente, utilizar la instrucción CudaSetDevice para seleccionar cada GPU, y utilizar la copia asíncrona entre GPUs: CudaMemcpyAsync, para simultanear el tráfico de datos entre GPUs.
Ordenación de las células
Durante el desarrollo del simulador (en sus diferentes versiones), se han utilizado diferentes modelos; unidimensionales ("hilos" de células), bidimensionales ("planos de células"), casos tridimensionales para desarrollo (como por ejemplo, estructuras esféricas) y casos tridimensionales realistas. Para casos uni y bidimensional, resulta sencillo determinar y programar las comunicaciones. Sin embargo, para grandes estructuras cardiacas tridimensionales, el problema se hace más complicado. Si numeramos las células de 1 a ![]() , y disponemos de
, y disponemos de ![]() GPUs, supondremos que las primeras
GPUs, supondremos que las primeras ![]() /
/![]() células van a la primera GPU, las siguientes van a la segunda, etc.
células van a la primera GPU, las siguientes van a la segunda, etc.
Entonces, el objetivo es determinar una ordenación o numeración de las células de forma que las comunicaciones sean mínimas.
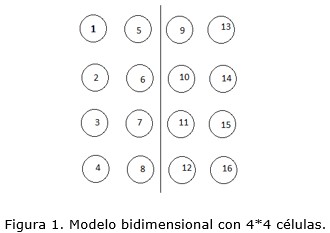
Si la ordenación de las células se hace de forma irreflexiva, la cantidad de comunicaciones entre GPUs puede ser mucho mayor de lo necesario. Para ilustrar este problema, consideremos un simple caso bidimensional con 4 por 4 células (ver figura 1);
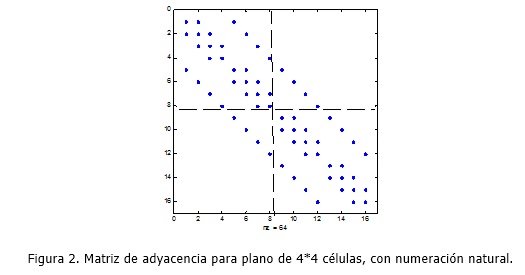
Si consideramos la matriz que relaciona cada célula con sus vecinas (consideramos sólo las situadas encima/debajo o a izquierda/derecha, no las vecinas en dirección oblicua), obtenemos una matriz característica de la discretización de la ecuación de Poisson, cuya estructura se puede observar en la figura 2:
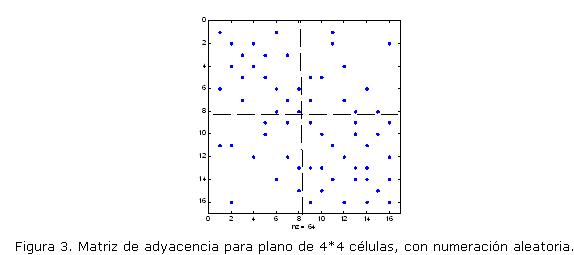
Las líneas internas indican la división natural en dos GPUs, asignando las primeras 8 células a la primera GPU y las otras 8 a la segunda. El número de valores de la segunda GPU que se deben comunicar a la primera es el número de elementos distintos de cero que hay en los bloques no diagonales fuera de la diagonal, en este caso 4 células. Si realizamos una ordenación aleatoria de las células, el número de valores a enviar puede aumentar considerablemente. En la figura 3 obtenida con el mismo plano pero con ordenación aleatoria, el número de valores a enviar sube a 11.
En un caso tridimensional con un número realista de células es más difícil obtener una ordenación natural, y el efecto de la ordenación puede ser mucho más importante.
El problema de ordenación para minimizar las comunicaciones es similar al problema de reordenar matrices dispersas para que, al calcular descomposiciones como LU o Cholesky, el relleno resultante sea mínimo. Una de las técnicas más habituales consiste en reordenar las filas y columnas de la matriz dispersa de forma que el ancho de banda de la matriz resultante se reduzca. Nosotros hemos optado por reordenar las células aplicando a la matriz de adyacencia el conocido reordenamiento de Cuthill-McKee inverso (Cuthill, 1969). Dicho reordenamiento es bien conocido en la teoría de solución de sistemas lineales dispersos. Tiene la propiedad de obtener un reordenamiento que, habitualmente, disminuye el ancho de banda.
En las figuras 4 y figura 5 se puede apreciar el efecto de la reordenación. Si se realiza la partición de células sobre la ordenación original, la cantidad de valores que se deben enviar entre las GPUs es muy grande.
Sin embargo, en la figura 5 se ve que la cantidad de valores que hay que enviar, una vez reordenadas las células, es muchísimo menor.
Una vez reordenadas las células, ya se puede proceder a dividir las células entre las GPUs en grupos más o menos iguales. Mediante una sencilla aplicación MATLAB, podemos determinar cuales tienen que ser los envíos entre cada GPU.
RESULTADOS Y DISCUSIÓN
El código multi-GPU obtenido se ha probado con un modelo tridimensional esférico de 163842 células. Con este número de células, la dimensión total del sistema es de 21·163,842 = 3, 440, 682. El tiempo simulado es 0.3 segundos. Los resultados obtenidos muestran la misma precisión que los resultados obtenidos con una sola GPU. El código se ha probado en una máquina con 4 GPUs Tesla 2050, con 448 cores y 6 Gb de memoria global cada una. También hemos experimentado con una máquina equipada con una GPU Geforce 590GTX. Esta GPU es una GPU dual, compuesta en realidad por 2 GPUs conectadas directamente, cada una con 512 cores y 1,5 Gb. Estas dos GPUs son capaces de comunicarse a más velocidad que las GPUs que se conectan a través de los puertos del sistema.
Es necesario mencionar que la máquina multiGPU fue accedida de forma remota y multiusuario, mientras que el trabajo con la máquina con la GPU dual fue realizado en modo local y con un solo usuario. Los resultados son llamativos; en la máquina multiGPU, con nuestro caso de prueba, el tiempo de ejecución pasa de 23.4 segundos con una GPU, 16 segundos con 2 GPU, y por último 11.95 segundos con 4 GPUs. Por otro lado, con la GPU dual en el mismo caso se tardó 12 segundos con una de las dos GPUs, y 6.7 segundos usando las dos GPUs de la GPU dual.
Los resultados prueban claramente que para este problema se pueden obtener reducciones de tiempo muy importantes usando varias GPUs. Especialmente llamativo es el caso de la GPU dual, que da un speedup casi perfecto.
CONCLUSIONES
Los resultados muestran que, para resolución de Sistemas de EDOs donde las ecuaciones diferenciales puedan evaluarse de forma independiente (como las diferentes células en este caso), utilizar múltiples GPUs ofrece un rendimiento muy apreciable, además de ser muy dispositivos muy económicos en comparación con CPUs de potencia similar. Nuestros próximos trabajos van orientados a obtener versiones MultiGPU de nuestros códigos con paso adaptativo.
AGRADECIMIENTOS
Este trabajo ha sido financiado por la Universitat Politècnica de València a través de su Programa de Apoyo a la Investigación y Desarrollo (PAID-06-11) y (PAID-05-12), por la Generalitat Valenciana a través de los proyectos PROMETEO/2009/013 y Ayudas para la realización de proyectos de I+D para grupos de investigación emergentes GV/2012/039, y por el Ministerio Español de Economía y Competitividad y el fondo europeo de Desarrollo Regional (FEDER) de la Comunidad Europea través del proyecto TEC2012-38142-C04.
También queremos agradecer al grupo de Computación de Altas Prestaciones de la Universidad Jaume-I de Castellón, y a su director Enrique Quintana, por permitirnos usar sus infraestructuras de cálculo.
REFERENCIAS BIBLIOGRÁFICAS
ALMENDRAL, J.; MORENO, J.; VAIDYANATHAN, R. et al. Activation of inward Rectifier Potassium Channels Accelerates a trial Fibrillation in Humans: Evidence for a Reentrant Mechanism, Circulation 114 (2006) p. 2434-2442.
ASCHER, U. M.; PETZOLD, L. R. Computer Methods for Ordinary Differential Equations and Differential-Algebraic Equations, SIAM, Philadelphia. Capítulos 3-4. 1998.
COURTEMANCHE, M.; RAMIREZ, R. J.; NATTEL, S. Ionic Mechanisms Underlying Human Atrial Action Potential Properties: Insights from a Mathematical Model. American Journal of Physiology-Heart and Circulatory Physiology 275 (1998) 1 Pt 2:H301-21.
CUDA. Online [Consultado en: Junio de 2013]. Disponible en: [http://docs.nvidia.com/cuda/cuda-c-programming-guide/index.html].
CUTHILL, E.; McKEE, J. Reducing the Bandwidth of Sparse Symmetric Matrices , en Proc. 24th Nat. Conf. ACM, p. 157-172, 1969.
FARBER R. CUDA Application Design and Development, Morgan Kaufmann Publishers, Waltham, MA, 2011.
MACLACHLAN, M. C., SUNDNES J., SPITERI R J. A Comparison of Non-Standard Solvers for ODEs Describing Cellular Reactions in the Heart, Computer Methods in Biomechanics and Biomedical Engineering 10 (2007) 317-326.
MARSH, M. E., ZIARAGATHI S. T., SPITERI R. J., The Secrets to the Success of the Rush-Larsen Method and its Generalizations, IEEE Transactions on Biomedical Engineering59 (2012) 2506-2515.
PEREGO M., VENEZIANI A., An Efficient Generalization of the Rush-Larsen method for solving Electro-Physiology Membrane Equations, Electronic Transactions on Numerical Analysis 35 (2009) 234-256.
PRESS, W. H. TEUKOLSKY, S.A., VETTERLING, S.A., FLANNERY, B.P. Numerical Recipes2nd Edition: The Art of Scientific Computing, Cambridge University Press, 1992.
REUMANN, M.; FARINA, D.; MIRI, R.; LURZ, S.; OSSWALD, B. y DOESSEL, O., Computer Model for the Optimization of AV and VV Delay in Cardiac Resynchronization Therapy, Medical & Biological Engineering & Computing 45 (2007) p. 845-854.
RUSH, S.; LARSEN, H., Practical Algorithm for Solving Dynamic Membrane Equations. IEEE Transactions on Biomedical Engineering 25 (1978) 389-392.
SHAMPINE, L. F. y REICHELT, M. W. The MATLAB ODE suite, SIAM Journal on Scientific Computing 18 (1997) 1-22.
SPITERI R. J. DEAN R. C. On the Performance of an Implicit-Explicit Runge-Kuttamethod in Models of Cardiac Electrical Activity, IEEE Transactions on Biomedical Engineering 55, 2008, 1488-95. doi: 10.1109/TBME.2007.914677.
SUNDNES J., ARTEBRANT, R., SKAVHAUG O., TVEITO A. A Second-Order Algorithm for Solving Dynamic Cell Membrane Equations, IEEE Transactions on Biomedical Engineering56 (2009) 2546-8. doi: 10.1109/TBME.2009.2014739.
Recibido: 23/09/2013
Aceptado: 31/10/2013