Mi SciELO
Servicios Personalizados
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Cubana de Ciencias Informáticas
versión On-line ISSN 2227-1899
Rev cuba cienc informat vol.9 no.2 La Habana abr.-jun. 2015
ARTÍCULO ORIGINAL
Clasificación de células cervicales con Máquinas de Soporte Vectorial empleando rasgos del núcleo
Cervical cell classification with Support Vector Machines using nucleus’ features
Solangel Rodríguez-Vázquez1*, Andy Vidal Martínez-Borges2
1 Universidad de las Ciencias Informáticas. Carretera San Antonio de los Baños Km 2½, Rpto. Torrens, La Lisa, Ciudad de la Habana.
2 Empresa de tecnología para la defensa, XETID. Carretera San Antonio de los Baños Km 2½, Rpto. Torrens, La Lisa, Ciudad de la Habana.
*Autor para la correspondencia: svazquez@uci.cu
RESUMEN
La citología convencional es una de las técnicas más utilizadas, siendo ampliamente aceptada, de bajo costo, y con mecanismos de control. Con el objetivo de aliviar la carga de trabajo a los especialistas, algunos investigadores han propuesto el desarrollo de herramientas de visión computacional para detectar y clasificar las transformaciones en las células de la región del cuello uterino. En este trabajo, se presenta el uso de las máquinas de soporte vectorial (SVM) como método computacional para la clasificación de las células cervicales en las condiciones normal y anómala, basándose solamente en las características extraídas de la región ocupada por el núcleo, sin hacer uso de las características del citoplasma. La importancia de este enfoque viene dada porque los núcleos son las zonas que pueden ser segmentadas más fácilmente en imágenes complejas de frotis de Papanicolaou. Dichas imágenes presentan un alto grado de células superpuestas y es difícil lograr diferenciar las fronteras exactas de las regiones ocupadas por los citoplasmas. A partir del estudio realizado, entre los kernels lineal y función de base radial (RBF) a partir de las medidas AUC, medida F, predictividad negativa y media H,se comprobó que RBF mostró un buen desempeño manteniendo valores de 0.91% de AUC. Los resultados obtenidos indican una reducción respecto a la tasa de falsos negativos en la prueba de Papanicolaou. Se utilizó la media H con el propósito de comparar los resultados de SVM con el kernel RBF respecto a otras investigaciones, obteniendo un 91.28% por encima de las mismas.
Palabras clave: células cervicales; clasificación; frotis de papanicolaou; máquinas de soporte vectorial.
ABSTRACT
The conventional cytology is one of the most used techniques, being widely accepted, inexpensive, and with control mechanisms. With the objective of alleviating the workload of to experts, some researchers have proposed the development of tools for computer vision to detect and classify the transformations in the cells of the cervix region. In this paper, is presented the use of the support vector machines (SVM) as a computational method for classification of cervical cells in normal and abnormal conditions, based solely on the extracted features of the region occupied by the nucleus without use of the characteristics of the cytoplasm. The importance of this approach lies in that the nuclei are the region that can be more easily segmented into complex images Pap smear. These images show a high degree of overlapping cells and is difficult to distinguish the exact boundaries of the regions occupied by the cytoplasm. As of study between the linear and RBF kernels on function AUC measures as F, H and half negative predictability was found that performed well RBF values keeping 0.91% AUC. The results indicate a reduction from the rate of false negative Pap test. H mean was used in order to compare the results of SVM with RBF kernel respect to other investigations, obtaining 91.28% above the same.
Key words: cervical cells; classification; pap smears; support vector machines.
INTRODUCCIÓN
La implementación de los programas de tamización mediante el uso de la citología cervical, ha posibilitado de forma efectiva la reducción de la mortalidad por cáncer de cuello uterino en los países desarrollados. Varios factores resultan críticos para el éxito de este tipo de programas; uno muy importante está relacionado con la capacidad que tienen los patólogos y citotecnólogos de realizar una adecuada interpretación (Lorenzo-Ginori and Rodríguez-Santos, 2012) lo que acarrea una elevada tasa de falsos negativos en la prueba.
Un diagnóstico adecuado de presencia o ausencia de lesiones preneoplásicas o neoplásicas de cuello uterino, depende del desarrollo de diferentes fases, como son: la recolección y preparación de los especímenes, es decir, la toma de la muestra, la fijación, el envío, la coloración y el montaje de las láminas (fase preanalítica); segundo, la interpretación microscópica y la elaboración del informe (fase analítica), y, tercero, la validación de los resultados mediante la comparación entre observadores y la correlación de la citología con la biopsia. Una buena parte de estos procedimientos son subjetivos y, en consecuencia, se convierten en procedimientos que dependen del operador.
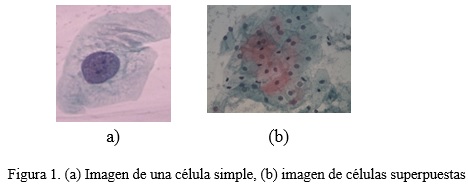
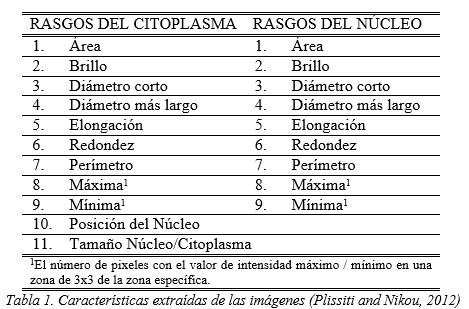
Existen diferentes técnicas que han sido llevadas a cabo en la clasificación de células cervicales como clasificadores bayesianos (Riana and Murni, 2009), redes neuronales artificiales (Mat-Isa et al., 2008), máquinas de soporte vectorial (SVM) (Huang et al., 2007) y búsqueda de vecinos más cercanos (Marinakis et al., 2009). La mayoría de estos métodos utilizan imágenes pre-segmentadas que contienen solo una célula, por lo que la segmentación correcta del núcleo y del citoplasma es factible (figura 1 (a)). En las imágenes que contienen grupos de células (figura 1 (b)), la detección de la frontera del citoplasma es un problema difícil debido a la superposición de las células. Sin embargo, la detección y segmentación de los núcleos en este tipo de imágenes ha sido abordado con éxito por varios estudios (Plissiti et al., 2011a) (Plissiti et al., 2011b). Las muestras que se toman en la prueba de Papanicolaou corresponden con la figura 1(b), por lo que se hace necesario el estudio de la clasificación de las células en función de las características del núcleo ya que es más fácil la detección de fronteras en los núcleos que en los citoplasmas.Las técnicas de clasificación que se utilizan en las imágenes de Papanicolaou se basan en el cálculo de las características extraídas tanto de la región del núcleo como del citoplasma, un ejemplo de las mismas se muestra en la tabla 1. Sin embargo, las características calculadas no presentan la misma capacidad de discriminación (Plissiti and Nikou, 2012). Para la determinación del conjunto de características más eficaz que se utiliza como entrada en un clasificador, se han propuesto algunos esquemas de selección de características que se refieren a algoritmos genéticos (Marinakis et al., 2009) y al enjambre de partículas de optimización (Marinakis et al., 2008). En el caso de la presente investigación es importante este análisis, debido a que la entrada para el clasificador es una matriz (donde los casos son cada una de las células a evaluar) con cada uno de los rasgos extraídos de las imágenes. (Lorenzo-Ginori et al., 2013)
Sobre la base de lo antes mencionado, se puede concluir que existe un problema abierto: lograr la clasificación correcta de las células empleando solo la información extraída de los núcleos, lo cual comprende entre otras cosas: determinar el subconjunto de rasgos con mejor capacidad de discriminación, y realizar la selección del clasificador que ofrezca mejores resultados.
La extracción de rasgos es uno de los pasos fundamentales en el procesamiento de imágenes debido a que mientras mejor sea la selección de los atributos más acertada será la clasificación final de las células. Esto hace que sea una de las limitantes en las investigaciones para la clasificación de imágenes debido a que si no se cuenta con los rasgos apropiados para clasificar la imagen los resultados obtenidos por el clasificador no serán óptimos tal como se necesita (Lorenzo-Ginori et al., 2013). En investigaciones como es el caso de (Plissiti and Nikou, 2012) se hace uso de los nueve rasgos del núcleo y técnicas como spectral clustering y fuzzy C-means con reducción de la dimensionalidad, a diferencia de la presente investigación que se dirige hacia el uso del clasificador SVMsin reducción donde solo se utilizan cinco rasgos de los nueve como área, perímetro, diámetro corto, diámetro más largo y la redondez. Esta selección de rasgos persigue demostrar que, a partir de un conjunto primario de rasgos geométricos, es posible realizar de forma efectiva la clasificación binaria de imágenes en la prueba de Papanicolaou, mediante la técnica SVM.
MATERIALES Y MÉTODOS
Para la construcción de las matrices de rasgos se hizo uso de las imágenes ya previamente segmentadas (figura 2(b)) pertenecientes a la base de datos Herlev, presentada en (Jantzen et al., 2005). Para ello se hizo uso de la herramienta Matlab y de las funciones propias de la misma. De esta forma se desarrolló un algoritmo que toma de la base de datos Herlev aleatoriamente un 80% de las imágenes y extrae de ellas los rasgos antes mencionados. A continuación se crea una matriz con los rasgos de las imágenes seleccionadas que será la matriz a utilizar para el entrenamiento del clasificador. Posteriormente se extrajeron los rasgos del 20% de las imágenes restantes y se conformó la matriz de rasgos que se utilizará para realizar las pruebas. Estas matrices tienen como característica fundamental que poseen vectores de rasgos diferentes lo que posibilita una evaluación correcta del funcionamiento del clasificador. Se realizaron varias corridas con dicho algoritmo en las que el 20% se fue “rotando” de modo que los conjuntos de entrenamiento y de prueba fueron modificados por cada iteración.
La base de datos Herlev consta de 917 imágenes, donde cada una está compuesta por una única célula (figura 1 (a)), y las muestras se distribuyen irregularmente en siete clases. Tres de ellas son consideradas como normales, y las cuatro restantes como anómalas. La descripción detallada de la base de datos se representa en la tabla 2.
Los subconjuntos dimensionales extraídos servirán como base de entrenamiento y de pruebas para la comparación entre los kernels (lineal, RBF) utilizados por las máquinas de soporte vectorial.
Las Máquinas de Soporte Vectorial (Schölkopf et al., 1999), en inglés Support Vector Machines (SVM), son técnicas de aprendizaje automático. Consisten en construir un hiperplano en un espacio de dimensionalidad muy alta, que separe las clases que se tienen. Una buena separación entre las clases permitirá una clasificación correcta.
Dado un conjunto de datos de entrenamiento ![]() , se desea encontrar el hiperplano óptimo que divida las dos clases de datos. El correspondiente hiperplano puede ser definido como: (Vapnik, 2000)
, se desea encontrar el hiperplano óptimo que divida las dos clases de datos. El correspondiente hiperplano puede ser definido como: (Vapnik, 2000)
![]()
donde x es un vector de datos, WT es el vector de parámetros del modelo y b es un término independiente que ofrece mayor libertad al momento de encontrar el hiperplano óptimo para clasificar los datos.
Dado un conjunto de punto linealmente separables, ilustrados en la figura 3 como cruces y círculos se puede usar la distancia r para calcular un margen de separación p, de la siguiente manera:
![]()
Así para asegurar encontrar el hiperplano, se minimiza con respecto a x y b :
![]()
con la restricción ![]()
Cuando los datos de prueba no son linealmente separables se pueden adoptar dos técnicas para resolver el problema: con optimización “margen suave” y a través de kernel. Para el primer método mencionado se agrega una variable Єi, la cual es usada para registrar la cantidad de errores cometidos por el clasificador en este proceso, quedando la ecuación 3 como sigue:
![]()
con la restricción ![]()
Al utilizar kernel se debe obtener el clasificador óptimo
![]()
donde ɑ es el multiplicador de Lagrage y k(xi,x) es una función kernel. Los kernels comúnmente usados (Cortes and Vapnik, 1995) son:
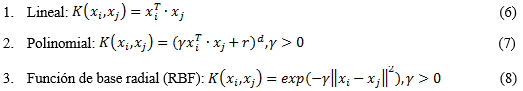
donde y, r y d son parámetros de los kernels.
Métricas de evaluación
La validez de una prueba se define como la habilidad para identificar correctamente aquellos que tienen una enfermedad y aquellos que no la tienen (de Ullibarri Galparsoro and Fernández, 1998). Es muy importante conocer detalladamente la capacidad de las pruebas diagnósticas para clasificar correctamente a los pacientes en categorías o estados en relación con la enfermedad.
La validez de esta prueba posee dos componentes importantes que brindan la exactitud de la misma: la sensibilidad y la especificidad. Para el caso de la presente investigación se utilizaron indicadores para evaluar el rendimiento del clasificador como la sensibilidad, especificidad (Altman and Bland, 1994a), predictividad positiva y negativa (Altman and Bland, 1994b) así como la tasa de clasificación correcta para evaluar el rendimiento del clasificador.
Otra forma de evaluar el rendimiento del clasificador utilizado fue por las curvas ROC. Dicha curva es una representación gráfica de la sensibilidad frente a (1 – especificidad) para un sistema clasificador binario según se varía el umbral de decisión. Se denomina umbral de decisión a aquel que decide si un caso , a partir del vector de salida del clasificador, pertenece o no a cada una de las clases (Burgueño et al., 1995). Los índices de efectividad empleados en esta investigación para evaluar las distancias en el clasificador fueron el área bajo la curva ROC (AUC), la predictividad negativa (Pn) y las medidas F y H, dadas por:

El rendimiento de la clasificación es calculado con el clasificador SVM para poder comparar el desempeño de los kernel lineal y RBF, no se utilizó el kernel polinomial debido a su alto costo computacional respecto a los kernel anteriormente mencionados. Además el rendimiento final se obtiene por la media de los resultados, después de la ejecución de este experimento con 3 modelos diferentes, en un esquema de validación cruzada de 5 iteraciones. Con el fin de estimar la capacidad de discriminación de los parámetros internos de los algoritmos se realizaron una serie de experimentos donde se comparó el rendimiento del mismo con los dos kernels y su resultado con los mismos conjuntos de datos. Como se mencionó anteriormente se realizaron una serie de corridas de la SVM, en cada una de ellas se empleó una base de entrenamiento con los rasgos extraídos de 734 imágenes (de ellas 540 enfermas y 194 sanas) y una base de pruebas compuesta por una matriz que contiene: sanas con 47 imágenes, enfermas con 134 imágenes con el objetivo de evaluar dos subconjuntos diferentes y así obtener valores que muestren la eficiencia real del clasificador.
A pesar de que las pruebas realizadas fueron con conjuntos de datos que contiene 5 rasgos extraídos de los núcleos de las imágenes, se desea que el clasificador no dependa en su funcionamiento de la cantidad de rasgos. Para ello se hace uso de un algoritmo Grid Search que determina los parámetros C y Gamma automáticamente utilizando la validación cruzada sobre la matriz de entrenamiento. Luego guarda el mejor valor de cada uno de los parámetros y lo utiliza en el momento de realizar la clasificación sobre las matrices de prueba. Esto posibilita que se puedan utilizar matrices de rasgos para la clasificación de las imágenes. Se decidió utilizar como apoyo a la implementación la Biblioteca de las Máquinas de Soporte Vectorial (LibSVM).
RESULTADOS Y DISCUSIÓN
Para la discusión de los resultados, se comprobaron los mismos conjuntos de datos para cada kernel Para las comparaciones de los resultados en cada kernels, de los conjuntos de datos obtenidos en el particionamiento, se utilizaron las tres particiones que mejores resultados mostraron en cuanto a las medidas Pn, AUC y las medias armónicas H y F.
Comparación entre los resultados obtenidos por los Kernels
Los resultados de la clasificación se comportan en esta técnica entre un 79% y 86% de predictividad negativa, de la misma manera que la predictividad positiva y el área bajo la curva ROC se mantienen entre rangos de valores que permiten validar la eficiencia del clasificador empleado para cada uno de los conjuntos de datos. Los valores obtenidos de acuerdo a las medidas F y H de igual forma se mantienen entre un 90-92% y 85-91% respectivamente, lo que muestra el nivel de efectividad del clasificador. Al realizar un análisis entre los valores obtenidos al emplear los kernels, estos se mantienen bastante similares con respecto a los valores de Pn. Se decide utilizar el kernel RBF debido a que el tiempo de ejecución del algoritmo con el kernel lineal es significativamente mayor en comparación con el RBF, es decir que mientras más grande sea la matriz de prueba más tiempo demorará el clasificador en mostrar el resultado. El costo computacional fue comprobado a través de la propia ejecución de las pruebas en las que se evidenciaba la demora de la respuesta del kernel linear con respecto al tiempo del kernel RBF.
Al comparar los resultados obtenidos en la investigación con los resultados expuestos en (Plissiti and Nikou, 2012) se evidencia una mejora del método SVM con el kernel RBF respecto a las técnicas spectral clustering y fuzzy C-means en función de H-Mean (Media Armónica)(tabla 9).
CONCLUSIONES
En este trabajo se ha presentado una aproximación a la clasificación binaria de células cervicales (es decir, en normales y anómalas) basada en la utilización de la técnica SVM empleando solo rasgos propios de los núcleos celulares. Fueron utilizados diferentes kernels: lineal y RBF. Para obtener el mejor rendimiento posible de estos kernels se ha realizado un ajuste a la validación cruzada a través de la incorporación del algoritmo Grid Search lo que permitió que los parámetros C y Gamma fueran calculados automáticamente y no sea necesario ponerlos manualmente en su funcionamiento. La implementación del clasificador no depende de la cantidad de rasgos, esto brinda la ventaja de que se pueda analizar el comportamiento de la clasificación con diferentes conjuntos de rasgos sin tener que variar el programa clasificador.
Los resultados obtenidos muestran una mejoría en comparación con los reportados en (Plissiti and Nikou, 2012), en cuanto a los resultados de la media H. Esto se debe a que, a través de esta medidaes posible evaluar el comportamiento de la tasa de falsos negativos, mientras mayor sea el porciento de la media H, menor será la tasa de falsos negativos lo que brinda un buen desempeño en la realización de la prueba de Papanicolaou. Como dirección de trabajo futuro, se implementarán otros clasificadores para realizar un análisis comparativo del desempeño de estos, conjuntamente con los utilizados en esta investigación. De igual forma, se introducirán otros rasgos y se realizará una selección de los mismos basada en su efectividad, con el propósito de reducir la dimensionalidad de las matrices de rasgos sin afectar significativamente el desempeño de los clasificadores. A más largo plazo, se investigará sobre el proceso de clasificación en varias clases para las imágenes de la prueba de Papanicolaou.
REFERENCIAS BIBLIOGRÁFICAS
ALTMAN, D. G. AND J. M. BLAND Statistics Notes: Diagnostic tests 1: sensitivity and specificity. Bmj, 1994a, 308(6947), 1552.
ALTMAN, D. G. AND J. M. BLAND Statistics Notes: Diagnostic tests 2: predictive values. Bmj, 1994b, 309(6947), 102.
BURGUE ÑO, M., J. GARCÍA-BASTOS AND J. GONZÁLEZ-BUITRAGO Las curvas ROC en la evaluación de las pruebas diagnósticas. Med Clin (Barc), 1995, 104(17), 661-670.
CORTES, C. AND V. VAPNIK Support-Vector Networks. Machine Learning, 1995/09/01 1995, 20(3), 273-297.
DE ULLIBARRI GALPARSORO, L. AND P. FERN ÁNDEZ Curvas ROC. Atención Primaria en la Red, 1998, 5(4), 229-235.
HUANG, P.-C., Y.-K. CHAN, P.-C. CHAN, Y.-F. CHEN, et al. Quantitative Assessment of Pap Smear Cells by PC-Based Cytopathologic Image Analysis System and Support Vector Machine. In D. ZHANG ed. Medical Biometrics. Springer Berlin Heidelberg, 2007, vol. 4901, p. 192-199.
JANTZEN, J., J. NORUP, G. DOUNIAS AND B. BJERREGAARD Pap-smear Benchmark Data For Pattern Classification 2005, 9.
LORENZO-GINORI, J. V., W. CURBELO-JARDINES, J. D. L ÓPEZ-CABRERA AND S. B. HUERGO-SUÁREZ. Cervical Cell Classification Using Features Related to Morphometry and Texture of Nuclei. In Progress in Pattern Recognition, Image Analysis, Computer Vision, and Applications. Springer, 2013, p. 222-229.
LORENZO-GINORI, J. V. AND I. RODR ÍGUEZ-SANTOS Aplicación de técnicas de visión computacional en la prueba de Papanicolaou. Medicentro Electrónica, 2012, 16(3), 196-198.
MARINAKIS, Y., G. DOUNIAS AND J. JANTZEN Pap smear diagnosis using a hybrid intelligent scheme focusing on genetic algorithm based feature selection and nearest neighbor classification. Computers in Biology and Medicine, 2009, 39(1), 69-78.
MARINAKIS, Y., M. MARINAKI AND G. DOUNIAS Particle swarm optimization for pap-smear diagnosis. Expert Systems with Applications, 2008, 35(4), 1645-1656.
MAT-ISA, N. A., M. Y. MASHOR AND N. H. OTHMAN An automated cervical pre-cancerous diagnostic system. Artificial Intelligence in Medicine, 2008, 42(1), 1-11.
PLISSITI, M. AND C. NIKOU. Cervical Cell Classification Based Exclusively on Nucleus Features. In A. CAMPILHO AND M. KAMEL eds. Image Analysis and Recognition. Springer Berlin Heidelberg, 2012, vol. 7325, p. 483-490.
PLISSITI, M. E., C. NIKOU AND A. CHARCHANTI Combining shape, texture and intensity features for cell nuclei extraction in Pap smear images. Pattern Recognition Letters, 2011a, 32(6), 838-853.
PLISSITI, M. E., C. NIKOU, MEMBER, IEEE, et al. Automated Detection of Cell Nuclei in Pap Smear Images Using Morphological Reconstruction and Clustering. IEEE TRANSACTIONS ON INFORMATION TECHNOLOGY IN BIOMEDICINE, 2011b, 15(2), 233–241.
RIANA, D. AND A. MURNI 2009. Performance evaluation of Pap smear cell image classification using quantitative and qualitative features based on multiple classifiers. In Proceedings of the International Conference on Advanced Computer Science and Information Systems, ACSIS2009.
SCH ÖLKOPF, B., C. J. C. BURGES AND A. J. SMOLA Advances in Kernel Methods: Support Vector Learning. Edtion ed.: MIT Press, 1999. ISBN 9780262194167.
VAPNIK, V. N. The Nature of Statistical
Recibido: 01/02/2015
Aceptado: 15/02/2015