Meu SciELO
Serviços Personalizados
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Cubana de Ciencias Informáticas
versão On-line ISSN 2227-1899
Rev cuba cienc informat vol.11 no.3 La Habana july.-set. 2017
ARTÍCULO ORIGINAL
Taxobanger v.1.0: Aplicación informática en R para el análisis taxonómico en bancos de germoplasma vegetal
Taxobanger v.1.0: Computer application in R for the taxonomic analysis in plant germplasm banks
Osmany Molina Concepción1*, Marilys Milián Jiménez1, Carmen C. Pons Pérez1, Lianet González Díaz1, Ricardo Grau Abalo2
1Instituto de Investigaciones de Viandas Tropicales (INIVIT), Cuba. INIVIT, Apartado 6, Santo Domingo, CP: 53 000, Villa Clara, Cuba
2Universidad Central “Marta Abreu” de Las Villas (UCLV), Cuba
*Autor para la correspondencia: taxonumeric@inivit.cu
RESUMEN
Los recursos fitogenéticos se han convertido en una prioridad científica, lo cual hace importante el análisis de esta diversidad mediante métodos cuantitativos que ayuden a agrupar poblaciones de un mismo género o especie. Con el objetivo de desarrollar una aplicación informática para el análisis taxonómico en las colecciones de germoplasma que se conservan en el Instituto de Investigaciones de Viandas Tropicales, se utilizó el lenguaje y entorno de programación para análisis estadístico y gráfico R. Se logró la aglutinación en un solo programa de funciones que permiten la obtención y manipulación de datos, la definición del nivel de medición de las variables y la ejecución de cuatro estrategias de clasificación que definen diferentes estructuras de datos a partir de la combinación de técnicas estadísticas que determinan aquellos conglomerados que se ajustan mejor a las características de las colecciones del germoplasma. Además, esta aplicación permite a los curadores de bancos de germoplasma contar con una herramienta informática que ayuda a la clasificación de la variabilidad conservada a partir de los rasgos que le caracterizan, sin necesidad de poseer amplios conocimientos en las técnicas estadísticas y computacionales. Esto es altamente cualificado debido a que ahorra tiempo en el procesamiento estadístico y brinda mayor visión y fiabilidad en los resultados obtenidos. El trabajo desarrollado favorece el manejo de la biodiversidad conservada en los bancos de germoplasma y amplía su utilización en el mejoramiento genético de las especies vegetales, pues permite un mejor uso de la información y una manipulación eficiente del material genético que se posee.
Palabras clave: clasificación, software libre, programación en lenguaje R, recursos fitogenéticos
ABSTRACT
Plant genetic resources have become a scientific priority, which makes it so important to analyze this diversity through quantitative methods that help to group populations of the same genus or species. The language and programming environment for statistical analysis and graph known as R, was used in order to develop an application software for the taxonomic analysis of the germplasm collections preserved in the Institute of Research of Tropical Root and Tuber Crops. Agglutination was achieved in a single program of functions that allows the collection and manipulation of data, the definition of the level of measurement of variables and the execution of four strategies for classification. These strategies define different data structures from the combination of statistical techniques that determine those conglomerates that better match the characteristics of germplasm collections. In addition, this application software allows the curators of germplasm banks to have a computer tool that helps in the classification of preserved variability according to its characters, no need to have an extensive knowledge in the statistics and computational techniques. This is highly qualified, because it saves time in statistical processing and provides broader vision and reliability in the obtained results. The present research favors the management of the biodiversity preserved in germplasm banks and extends its use in the genetic improvement of plant species, since it allows a better use of the information and an efficient handling of the genetic material.
Key words: Classification, free software, R programming language, genetic resources
INTRODUCCIÓN
Aunque la interacción de la estadística matemática y la biología no es algo nuevo, el extraordinario desarrollo de la informática en los últimos tiempos ha contribuido a popularizar el empleo de novedosos y más complejos métodos de análisis e interpretación. Un ejemplo de lo anterior lo constituye la taxonomía numérica a partir del análisis multivariado que se le realiza a los elementos morfoagronómicos, insoenzimáticos y moleculares de las colecciones de germoplasma.
Los análisis estadísticos ponen a disposición un conjunto de herramientas muy amplio, muchas de las cuales han sido desarrolladas desde hace algún tiempo, pero que han experimentado en los últimos años una importante revolución con el planteamiento de nuevos métodos, sobre todo motivados por la utilización de programas computacionales como herramientas de cálculo.
La necesidad de comprender las relaciones entre muchas variables hace de los métodos multivariados una técnica inherentemente compleja (Bramardi, 2002), además se necesita mayor cantidad de cálculos matemáticos para aplicarlos que para hacer inferencias en un conjunto univariado de datos, por tanto, es obvio que para ejecutar estos tipos de técnicas se hace imprescindible el uso de un software que, no solo efectúe los algoritmos de cálculo, sino que guíe al usuario que no es experto en estadística en esta complicada tarea y le ayude a interpretar los resultados.
En términos generales, el análisis multivariado se refiere a todos aquellos métodos estadísticos que analizan simultáneamente medidas múltiples (más de dos variables) de cada individuo (Hair et al., 1992). En la actualidad, los bancos de germoplasma de Cuba y de otros países poseen su documentación, donde se recogen datos morfoagronómicos y características detectables visualmente que sólo se expresan como reacción a estímulos del medio ambiente, denominadas de evaluación; pero la mayoría de sus curadores no tienen los conocimientos estadísticos ni las herramientas informáticas necesarias para llevar a cabo una taxonomía numérica específica para estos casos, haciendo un mejor uso de la información extraída y una manipulación eficiente del material genético que poseen (Franco y Hidalgo, 2003).
Los datos se pueden analizar mediante el empleo de métodos simples o complejos, que van desde el uso de gráficos y estadísticos de tendencia central y dispersión hasta los multivariados. El análisis tiene el propósito de reducir el volumen de información en trabajos de esta naturaleza y llegar a conclusiones acerca de la variabilidad y la utilidad del germoplasma, por tanto, los datos deben representar fielmente las características de las accesiones.
Existen actualmente gran variedad de programas computacionales relacionados con el almacenamiento de datos de bancos de germoplasma, así como programas estadísticos propietarios utilizados frecuentemente en el análisis y obtención de resultados, pero el empleo eficiente de estos últimos requiere de la integración a un alto nivel de conocimientos estadísticos e informáticos para manipular correctamente sus potencialidades e interpretar adecuadamente los resultados obtenidos por parte de los curadores y especialistas en mejoramiento genético.
Es innegable que el análisis estadístico con aplicaciones informáticas presenta ventajas para el usuario: se disponen los datos en una estructura tabular o en una matriz de datos, se permiten varias vías rápidas y muy seguras para detectar errores, se logra simplicidad de ejecución y precisión de los cálculos obtenidos, se hace viable la manipulación de voluminosas bases de datos, así como una rápida reproducibilidad de los análisis estadísticos, se proporciona amplia facilidad y flexibilidad en el manejo de los gráficos.
En los últimos años, ha surgido con fuerza como alternativa de software libre en muy distintos ambientes docentes y de investigación, un lenguaje de programación especialmente indicado para el análisis estadístico, denominado R (Ihaka y Gentleman, 1996).
El R (R Development Core Team, 2017b) es un lenguaje de programación principalmente orientado al análisis estadístico y visualización de información cuantitativa y cualitativa. Fue oficialmente presentado en 1997 y es un software libre que se rige por la licencia general pública (“General Public License” o GPL) de la fundación de software libre (“Free Software Foundation” o GNU). Una de sus grandes fortalezas es que puede ser ampliado mediante paquetes que extienden sus funcionalidades. Actualmente hay más de 10683 paquetes publicados con licencias libres y disponibles en un repositorio general (R Development Core Team, 2017a). A diferencia de la mayoría de los programas que tienen interfaces tipo ventana, R es manejado a través de una consola en la que se introduce comandos propios de su lenguaje para acceder a todos los procedimientos, sin embargo, es posible diseñar interfaces gráficas para facilitar la interacción con los usuarios no familiarizados con su consola (Salas, 2008).
R permite la interconexión con otros softwares de código abierto como el Java y el Weka, (Waikato Environment for Knowledge Analysis) lo que aumenta sus potencialidades de una manera impresionante.
El objetivo de este trabajo fue crear una interfaz en lenguaje Rque facilite realizar la taxonomía numérica en bancos de germoplasma con vista a ayudar a los curadores en la interpretación y extracción de información útil a partir de los datos disponibles. Se logró la aglutinación en un solo programa de funciones que permiten la obtención y manipulación de datos, la definición del nivel de medición de las variables y la ejecución de cuatro estrategias de clasificación que definen diferentes estructuras de datos a partir de la combinación de técnicas estadísticas que determinan aquellos conglomerados que se ajustan mejor a las características de las colecciones del germoplasma.
MATERIALES Y MÉTODOS
El trabajo se desarrolló por el grupo de Bioinformática del Instituto de Investigaciones de Viandas Tropicales (INIVIT), partiendo de un diseño logrado sobre el análisis de los requerimientos, que son el conjunto de técnicas y procedimientos utilizados para conocer los elementos necesarios a definir y especificar su función, vincular la interfaz con otros elementos del sistema y establecer los enlaces de diseño que debe cumplir el programa.
Los requerimientos fueron expuestos por investigadores que manejan los bancos de germoplasma y que tienen previa experiencia en el desarrollo de los análisis multivariados aplicados a la taxonomía numérica mediante otros programas estadísticos. A partir de esto fueron seleccionados los siguientes análisis que incluyen métodos de ordenamiento y clasificación de: conglomerados, discriminantes, componentes principales y correspondencia múltiple. Y como eje fundamental de la aplicación cuatro variantes combinatorias de datos, que permite mezclar datos cualitativos y cuantitativos.
El diseño de la aplicación fue logrado sobre el análisis de los requerimientos, que son el conjunto de técnicas y procedimientos utilizados para conocer los elementos necesarios a definir y especificar su función, vincular la interfaz con otros elementos del sistema y establecer los enlaces de diseño que debe cumplir el programa.
El procesamiento incluye un sistema de asistencia al usuario final que le sugiere los pasos a seguir en el análisis de sus datos y ayuda a interpretar los resultados, proporcionándole opiniones sobre la forma más correcta de proceder, mediante el cálculo de índices y la visualización a través de tablas y gráficos de los resultados. Para lograr este sistema se confrontó con criterios dados por los especialistas de los bancos de germoplasma y se aprovecharon los conocimientos de expertos en estadística.
La programación de los algoritmos se ejecutó en el lenguaje R, utilizando sus amplias potencialidades al tener disponible en su sitio en Internet una gran cantidad de paquetes con una vasta variedad de funciones que se pueden utilizar en la programación. Entre estos paquetes está el tcltk2 (Grosjean, 2012) que incluye múltiples elementos utilizados en la confección de la interfaz de usuario, entre ellos: ventanas, menús, barras de desplazamiento, botones, botones radiales, cuadros de texto, etiquetas de texto, cuadros de selección, cajas de listas, conjuntos, entre otros. Además fueron incluidas funciones pertenecientes a otros paquetes tales como: XLConnect (Studer, 2017), RODBC (Ripley y Lapsley, 2017), FactoMineR (Sebastian, 2008), GPArotation (Bernaards y Jennrich, 2005), cluster (Maechler et al., 2017), clusterSim (Walesiak y Dudek, 2017), clue (Hornik, 2017) y los paquetes stats que forma parte de la librería básica de R que se instala por defecto.
Para la ejecución de la interfaz debe instalarse primeramente la consola de R y a partir de ella, tecleando una instrucción (source (“start.r”)), se llama al fichero principal que contiene la ventana de entrada; pero también se puede conformar el fichero de configuración del R, que está dentro de la carpeta donde este software se instala (Rprofile.site), para que automáticamente se haga la ejecución. Se requiere de una plataforma WindowsÒ 9x o superior y capacidad en disco duro de 750 Mb como mínimo.
RESULTADOS Y DISCUSIÓN
Como resultado se obtuvo una interfaz con un diseño atractivo, amistoso y de fácil manipulación, de forma tal que realmente constituye una herramienta eficaz en manos de los usuarios potenciales y les ayuda en la conservación del germoplasma y en el uso más eficiente de los recursos fitogenéticos.
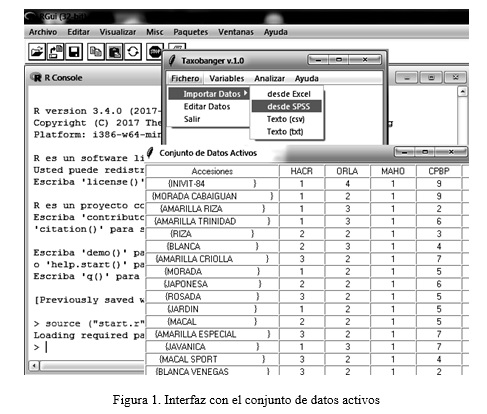
La interfaz está compuesta por una ventana principal con una barra de menús en la parte superior y al usuario cargar los datos. La primera opción de la barra de menús “Fichero” permite abrir el fichero de datos en formatos: Excel (.xls o .xlsx), SPSS (.sav) y fichero de texto (.csv o txt), además tiene una opción Editar Datos una vez cargados en memoria (Figura 1).
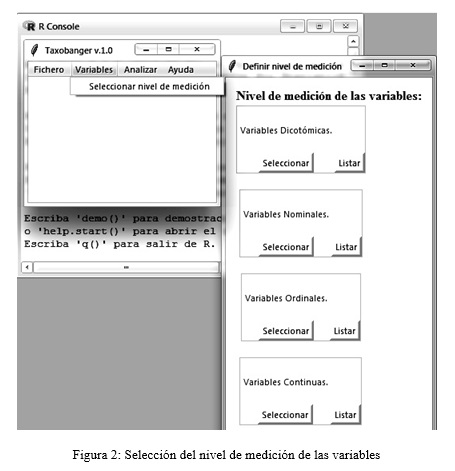
A través de la segunda opción de la barra de menús “Variables” se establecen los niveles de medición de las variables que pueden ser: dicotómicas, nominales, ordinales y continuas (Figura 2).
Mediante la opción “Analizar” de la barra de menús se accede a los análisis multivariados. En la ejecución de los métodos se muestran los resultados en forma de tablas o gráficos, junto a sugerencias o interpretaciones ofrecidas, con vista a ayudar al curador a comprender las salidas obtenidas.
Estos métodos se encuentran catalogados en dos tipos: de Reducción de datos y Clasificar. Dentro de los métodos de reducción de datos se encuentran: Análisis de Correspondencias Múltiple y Análisis de Componentes Principales.
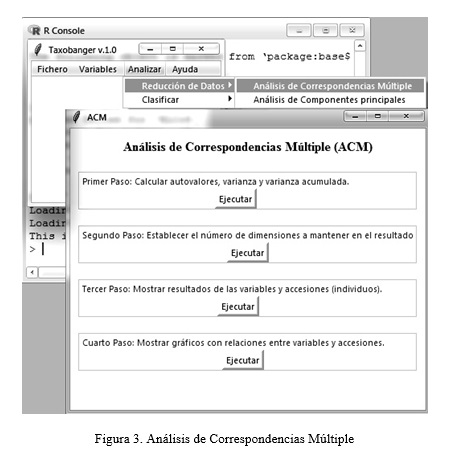
Análisis de Correspondencias Múltiple
Para este análisis se pueden seleccionar variables discretas (ordinales y nominales). Inicialmente se calculan los autovalores, la varianza y la varianza acumulada para poder definir el número de componentes, luego se muestran los resultados en tablas acompañados de gráficos que ayudan al usuario al razonamiento estadístico y permiten observar la correspondencia entre las variables (Figura 3).
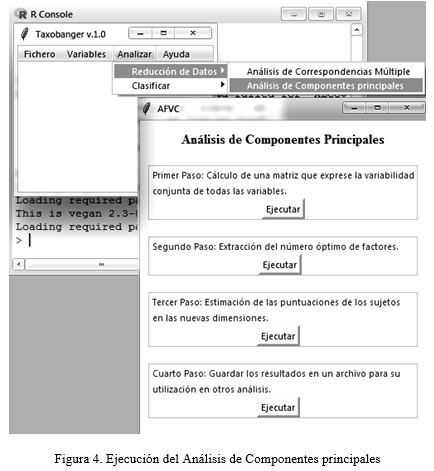
Análisis de Componentes Principales
Para este análisis se pueden seleccionar solo variables continuas, primeramente, se calcula la matriz de correlaciones con el propósito de analizar si las variables están correlacionadas y por tanto es factible llevar a cabo el análisis. Luego se selecciona el número de factores óptimos, a través de métodos analíticos y visuales (Figura 4). Por último, se ofrecen las puntuaciones factoriales para cada individuo junto a varios gráficos que permiten analizar el comportamiento tanto de las variables como de los individuos en las nuevas dimensiones.
Dentro de los métodos de clasificación están programados: Análisis de Conglomerados y Conglomerados Mixto.
Análisis de conglomerados para variables continuas
Para este análisis se pueden seleccionar variables continuas, dentro de las que se pueden incluir las contribuciones obtenidas para los individuos en las nuevas dimensiones a través de uno de los métodos anteriores de reducción de datos. En primer lugar, se lleva a cabo una conglomeración jerárquica y para esto, primeramente, se normalizan las variables, posteriormente se selecciona la medida de distancia mediante uno de los métodos contemplados y se comparan los resultados en dependencia del coeficiente de correlación cofenético obtenido para identificar cual es más idónea en el conjunto de datos que se procesa. En el próximo paso se establece mediante un proceso iterativo el método de conglomeración más adecuado (Figura 5) y el número de conglomerados óptimo, calculando índices y analizando las salidas a través de gráficos. Por último, se efectúa una conglomeración particional con el número de conglomerados anteriormente establecido y se obtiene el conglomerado a que pertenece cada individuo.
Conglomerados para variables mixtas
La meta principal de esta aplicación es la concepción de cuatro variantes combinatorias de datos, que permite mezclar datos cualitativos y cuantitativos (Figura 6).
Con las bases de datos de prueba de las colecciones de malanga (Xanthosoma spp.), ñame (Dioscorea spp.) y plátanos (Musa spp.) que se conservan del Instituto de Investigaciones de Viandas Tropicales (INIVIT), se llevaron a cabo cuatro variantes combinatorias de datos para probar la eficacia de éstos con la estructura de análisis propuesta a partir de un conjunto de técnicas estadísticas para determinar los conglomerados que se ajustan mejor a las características del germoplasma en estudio.
En la primera variante se realizó un estudio de la métrica de (Gower, 1971) para variables mixtas donde se integran las variables cualitativas (nominales y ordinales) y las cuantitativas, la misma está implementada en la función gowdis(), descrita en el paquete FD (Laliberté y Legendre, 2010; Laliberté et al., 2015). En la segunda se integraron las variables cualitativas a las cuantitativas codificadas (según Sistema de Descriptores Mínimos) tratadas como ordinales. En esta variante, las características cuantitativas son llevadas a rangos establecidos para estas colecciones, está transformación reduce el impacto de los ouliers (valores fuera de rango) en los datos (Milligan y Cooper, 1988). A la matriz de datos resultantes se le aplicó la métrica de Gower.
En la variante 3 se integraron las variables cualitativas transformadas a través del análisis de correspondencia múltiple (Tenenhaus y Young, 1985) con la función MCA() descrita en el paquete FactoMineR, tomando las dimensiones que acumularon una varianza superior a uno con las cuantitativas normalizadas para lo cual se usó la función data.Normalization() del paquete clusterSim con transformación por puntuaciones Z (z-score). Posteriormente se unieron ambos resultados en una sola matriz de datos continuos. En la cuarta variante a las variables cualitativas se le realizó un procedimiento similar al empleado en la variante tres, y con las variables cuantitativas después de estandarizada con la función data.Normalization() del paquete clusterSim se realizó un Análisis de Componentes Principales mediante la función precomp()del paquete stats, se tomaron las mayores contribuciones a los ejes factoriales de las accesiones. Con la unión de ambos resultados se conformó una matriz de datos continuos. Como medida para evaluar las diferencias y similitudes entre objetos para variables cuantitativas sobre las variantes tres y cuatro, se usó la distancia Euclídea de la función dist() descrita en el Paquete stats.
Para el análisis de las cuatro variantes combinatorias de datos se usaron datos procedentes de un estudio de los diferentes genotipos de las colecciones de ñame (Dioscorea spp.), malanga (Xanthosoma spp.) y plátanos (Musa spp.) que se conserva en el Instituto de Investigaciones de Viandas Tropicales (INIVIT).
En esta investigación se usaron los métodos de aglomeración jerárquicos de Ward o varianza mínima (Minimum Variance Clustering (Ward, 1963), Promedio ó UPGMA (Unweighted Pair-Group Method using Arithmetic Averages) (Sneath y Sokal, 1973), agrupación de enlace simple (Single Linkage Agglomerative Clustering)(Gower, 1967), agrupación de enlace completo (Complete Linkage Agglomerative Clustering) (Sorensen, 1948) con la función hclust() en el paquete stats que forma parte de los paquetes de R que se instalan por defecto. También se encuentra el paquete cluster (Maechler et al., 2005) que amplía la gama de análisis de conglomerados, pues incluye además métodos particionales.
En la búsqueda de mejores algoritmos de clasificación aparece una tendencia a combinar varios algoritmos de agrupamiento en el mismo problema. La base de estos algoritmos está en la lógica de utilizar el criterio de varios expertos y combinarlos en aras de lograr un mejor rendimiento.
El paquete clue (Hornik, 2017) permite crear y analizar combinación de agrupamientos, para ambas representaciones de los datos: jerárquica y no jerárquica y obtener una estructura consenso. Para aglutinar los resultados de los diferentes algoritmos de aglomeración se usó la función cl_ensemble() de este paquete, en aras de lograr una mejor calidad de los resultados alcanzados por los algoritmos individuales y compensar posibles errores cometidos en el desempeño de cada uno. El árbol consenso, resultado de combinar toda la información de los diferentes árboles en un dendrograma final, se obtuvo en dos variantes, utilizando las distancias Euclidean, Manhattan y Majority disponibles en esta función para los algoritmos jerárquicos.
Al finalizar cada análisis multivariado, se ofrece la posibilidad de salvar los resultados en un fichero de datos, que puede ser posteriormente cargado y utilizado en procesamientos posteriores.
De esta forma, los curadores cuentan con una herramienta informática que les ayuda a escoger la mejor variante de procesamiento, sin necesidad de poseer amplios conocimientos, ni en las técnicas estadísticas ni en las computacionales. Lo que anteriormente podían hacer mediante varios programas estadísticos específicos por separado y dominando a fondo cómo manipular los datos, cómo trabajar con un software para profesionales en estadística e interpretar los resultados, ahora se encuentra aglutinado en uno solo, que además les brinda una metodología de desarrollo de los análisis y les facilita la comprensión. Esto debe ser altamente apreciado debido a que les ahorra tiempo en el procesamiento estadístico y brinda mayor visión y fiabilidad en los resultados obtenidos.
CONCLUSIONES
Se obtuvo una aplicación informática que permite la obtención y manipulación de datos, la definición del nivel de medición de las variables y la ejecución de métodos multivariados de reducción de datos y clasificación.
La combinación de las técnicas estadísticas empleadas en las cuatro variantes combinatorias de datos, por su flexibilidad, pueden ser aplicadas a otros estudios de clasificación en bancos de germoplasma vegetal.
La aplicación informática es un software libre, lo cual amplía grandemente sus posibilidades de acceso y distribución.
REFERENCIAS BIBLIOGRÁFICAS
BERNAARDS, C. A. y ROBERT I. J. Gradient Projection Algorithms and Software for Arbitrary Rotation Criteria in Factor Analysis. Educational and Psychological Measurement, 2005, 65: 676–96.
BRAMARDI, S. J. Análisis Multivariado. Su Aplicación En La Caracterización de Recursos Genéticos. Facultad de Ciencias Agrarias, Univ. Conahue, Estación Exp. INTA. Argentina, 2002, 60.
FRANCO, T. L. y HIDALGO, R. Análisis Estadístico de Datos de Caracterización Morfológica de Recursos Fitogenéticos. Boletín Técnico IPGRI 8. Cali, Colombia: Instituto Internacional de Recursos Fitogenéticos (IPGRI). 2003. 89p.
GOWER, J. C. AGeneral Coefficient of Similarity and Some of Its Properties. Biometrics, 1971, 27: 857–71.
GOWER, J. C. AComparison of Some Methods of Cluster Analysis. Biometrics, 1967, 23: 623–28.
GROSJEAN, P. SciViews: A GUI API for R. Belgium: UMONS, 2012. Fecha de consulta: 12 de abril 2017, Disponible en: https://cran.r-project.org/web/packages/tcltk2/index.html.
HAIR, J. F.; ANDERSON, R. E.; TATHAM, R.L. y BLACK, W.C. Multivariate Data Analysis. Nueva York: MacMillan Publ. Co. 1992,544p
HORNIK, K. “Clue: Cluster Ensembles. R Package Version 0.3-53.” 2017. Fecha de consulta: 17 de febrero 2017, Disponible en: https://CRAN.R-project.org/package=clue.
SEBASTIEN, L.; JOSSE, J. y HUSSON, F. FactoMineR: An R Package for Multivariate Analysis. Journal of Statistical Software, 2008, 25(1), 1-18. DOI:10.18637/jss.v025.i01.
IHAKA, R. y GENTLEMAN, R. R: A Language for Data Analysis and Graphics. Journal of Computational and Graphical Statistics, 1996, 5: 299–314.
LALIBERTÉ, E. y LEGENDRE, P. A distance-based framework for measuring functional diversity from multiple traits. Ecology, 2010, 91:299-305.
LALIBERTÉ, E.; LEGENDRE, P. y SHIPLEY, B. FD: Measuring Functional Diversity from Multiple Traits, and Other Tools for Functional Ecology. R Package Version 1.0-12. 2015. Fecha de consulta: 22 diciembre de 2016, Disponible en: https://cran.r-project.org/web/packages/FD/FD.pdf.
MAECHLER, M.; ROUSSEEUW, P.; STRUYF, A.; HUBERT, M. y HORNIK, K. Cluster: Cluster Analysis Basics and Extensions. R Package Version 2.0.6.2017. Fecha de consulta: 27 marzo de 2017, Disponible en: https://cran.r-project.org/web/packages/cluster/index.html.
MILLIGAN, G W. y COOPER, M. C. A Study of Standardization of Variables in Cluster Analysis. Journal of Classification, 1988,5: 181–204.
R DEVELOPMENT CORE TEAM. Contributed Packages. 2017a. Fecha de consulta: 17 junio de 2017, Disponible en: https://cran.r-project.org.
R DEVELOPMENT CORE TEAM. R: A Language and Environment for Statistical Computing (version 3.4.0). R Foundation for Statistical Computing. Vienna, Austria. 2017b. Fecha de consulta: 7 abril de 2017, Disponible en: http://www.r-project.org/.
RIPLEY, B. y LAPSLEY, M. RODBC: ODBC Database Access. 2017. Fecha de consulta: 23 de mayo 2017, Disponible en: https://cran.r-project.org/web/packages/RODBC/RODBC.pdf.
Salas, C. ¿Por Qué Comprar Un Programa Estadístico Si Existe R?,Ecología Austral, 2008, Vol. 18: 223–31.
SNEATH, P.H. A. y SOKAL, R. R. Numerical Taxonomy. The Principles and Practice of Numerical Classification. 1973. San Francisco, California, USA: W. H. Freeman and Co.
SORENSEN, T. A. AMethod of Establishing Groups of Equal Amplitude in Plant Sociology Based on Similarity of Species Content and Its Application to Alalysies of Vegetation on Danish Commons. Biologiske Skrifter, 1948, 5: 1–34.
STUDER, M. Excel Connector for R. 2017. Fecha de consulta: 23 de mayo 2017, Disponible en: https://cran.r-project.org/web/packages/XLConnect/index.html.
TENENHAUS, M. y YOUNG, F.W. An Analysis and Synthesis of Multiple Correspondence Analysis, Optimal Scaling, Dual Scaling, Homogeneity Analysis and Other Methods for Quantifying Categorical Multivariate Data.Psychometrika, 1985, 50(91). doi:10.1007/BF02294151.
WALESIAK, M. y DUDEK, A. clusterSim: Searching for Optimal Clustering Procedure for a Data set. 2017. Fecha de consulta: 3 de abril 2017, Disponible en: http://cran.es.r-project.org/web/packages/clusterSim/clusterSim.pdf.
WARD, J.H. Jr. Hierarchical Grouping to Optimize an Objective Function. Journal of the American Statistical Association, 1963, 58: 236–44.
Recibido: 20/05/2017
Aceptado: 25/06/2017