Introducción
Los coronavirus constituyen una familia de virus presentes en ciertos animales, que al mutar se convierten en un agente infeccioso capaz de provocar enfermedades graves en los seres humanos, tales como el síndrome respiratorio agudo severo (SARS) y el síndrome respiratorio del oriente medio (MERS) en el medio oriente y recientemente en China: el SARS-COV2, que provoca la COVID-19, la cual ha causado la muerte de miles de personas en todo el mundo, lo que ha determinado que la Organización Mundial de la Salud la haya decretado como una pandemia. El SARS-COV-2 se transmite de persona a persona y provoca fundamentalmente afectaciones respiratorias que pueden llegar hasta la muerte de los infectados. La alta transmisibilidad del virus y la existencia de pacientes asintomáticos son factores que complejizan considerablemente la contención del virus. El análisis detallado de una cadena de contagio pudiera aportar elementos de utilidad tanto para los análisis virológicos como epidemiológicos. Por ejemplo, se podría verificar la presencia de ciertos síntomas en determinada cadena y compararlos con los síntomas de otra cadena de contagio. De existir diferencias se podría analizar un grupo de causas que pudieran ser biológicas, sociales o ambientales. Por ejemplo, se podría tratar de cepas diferentes del virus.
El análisis de las cadenas de contagio también podría ayudar a constatar las modificaciones (si ocurren) que sufre el virus al ser transmitido, tanto por la incidencia del trasmisor como del ambiente en que transcurre. Además, se podrían efectuar análisis temporales, a partir de determinar el tiempo trascurrido entre el primer caso de la cadena y el último. Este análisis pudiera ser útil en la definición de estrategias, con el propósito de reducir el contagio en la población, al estar incluido en este tiempo el periodo de transmisibilidad o más elevada virulencia en los infectados. Sin embargo, durante el análisis, el alto número de contagio determina que las cadenas sean extensas, lo que hace más complejo el trabajo de los investigadores. Esta situación provoca que sea difícil realizar un análisis con profundidad y que se dificulte la caracterización de la enfermedad. Además, la falta de herramientas que apoyen el desempeño de los investigadores determina que su trabajo no se pueda realizar con la agilidad que demanda la actual pandemia. Por lo tanto, en esta investigación se aborda el problema de cómo representar la información asociada a las cadenas de contagio de COVID-19 de manera que se agilice su análisis y se puedan obtener rasgos que contribuyan a caracterizar la enfermedad.
En la búsqueda de enfoques para tratar el problema planteado se conoció de la utilidad de las ontologías para la representación del conocimiento en diversos dominios.1,2,3,4,5,6,7) La representación del conocimiento mediante ontologías permite explotar la capacidad de razonamiento sobre estas. En este sentido, se puede validar la información y además se puede realizar el análisis automatizado y llegar a inferir un nuevo conocimiento.
La revisión de la literatura permitió identificar un grupo de investigaciones que utilizan ontologías para contribuir al dominio de la epidemiología. Pesquita y otros8 desarrollaron un modelo ontológico para apoyar el trabajo epidemiológico. Sin embargo, a pesar del notable aporte en esta investigación, su contribución se enfoca en la representación de indicadores que son de interés epidemiológico que no habían sido considerados en ontologías previas.
Hulo y otros9 siguieron un enfoque ontológico para apoyar los estudios virológicos. En este caso, el estudio se centra en formalizar la representación del ciclo de vida de los virus, para lo que incluye en las ontologías los términos más relevantes asociados con el área de estudio. Otros trabajos han adoptado enfoques ontológicos para analizar enfermedades infecciosas o comportamiento de virus en plantas y animales.10 Los trabajos revisados han permitido comprobar las potencialidades de las ontologías para representar y analizar información del área de la epidemiología o del análisis virológico. Además, se pudo constatar que las ontologías revisadas no se enfocan en el análisis de las cadenas de transmisión. A partir de estas observaciones, en esta investigación se desarrolla una ontología con el propósito de superar esta brecha. El objetivo de este trabajo fue presentar un modelo ontológico para la representación y el análisis de las cadenas de contagio por COVID-19. De esta manera, hemos querido contribuir a mejorar el análisis sobre las características de la enfermedad. La contribución fundamental de esta investigación radica en que el análisis se enfoca fundamentalmente en las cadenas de contagio, a diferencia de trabajos precedentes que se enfocan en los casos particulares. Los resultados de este enfoque pudieran ser de utilidad para el trabajo epidemiológico, especialmente en países como Cuba, donde el riguroso seguimiento de las cadenas de contagio ha demostrado ser una medida efectiva de contención del virus.
Métodos
La ontología se desarrolló siguiendo una metodología reconocida en la comunidad científica y ampliamente utilizada. Además, se validaron sus condiciones como sistema lógico-formal a partir del uso de razonadores.
El concepto de ontología ha sido ampliamente estudiado en la comunidad científica. Una ontología especifica un vocabulario relativo a un cierto dominio. Este vocabulario define entidades, clases, propiedades, predicados y funciones, además de las relaciones entre estos componentes.11,12,13,14) La ingeniería ontológica permite la representación de conocimiento, así como la identificación de un contexto y la verificación de la dependencia de información más fácilmente que con el uso del modelado de datos o bases de datos.
Dentro de los lenguajes para especificar ontologías se destacan: Ontolingua, XML Schema, RDF (Resource Description Framework), RDF Schema (o RDF-S) y OWL (Web Ontology Language). OWL se basa en un modelo lógico que hace posible definir y describir los conceptos.
El modelo OWL se distingue por su conjunto de operadores: intersección, unión y negación. Está basado en un modelo lógico que permite definir los conceptos tal y como son descritos. Además, la posibilidad de utilizar razonadores permite chequear automáticamente la consistencia de los modelos representados. Estas ventajas determinaron que en esta investigación se asuma OWL como el lenguaje para la representación de las ontologías.
Para la creación de la ontología se utilizó la herramienta Protégé, que es un editor de código abierto desarrollado en la Universidad de Stanford,15 multiplataforma, usado para construir ontologías, y posee una arquitectura flexible y extensible. Es reconocida como una de las herramientas más utilizadas para la ingeniería ontológica. Las ontologías creadas en Protégé pueden exportarse en varias sintaxis, tales como RDF/XML y OWL/XML. En el proceso de inferencia se pueden utilizar los razonadores FaCT++, Hermit y Pellet.
Es crucial guiarse por una metodología sólida para obtener una ontología de calidad. Por lo tanto, se analizaron varias metodologías.11,16,17) El desarrollo de la ontología que se presenta en esta investigación estuvo guiado por la metodología propuesta por Noy y McGuinness, (15 la cual ha sido ampliamente adoptada. Incluye los siguientes pasos: determinar el dominio y el alcance de la ontología, considerar reutilizar ontologías existentes, enumerar los términos relevantes de la ontología, definir las clases y la jerarquía de clases, definir las propiedades (llamadas relaciones o slots) de las clases, definir facetas y/o restricciones sobre los slots o relaciones y finalmente definir instancias.
Resultados
La ejecución de la metodología descrita en la sección anterior permitió obtener una ontología para la descripción y el análisis de cadenas de contagio de COVID-19. A continuación se describen los resultados alcanzados al desarrollar las seis actividades que concibe la metodología adoptada:
Paso 1: Determinar el dominio y el alcance de la ontología. La ontología tiene el objetivo de proveer los mecanismos para representar y analizar información referente a pacientes enfermos con la COVID-19. Se hace énfasis en las cadenas de contagio, con el propósito de mostrar información que pudiera ser útil en los estudios que se desarrollan sobre la enfermedad. Se parte de la premisa de que cierta información sobre pacientes de forma aislada pudiera ser de poca relevancia, pero si el análisis se realiza sobre una cadena de contagio se pudieran arribar a conclusiones de interés para la comunidad científica y para las autoridades sanitarias y gubernamentales que luchan por la contención de la COVID-19. Se identificaron patrones cuya presencia pudiera ameritar un estudio más detallado para estudiar posibles causas o implicaciones. Para precisar el objetivo de la ontología se formularon diez preguntas de competencia (PC):
PC1. ¿Qué pacientes pertenecen a una cadena de contagios?
PC2. ¿Qué personas han contagiado directa o indirectamente a otra?
PC3. ¿Qué cadenas incluyen fallecidos?
PC4. ¿Qué cadenas con personas de un solo sexo existen?
PC5. ¿Qué cadenas incluyen pacientes de diferentes municipios?
PC6. ¿Qué cadenas incluyen pacientes pediátricos?
PC7. ¿Cuáles son las cadenas extensas?
PC8. ¿Qué cadenas existen donde todos sus miembros se hayan recuperado de la enfermedad?
PC9. ¿Qué cadenas presentan solo personas asintomáticas?
PC10. ¿Qué tiempo se ha mantenido activa una cadena determinada?
Paso 2: Reutilizar ontologías existentes. Cuando se crea una ontología es conveniente comprobar si se pueden reutilizar conceptos de otras. En la sección II se verificó que -aunque existen ontologías para describir aspectos epidemiológicos- no se enfocan en las cadenas de contagio. Por lo tanto, no se reutilizaron elementos ontológicos. Sin embargo, la integración con otras ontologías es una de las líneas futuras de la investigación.
Paso 3: Enumerar los términos relevantes de la ontología. A partir de la revisión de la literatura, así como del continuo seguimiento de la información referente a la enfermedad, se identificó un conjunto de términos que resultan imprescindibles para modelar la ontología. Entre los términos más relevantes se encuentran: persona, paciente, cadena de transmisión, estado del paciente, fecha de contagio, síntomas de la enfermedad, entre otros. Estos términos de la realidad constituyen la base para definir los elementos estructurales de la ontología.
Paso 4: Definir las clases y la jerarquía de clases. En este paso se comienza a implementar la ontología. A partir de los términos definidos en el paso anterior se realiza el análisis para determinar cuáles se modelan en la ontología como clases. Como resultado se definieron 30 clases en la ontología. Las clases “Persona” y “Cadena_Transmisión” son dos de las fundamentales en el modelo ontológico. A su vez, cada una de estas clases conlleva otras con el objetivo de caracterizar a los individuos que la componen y así ofrecer análisis de interés. En la figura 1 se muestra la jerarquía de las clases “persona” y “Cadena_Transmisión”. Se definieron las clasificaciones por las que puede transcurrir una persona que ha sido diagnosticada con la enfermedad: Persona_Fallecida, Persona_Recuperada y Paciente_Activo. A su vez, un paciente activo puede ser Paciente_Estable o Paciente_Grave. Además, el que está en estado de gravedad significativo, con peligro para su vida, se le cataloga como Paciente_Crítico. Adicionalmente, se incluyó la clase Paciente_Asintomático por el alto número de pacientes con este comportamiento y precisamente por ser una de las características que influyen considerablemente en la alta transmisibilidad del virus. Resulta interesante la jerarquía de la clase. Por lo tanto, un razonador puede inferir de forma automatizada los individuos que pertenecen a estas clases, lo que constituye un aspecto clave en las tareas de clasificación. Aprovechando esas potencialidades, se definieron varias clases definidas para clasificar las cadenas de contagio. La riqueza expresiva de OWL 2 permitió definir las condiciones necesarias y suficientes para varios tipos de cadenas de transmisión.
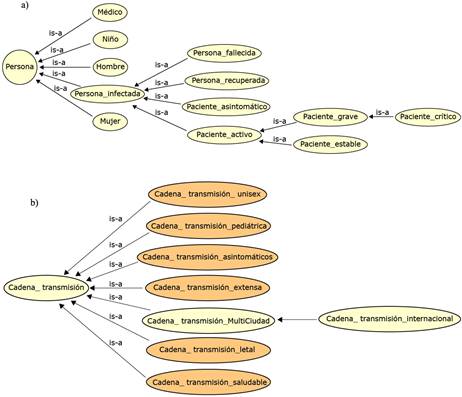
Fig. 1 Clases y jerarquía de clases: a) Jerarquía de la clase “persona”. b) Jerarquía de la clase Cadena_Transmisión.
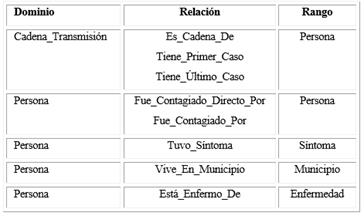
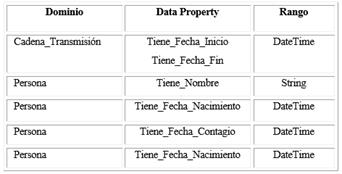
Paso 5: Definir las propiedades. Las propiedades de una ontología son de dos tipos: propiedad de objeto (object property) o de dato (data property).18 Las propiedades de objeto establecen a los objetos de una clase de atributos cuyos valores sean objetos. Por lo tanto, este tipo de propiedades permite relacionar individuos. Para restringir las clases que pueden intervenir en una propiedad, en OWL se define el dominio y el rango. En el cuadro 1 se muestran las propiedades que tienen las clases “Cadena_Transmisión” y “Persona” como dominio. Por ejemplo, un individuo de la clase “Cadena_Transmisión” “Es_Cadena_De individuos” de la clase “Persona”. En este caso, la propiedad “Es_Cadena_De” tiene como dominio y rango a las clases “Cadena” y “Persona”, respectivamente. Solo se incluyen las propiedades de estas clases, pero la ontología contiene 25 object properties en total. Además, como las object properties permiten establecer relaciones binarias entre individuos, cada propiedad posee una propiedad inversa. Por ejemplo, la propiedad “Fue_Contagiado_Directo_Por” posee la propiedad inversa “Contagio_Directo_A”, que permite establecer que si “A Contagio_Directo_A B”, entonces “B Fue_Contagiado_Directo_Por A”.
Se adicionaron varias data properties que permiten caracterizar los individuos de las clases. En el cuadro 2 se muestran las de las clases “Persona” y “Cadena_Transmisión”.
Paso 6: Definir facetas y/o restricciones sobre los slots o relaciones. En este paso se definió un conjunto de restricciones existenciales, universales y de cardinalidad. A continuación se describen las restricciones establecidas para las clases definidas que se incluyen en la ontología (Fig. 2). Como condición necesaria y suficiente para la clase “Cadena_Transmisión_Unisex” se definió que solo podía tener personas del mismo sexo. En este caso se definió una restricción universal, caracterizada en OWL por la expresión only. Para la clase “Cadena_Transmisión_Pediátrica” se definió que en la cadena debía haber al menos un niño. Se modeló a partir de una restricción existencial, identificada en OWL por la expresión some. En el caso de la clase “Cadena_Transmisión_Asintomáticos” se definió que todos los miembros de la cadena debían ser asintomáticos. Para esto, se definió una restricción universal. Para la clase “Cadena_Transmisión_Extensa” se creó una restricción de cardinalidad que define que deben tener un mínimo 5 de personas. Este umbral se estableció como ejemplo; en un entorno real sería mayor. Se considera una “Cadena_Transmisión_Letal” si entre sus miembros hay algún fallecido. Por lo tanto, se definió como una restricción existencial. Adicionalmente, se definió que en una “Cadena_Transmisión_Saludable” todos sus miembros son personas recuperadas. Por la tanto, también se definió mediante una relación universal.
Para poder identificar los individuos que pertenecen a la clase “Cadena_Transmisión_MultiCiudad” se aprovecharon las potencialidades del Semantic Web Rule Language (SWRL), que permite especificar relaciones más complejas y complementa a OWL con vista a mejorar la expresividad. En este caso, se definió que en una “Cadena_Transmisión_MultiCiudad” existirían miembros que pertenecen a diferentes ciudades.
Finalmente, para determinar el tiempo de actividad de una cadena se definió que su fecha de inicio sería la fecha de contagio del primer caso y la fecha fin sería la fecha de contagio del último caso. Esta regla también se modeló utilizando SWRL.
Paso 7: Definir instancias. En este paso se definen individuos para las clases especificadas y se establecen los axiomas correspondientes.
Caso de estudio
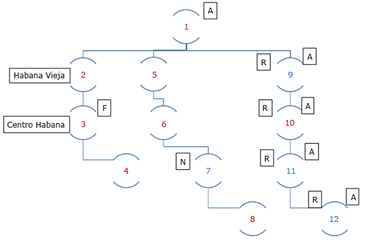
Para ilustrar la utilidad de la ontología desarrollada y demostrar su aplicabilidad, a continuación, se presenta un caso de estudio. Se modelarán las cadenas de contagio mostradas en la figura 3. El Paciente_1 contagió a los pacientes 2, 5 y 9. En la figura, el color del número de los pacientes muestra su sexo. Como se puede observar, existen tres cadenas: 1, 2, 3, 4; 1, 5, 6, 7, 8; y 1, 9, 10, 11, 12. Adicionalmente, se indica cierta información de los pacientes; se ilustrará cómo se describe utilizando la ontología desarrollada y cómo se explota la capacidad de razonamiento de este modelo ontológico. En la figura 4 se muestran diferentes vistas de Protégé que indican cómo se representó la información en la ontología.

Fig. 4 Inferencias sobre las cadenas de transmisión. a) Pacientes que pertenecen a la clase “Hombre”. b) Muestra que “Paciente_3” pertenece a la clase “Persona_Fallecida”. c) Municipio de residencia de “Paciente_2”. d) pacientes que pertenecen a la clase “Paciente_Asintomático”. e) Municipio de residencia de “Paciente_3”.
Además de la información individual de los pacientes, se definieron mecanismos para identificar las cadenas de contagio. A partir de la propiedad “Contagio_Directo_A” se especificó que el Paciente_1 contagió a los pacientes 2, 5 y 9. Sucesivamente se estableció la transmisión directa del resto de los pacientes. De esta manera, el razonador infirió que la Cadena_1 contenía a los pacientes 1, 2, 3 y 4 y respondió a la PC1. En la figura 5 se muestran las inferencias sobre las cadenas de transmisión. Para alcanzar la inferencia del razonador, se definió la propiedad “Contagio_A” como transitiva, a partir de la riqueza expresiva de OWL. Además, como subtipo de esta propiedad, se definió el “Contagio_Directo_A”. De esta manera, se logró la trazabilidad de todas las personas que de manera indirecta contagiaron a un paciente determinado y correspondió al “Paciente_8”. Igualmente, se infirieron los miembros de la “Cadena_2” y de la “Cadena_3”. Dado que todos los miembros de la “Cadena_1” eran hombres, se debió clasificar como “Cadena_Transmisión_Unisex”. Luego de modelarlo en la ontología, se infirió que la “Cadena_1” se clasificaba como “Cadena_Transmisión_Unisex”, lo que respondió a la PC4. Al definir que el “Paciente_3” pertenecía a la clase “Persona_Fallecida”, el razonador clasificó a la “Cadena_1” como “Cadena_transmisión_Letal”, y dio respuesta a la PC3. También se definió que los pacientes 2 y 3 vivían en los municipios “Habana_Vieja” y “Centro_Habana”, respectivamente. Por lo tanto, la “Cadena_1” también se clasificó como “Cadena_transmisión_MultiCiudad” y respondió a la PC5. Los pacientes menores de edad debían incluirse en la clase Ninno. De esta manera, las cadenas que incluyeron a miembros de esta clase fueron clasificadas automáticamente como “Cadena_transmisión_Pediátrica”. En este caso, se definió que el “Paciente_7” era un niño. A partir de esta definición, la cadena que incluyera al “Paciente_7” debía clasificarse como “Cadena_transmisión_Pediátrica”. La “Cadena_2” fue clasificada como “Cadena_transmisión_Pediátrica” porque incluyó al “Paciente_7”, lo que respondió la PC6. Una cadena de transmisión extensa posee al menos 5 pacientes. Por lo tanto, como “Cadena_2” poseía 5 miembros y también fue clasificada como “Cadena_transmisión_Extensa”, lo que respondió la PC7. Se definió que la cadena en la que todos sus miembros fueran asintomáticos se clasificaría como “Cadena_Transmisión_Asintomáticos”. De esta manera, se definió que los pacientes 1, 9, 10 y 11 pertenecían a la clase “Paciente_Asintomático”. Además, como todos correspondían a “Paciente_Recuperado”, la “Cadena_3” fue clasificada como “Cadena_Transmisión_Saludable” y “Cadena_Transmisión_Asintomáticos”, lo que respondió las PC8 y PC9.
Finalmente, se definieron mecanismos en la ontología para determinar la fecha de inicio y de fin de las cadenas a partir de la fecha establecida del primero y del último caso.
Durante la investigación se identificó un grupo de cadenas con un comportamiento interesante. La detección de estas cadenas de contagio permitiría realizar un análisis más profundo y así detectar las posibles causas que provocan ese comportamiento. Además, las cadenas en las que se combina un grupo de patrones podrían ser las que más atención debieran recibir. Por ejemplo, una cadena que sea de pacientes de un solo sexo y donde todos sean asintomáticos ameritaría ser estudiada en profundidad. En este estudio se ha identificado un grupo de cadenas que pudieran ser interesantes; sin embargo la contribución no está en estas cadenas, sino en la explotación de los beneficios de OWL para establecer los mecanismos para caracterizar las cadenas y constatar su capacidad para el análisis. Por lo tanto, esta ontología se pudiera enriquecer con la definición de otros tipos de cadenas. Por ejemplo, resultaría de interés verificar el comportamiento de las mujeres embarazadas que contraen la enfermedad o las que recientemente tuvieron un hijo.
Los resultados del análisis automatizado de las cadenas de contagios podrían contribuir al trabajo epidemiológico o a trazar nuevas medidas por los gobiernos, considerando el comportamiento social. Por ejemplo, si al analizar la fuente de contagio de los niños menores de un año que se han enfermado con la COVID-19 se observa que en la mayoría de los casos no se han contagiado a través de su madre, demostraría que no se está prestando la suficiente atención a este grupo etario. Por lo tanto, se podrían realizar campañas comunicacionales para reforzar la conciencia sobre el cuidado de los niños o de proveer buenas prácticas a las madres para que mantengan a sus hijos fuera de peligro.
Igualmente, a partir de este enfoque se pudiera buscar un índice que considere los integrantes de una cadena y el tiempo transcurrido entre el primer y el último caso. Así, una cadena que tenga un índice elevado podría indicar que no se están siguiendo las medidas adecuadas para la contención o que por algún motivo el virus se está expandiendo rápidamente. Adicionalmente, es posible incorporar indicadores de utilidad para el trabajo epidemiológico; por ejemplo, el índice reproductivo básico de cada paciente, así como el promedio de una cadena. Esos análisis podrían aportar insumos al trabajo epidemiológico y evaluar las medidas que se aplican.
Aunque el trabajo se ha enfocado principalmente en ilustrar las potencialidades del modelo ontológico para encontrar información de interés para los análisis de las cadenas de contagio, también se han definido mecanismos para hacer análisis en pacientes individualmente. Por ejemplo, se definió la clase Enfermedad. De esta manera, se puede verificar para cada paciente qué enfermedades previas tenía. También se podría correlacionar el comportamiento de un paciente con ciertas enfermedades. Puede ser que exista una enfermedad en la que todas las personas que la padecieron terminaron falleciendo. Ese hallazgo despertaría la atención sobre esa enfermedad y el estudio de las causas de ese comportamiento. Un análisis similar puede hacerse para los síntomas. Una de las líneas planteadas para continuar la investigación es integrar la ontología desarrollada con otras que describen aspectos específicos, como la Infectious Disease Ontology (IDO)19 y otras.20,21,22,23) Igualmente, se ampliaría la ontología para representar las secuelas que manifiestan los recuperados de la COVID-19.
Por otra parte, aunque fueron definidos los mecanismos para obtener información relevante, este modelo ontológico puede ser consultado por diferentes agentes de software que soporten OWL. Además, se puede explotar el modelo para realizar consultas a partir de diferentes lenguajes, tal como SPARQL.24 La ontología desarrollada puede consultarse en: https://drive.google.com/file/d/1QpVziB0Z4Ki-kRPy8g5_heb1Mgx8i_54/view?usp=sharing
Conclusiones
En este trabajo se ha descrito una ontología desarrollada con el propósito de representar y analizar información sobre cadenas de contagios de COVID-19. Se ha ilustrado cómo se explota la riqueza expresiva de OWL para representar diferentes aspectos de las cadenas de contagios. A partir de este modelo ontológico se puedan realizar análisis automatizados de la información representada y arribar a inferencias de interés para el seguimiento de la enfermedad. La ontología se construyó siguiendo una metodología sólida que contribuyó a garantizar su calidad. La validación de la ontología corroboró que está correctamente diseñada, cumple con las especificaciones como sistema lógico-formal y satisface los requisitos para los que fue creada.
No es objetivo de este trabajo sustituir otros enfoques. El propósito fundamental es proveer a la comunidad de una alternativa y así contribuir al enfrentamiento a la COVID-19. Esta investigación se plantea continuar en dos líneas. Primeramente se trabajará en la integración con otras ontologías que comparten conceptos sobre epidemiología y enfermedades. Luego se planteará el desarrollo de un sistema de información basado en el modelo ontológico. Aunque la motivación de esta investigación está centrada en el estudio de la COVID-19, la ontología resultante puede ser utilizada para representar y analizar el comportamiento de otras enfermedades de transmisión entre humanos.