INTRODUCCIÓN
La disentería bacilar fue descrita en los papiros de Ebers 2000 años antes de nuestra era (a.n.e) por Hipócrates, Padre de la Medicina, en el siglo V a.n.e.1Shigella fue descubierta hace más de 100 años por el microbiólogo japonés Shiga. En la década de 1890 fue reconocida como agente etiológico de la disentería bacilar o shigelosis y fue adoptada como género en la década de 1950.2 Ya desde la antigüedad se describía el cuadro clínico de esta enfermedad, en la actualidad se reconoce a la shigelosis por la presencia de espasmos abdominales, diarrea, fiebre y heces sanguinolentas.1
El género Shigella pertenece a la familia Enterobacteriaceae, tribu Escherichia, con cuatro especies reconocidas: Shigella dysenteriae (S. dysenteriae), descrita por Shiga en 1896, Shigella flexneri (S. flexneri), descrita por Flexner en 1900, Shigella sonnei (S. sonnei), descrita por Sonne en 1915 y Shigella boydii (S. boydii), descrita por Boyd en 1938.2
Shigella dysenteriae (Grupo A), es considerado el más patógeno, presenta 15 serotipos, el serotipo 1 produce la toxina Shiga, involucrada en el síndrome urémico-hemolítico, que tiene una mortalidad cercana al 20%; Shigella flexneri (Grupo B), presenta ocho serotipos, algunos están involucrados con el síndrome de Reiter, un tipo de artritis reactiva ligada a factores genéticos y a la infección con algunos patógenos bacterianos; Shigella boydii (Grupo C), cuenta con 19 serotipos (inicialmente numerados del 1 al 20, pero el serotipo 13 fue reclasificado como Escherichia albertii) y Shigella sonnei (Grupo D), cuenta con un solo serotipo. Los grupos C y D causan un cuadro infeccioso generalmente auto limitado, con una baja mortalidad.3
Shigellas son bacilos gramnegativos anaerobios facultativos, pero se multiplican mejor en condiciones aeróbicas. Las colonias son convexas, circulares, transparentes y con bordes intactos que alcanzan un diámetro de casi dos milímetros en 24 horas.4 Basándose en estas características se realiza su determinación en los medios de cultivo para su posterior identificación mediante estudios fisiológicos.
Las infecciones por Shigella casi siempre están limitadas al tubo digestivo; la invasión de la circulación sanguínea es poco frecuente. Shigellas son muy transmisibles, la dosis infecciosa es del orden de 103 microorganismos (en tanto que por lo general es de 105 a 108 para Salmonellas y Vibrios). El proceso patológico esencial es la invasión de las células del epitelio de la mucosa (las células M) por la fagocitosis activada, el escape de la vacuola fagocítica, la multiplicación y la diseminación dentro del citoplasma de la célula epitelial y su paso a las células adyacentes. Los microabscesos de la pared del intestino grueso y la porción terminal del intestino desencadenan necrosis de la mucosa, ulceración superficial, hemorragia y formación de una pseudomembrana en la zona ulcerosa que consta de fibrina, leucocitos, residuos celulares, una mucosa necrótica y bacterias. A medida que cede el proceso el tejido de granulación llena las úlceras y se forma un tejido cicatrizal.4
La gravedad de la infección y la tasa de mortalidad dependen, por un lado, de factores relacionados al huésped como la edad (los niños menores de cinco años son los más afectados), el estado nutricional y el inmunológico de la persona afectada y el serogrupo y el serotipo de la bacteria.5
Shigellas son transmitidas por los alimentos, los dedos, las heces y las moscas de persona a persona. La shigelosis se ha convertido en un problema importante en las guarderías en los Estados Unidos (EEUU). S. dysenteriae puede propagarse ampliamente; se ha intentado quimioprofilaxis masiva por períodos limitados (en personal militar), pero las cepas resistentes de Shigellas tienden a surgir con rapidez.4
Los seres humanos son el único reservorio para Shigella. Se estima que cada año se producen en los EEUU casi 450 000 infecciones por Shigella; esta cifra palidece si se compara con los 150 millones de casos que ocurren cada año en todo el mundo. S. sonnei es responsable de casi un 85% de las infecciones en EEUU, pero en los países en desarrollo predomina S. flexneri. Se producen epidemias por S. dysenteriae, una especie especialmente virulenta, en África y América Central, y la mortalidad por caso es del cinco al 15%. La shigelosis es una enfermedad principalmente pediátrica y el 60% de las infecciones afectan a niños menores de 10 años.6
En Chile S. flexneri y S. sonnei constituyen más del 80% de las cepas de Shigella aisladas, con una proporción similar entre ambas; predomina S. sonnei cada dos o tres años.7
En Cuba, un país tropical, con una población de poco más de 11 millones de habitantes, los factores climáticos favorecen que la enfermedad diarreica aguda (EDA) sea una causa importante de atención médica en presencia de condiciones higiénicas sanitarias desfavorables. En el año 2015 fueron informadas más de un cuarto de millón de atenciones médicas por EDA, con 507 mil consultas, para una tasa de 34,4x1 000 habitantes.8,9,10
Un estudio realizado en el año 2017 en el Hospital Pediátrico Universitario “José Luis Miranda” de la Provincia de Villa Clara entre los años 2006 y 2015 mostró que los aislamientos por años de Shigella sin precisar especies (spp) y Salmonella spp ocuparon el primer y el segundo lugar en todos los años, excepto en 2013 y 2014, en los que Vibrio cholerae predominó.8
La resistencia antimicrobiana ha complicado el tratamiento de la shigelosis desde 1940, cuando se detectaron las primeras cepas de Shigella resistentes a sulfonamidas en Japón. Subsecuentemente, Shigella adquirió resistencia a la tetraciclina y el cloranfenicol durante la década de 1950, con lo que la ampicilina se convirtió en el fármaco de elección. A principios de 1980 aparecieron las cepas resistentes a la ampicilina y se diseminaron en Asia y África. Posteriormente, el cotrimoxazol se convirtió en el tratamiento de elección y fue efectivo hasta mediados de 1980. Hasta el año 2007 las fluoroquinolonas, los ß-lactámicos y el cotrimoxazol eran los antimicrobianos de elección para el tratamiento de la shigelosis; sin embargo, su utilización nuevamente se vio restringida debido al surgimiento de cepas resistentes.11,12
La creciente resistencia a los antimicrobianos limita el uso de tratamientos empíricos; en la última década se ha informado un incremento de cepas resistentes a la ampicilina, el trimetoprim-sulfametoxazol, el cloranfenicol y el ácido nalidíxico. La aparición de aislamientos de cepas multidrogorresistentes (MDR) -entiéndase resistencia a tres o más familias de antimicrobianos- de Shigella es una preocupación creciente en todo el mundo.5,7
Es importante para la Provincia de Villa Clara tener identificadas las cepas de Shigella que circulan y los patrones de resistencia antimicrobiana, lo que permitirá el uso adecuado de los antibióticos en el accionar médico, así como la prevención de complicaciones asociadas a esta enfermedad. Se decidió realizar la presente investigación con el objetivo de caracterizar la resistencia antimicrobiana de cepas de Shigella aisladas en niños atendidos en el Hospital Pediátrico Universitario “José Luis Miranda” en el período de enero a diciembre de 2018.
MÉTODOS
Se realizó un estudio descriptivo transversal con el objetivo de caracterizar la resistencia antimicrobiana de cepas de Shigella aisladas en niños atendidos en el Hospital Pediátrico Universitario “José Luis Miranda” de la Ciudad de Santa Clara, Provincia de Villa Clara, en el período de enero a diciembre de 2018.
La muestra fue seleccionada por un muestreo no probabilístico intencionado y quedó conformada 25 cepas a las que se les realizó antibiograma por el método de difusión de Kirby Bauer para determinar la susceptibilidad a los siguientes antimicrobianos: ácido nalidíxico, ciprofloxacino, cloranfenicol, ampicillina, sulfametoxazol-trimetoprim, ceftriaxona y amikacina.
Las cepas de Shigella fueron aisladas e identificadas en el Laboratorio de Microbiología del Hospital Provincial Ginecoobstétrico Universitario “Mariana Grajales” de las mismas ciudad y provincia; se siguieron las normas y los procedimientos organizativos (PNO) establecidas en el laboratorio.
Se operacionalizaron las variables: especie de Shigella, antimicrobianos empleados, resistencia microbiana y patrones de resistencia microbiana.
El análisis estadístico de los resultados se realizó con el paquete SPSS® versión 15.0 (SPSS Inc., Chicago, IL, EEUU). Se emplearon métodos de estadística descriptiva para el procesamiento de la información mediante la determinación de frecuencias absolutas y relativas; los resultados se representaron en tablas y gráficos.
Se tuvieron presentes las normas éticas establecidas en el contexto y los principios bioéticos de autonomía, beneficencia, no maleficencia y justicia.
RESULTADOS
S. sonnei fue la especie más aislada, con 19 cepas, para un 76% del total de aislamientos, en segundo lugar se encontró S. flexneri (20%) y solo el 4% correspondió a S. boydii; no se aisló S. dysenteriae.
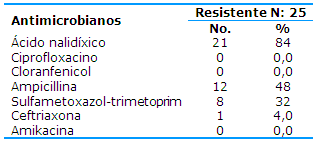
Shigella spp. mostró un 84% de resistencia al ácido nalidíxico y a la ampicillina un 48%; sin embargo, la totalidad de las cepas no mostraron resistencia al ciprofloxacino, al cloranfenicol y a la amikacina (Tabla 1).
Fuente: Registro de sección de coprocultivo
Shigella sonnei mostró una marcada resistencia al ácido nalidíxico, con 18 cepas resistentes, para un 72% del total de aislamientos, seguido de la ampicillina y el sulfametoxazol-trimetoprim, con el 40% y el 32%, respectivamente, mientras que ninguna cepa fue resistente al ciprofloxacino, al cloranfenicol y a la amikacina. Se encontró un por ciento de resistencia bajo (4%) para la ceftriaxona.
Se encontraron siete patrones de resistencia antimicrobiana, el patrón predominante fue el cinco, con resistencia solo al ácido nalidíxico, presente en el 32% de los casos; los patrones con resistencias a dos antimicrobianos (AN+AMP y AN+STX) fueron observados en el 20% (5) y el 16% (4). Se encontraron patrones de resistencia a tres antimicrobianos (AN+AMP+STX) en el 12% (3) y a cuatro antimicrobianos (AN+AMP+STX+CRO) en el 4% (1) -Tabla 2-.
AN: ácido nalidíxico; AMP: ampicilina; STX: sulfametoxazol-trimetoprim; CRO: ceftriaxona-cefotaxima
Fuente: Registro de sección de coprocultivo
DISCUSIÓN
La disentería bacilar causada por Shigella constituye una de las causas más comunes de EDA con sangre. Se asocia con frecuencia a malos hábitos higiénicos, con elevada incidencia en países subdesarrollados o en vías de desarrollo como Cuba. Esta infección tiene como consecuencias, en la edad pediátrica, un grupo de complicaciones entre las que se encuentran la deshidratación, los desequilibrios ácido básicos, el choque tóxico e incluso la muerte.13,14
La shigelosis posee gran relevancia por ser una enfermedad que precisa de tratamiento antimicrobiano, con una elevada transmisión por su pequeña dosis infecciosa y por las posibles complicaciones que puede desencadenar, es por esto que en las Salas de Pediatría se tiene un manejo protocolizado que permite actuar con premura ante la sospecha de infección por Shigella. En el Hospital Pediátrico Universitario “José Luis Miranda” se toma como premisa en estos casos, después del control de las complicaciones, el uso de antimicrobianos. El Laboratorio de Microbiología juega un papel esencial en el diagnóstico específico del serogrupo de Shigella, lo que es útil en el momento de emitir un pronóstico y evaluar conductas a seguir. Es importante esclarecer que de rutina no se realizan a todos los pacientes pruebas de susceptibilidad antimicrobiana, de ahí que estudios como este en el que se determina cuáles son los serogrupos más aislados y además se determinan los patrones fenotípicos de resistencia constituyen una herramienta valiosa para el Especialista en Pediatría porque permite que los tratamientos se sustenten en un fundamento de base científica.
S. sonnei fue la especie más frecuente en la presente investigación, seguida de S. flexneri. Baca Carlos y colaboradores, en Perú, informan supremacía de S. flexneri y, al igual que en esta investigación, no notifican aislamientos de Shigella dysenteriae.3
Marcoleta y colaboradores, en la Región Metropolitana de Chile, informa supremacía de Shigella sonnei.7 Della Gaspera y colaboradores informan sobre un brote de Shigella sonnei en julio de 2012 en la Cuidad de Lujan, Buenos Aires.15
Los aislamientos de Shigella, a nivel mundial, difieren en el serogrupo según su distribución social; se encuentran con más frecuencia Shigella dysenteriae en países subdesarrollados en los que predominan el hacinamiento y las malas condiciones socio económicas e higiénico sanitarias. En los países desarrollados se han aislado con menor frecuencia como causas de EDA y Shigella sonnei es la que prevalece, relacionada con cuadros más larvados y que llevan, con menor medida, a la aparición de las complicaciones de la enfermedad.
La resistencia de Shigella a los antimicrobianos está en incremento debido a la aparición de mecanismos de resistencia ante antimicrobianos como el trimetoprim-sulfametoxazol, el ciprofloxacino y la ampicilina. Para realizar una interpretación correcta del antibiograma se debe tener en cuenta la resistencia intrínseca del microorganismo en cuestión; en el caso de Shigella no se ha demostrado resistencia intrínseca a los beta lactámicos, pero se debe tener la precaución en el uso de aminoglucósidos, cefalosporinas de primera y segunda generación y cefamicinas, que pueden presentar susceptibilidad in vitro y no tener efectividad clínica, por lo que no deben ser notificados como susceptibles. Se han observado brotes de infección producida por cepas resistentes a azitromicina y a cefalosporinas de tercera generación.16
Baca Carlos y colaboradores, en Perú, no encontraron resistencia de Shigella al ciprofloxacino, al ácido nalidíxico y al aztreonam. El presente estudio discrepa con por cientos de resistencias para ácido nalidíxico que fueron altos (84%), pero coincide en el caso de ciprofloxacino.3
Manera, en Córdova, no se encontró resistencia a nitrofuranos, cefalosporinas de tercera generación, fosfomicina y ciprofloxacino.7
Della Gaspera, en el estudio de un brote de Shigella sonnei en julio de 2012 en la Cuidad de Lujan, Buenos Aires, encontró un único perfil de susceptibilidad, todos resultaron ser sensibles a los siguientes antibióticos: ampicilina, cloranfenicol, ciprofloxacino, furazolidona y cefotaxima; asimismo, mostraron resistencia al trimetoprim-sulfametoxazol.15
La Organización Mundial de Salud (OMS) estima que aproximadamente el 50% de los antibióticos son administrados innecesariamente, lo que contribuye al aumento de la resistencia bacteriana que, a su vez, incrementa la mortalidad por enfermedades infecciosas y genera un grave problema de salud pública. El uso inadecuado de los antibióticos representa un riesgo para la salud y un despilfarro de recursos económicos en los servicios de salud.17
La proliferación de cepas resistentes como consecuencia del uso de antibióticos es un proceso natural que se ha acelerado por la aplicación inadecuada de ciertos fármacos y, pese a ser un problema inevitable, diversas organizaciones como la OMS y los Centros para el Control y Prevención de Enfermedades (CDC), a través de los años, se han propuesto implementar numerosas estrategias para prevenir y solucionar, o al menos tratar de retrasar, este proceso.17
Según criterio del autor una de las formas de prevención de la resistencia o la multiresistencia es contar con los patrones fenotípicos de resistencia de las cepas circulantes en la localidad en la que se trabaja porque permite la correcta implementación de políticas de uso de antimicrobianos.
Si bien el patrón de resistencia predominante en esta investigación fue el resistente a un solo antimicrobiano (ácido nalidíxico), hay que resaltar que se encontraron por cientos no despreciables de cepas multirresistentes con patrones fenotípicos de resistencias a tres antimicrobianos (AN+AMP+STX) en un 12% y a cuatro antimicrobianos (AN+AMP+STX+CRO) en 4%. Estos resultados ofrecen evidencias que justifican mantener la vigilancia de la resistencia de Shigella en la provincia y en el país pues algunos de los antimicrobianos involucrados están considerados de primera línea en el tratamiento de las shigelosis como el trimetoprim-sulfametoxazol y el ampicillin, además de cefalosporinas de tercera generación.
Marcoleta, en Chile, informa un patrón de multiresistencia en un 65,7%, superior al encontrado en este trabajo, solo el 17% presentó resistencia a uno o dos antibacterianos.7
La prevención de la aparición de cepas de Shigella multidrogorresistentes constituye una de las prioridades de la OMS porque si prevalecieran ante los casos diagnosticados fuera mucho mayor el riesgo de transmisibilidad y de mortalidad por esta enfermedad infecciosa. Corresponde a los actores clínicos y microbiológicos socavar, con inteligencia, esta epidemia que se avecina ante el tema de la resistencia antimicrobiana.
CONCLUSIONES
El serogrupo más representativo en la muestra fue el Grupo D (Shigella sonnei) y no hubo aislamientos de Shigella dysenteriae. Se observó una elevada resistencia al ácido nalidíxico y una sensibilidad al ciprofloxacino en la totalidad de las cepas de estudio. Se constató multidrogorresistencia en un pequeño por ciento no despreciable de la muestra, lo que alerta sobre la necesidad de mantener una activa vigilancia en este problema de salud.