INTRODUCCIÓN
En su sentido moderno, la biología molecular pretende explicar los fenómenos de la vida a partir de sus propiedades macromoleculares.1 Con la aparición de los primeros casos diagnosticados con el virus SARS-CoV-2,2 en Cuba se implementaron un conjunto de medidas, para evitar el contagio y la propagación del virus, no obstante, el número de casos diarios con la COVID-19 comenzó a aumentar de forma paulatina, lo cual trajo consigo el incremento de la cantidad de ingresos diarios en los hospitales. Llegaron a superar los miles de casos, por lo que el personal de la salud, a pesar del esfuerzo realizado, tuvo que doblar voluntades en cuanto a la aplicación del protocolo establecido para el tratamiento de la enfermedad y el procesamiento de las muestras diarias.
En algunos casos se implementaron mecanismos automatizados, que fueron desarrollados para el control en el acceso a la información de forma oportuna, el fraccionamiento, mejora en el control, trazabilidad y uso de esta, además de la sustitución de procesos manuales. Durante los primeros periodos de la pandemia fueron utilizados por los hospitales, en el agrupamiento de los resultados de las pruebas de reacción en cadena de la polimerasa (PCR por sus siglas en inglés)3 de pacientes. Con el perfeccionamiento de los protocolos, los formatos de los documentos empleados sufrieron cambios, dificultando su empleo. Se retrasó el trabajo de los especialistas en la agrupación y centralización de la información, lo cual conllevo a que nuevamente se comenzaran a registrar los resultados en documentos Excel, con la dificultad y complejidad en los procesos de agrupación y la confusión, debido al gran volumen de información procesada y en algunos casos, errores humanos. Todo ello se reflejó en la recepción de las bases de datos de los centros de aislamiento, el montaje de las muestras en el equipo de PCR y al brindar información a la población sobre el estado de un resultado del estudio realizado.
Para enfrentar dicha problemática, se elaboró un sistema que automatiza la gestión, análisis de las muestras en los laboratorios, el proceso de planificación de pruebas en los pacientes ingresados y el control de los materiales empleados en dichos exámenes. Eliminando los procedimientos manuales, logrando el ahorro de recursos y la no introducción de errores humanos.
El objetivo de este trabajo es presentar un sistema que automatiza los procesos de análisis, procesamiento y resultado final de las muestras de PCR, además del control de los suministros médicos en los laboratorios y centros de toma de muestras.
MÉTODOS
Para la implementación del sistema se utilizaron los siguientes sistemas de desarrollo de programas, con herramientas de código abierto:
Lenguaje de programación php:4,5 de código abierto y adecuado para el desarrollo web; puede ser incrustado en HTML.6
Marco de trabajo ExtJs:7 desarrollo de aplicaciones web interactivas con AJAX,8 DHTML9 y DOM,10 utilizado para la conformación de las vistas.
Gestor de bases de datos Postgresql:11 las consultas relacionales se basan en SQL, mientras que las no relacionales con JSON.12 Además, se definieron procedimientos en otros lenguajes: Pearl13 o Python.14,15
Servidor de correo Zimbra:18 utilizado para las notificaciones y resúmenes estadísticos que se obtienen por el sistema automatizado.
Se realizó la integración con los dispositivos médicos utilizados para el análisis de las muestras:
RESULTADOS
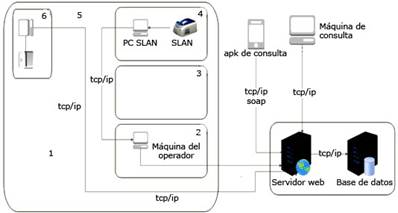
En el siguiente esquema se ilustra la interrelación de los equipos y componentes que conforman el sistema automatizado (Fig. 1):
En la figura 1, las funciones que realiza cada componente son las siguientes:
Máquina SLAN: encargada, a través del software de interacción con el SLAN, de obtener los datos de este.
SLAN: equipo médico encargado del análisis y procesamiento de las muestras.
Máquina del operador: encargada de gestionar y controlar todas las operaciones que se realizan en el sistema.
Aplicación (apk) para consulta: para conocer el estado de los resultados de los pacientes ingresados.
Máquina de consulta: utilizada para obtener información necesaria, para el trabajo con los resultados de los pacientes.
Servidor web: encargado de recibir, enviar y procesar las peticiones hechas por el operador, la apk y la computadora (PC) de consulta hacia el servidor de bases de datos.
Servidor de base de datos: encargado del almacenamiento de la información que se tramita desde el servidor web.
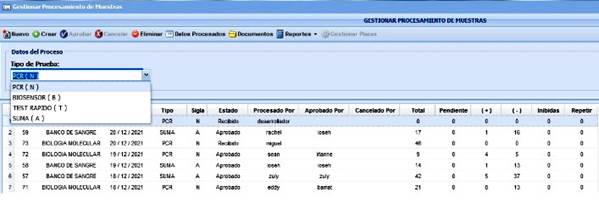
A continuación, se detalla el flujo del sistema automatizado para el procesamiento de las muestras: el sistema comienza con el establecimiento en un fichero de la información del paciente, la cual es obtenida de los ficheros que llegan desde los centros de aislamiento. El operador incorpora al documento el día de trabajo y selecciona que tipo de análisis realizar, (PCR, SUMA, Biosensor o test rápido) (Fig. 2).
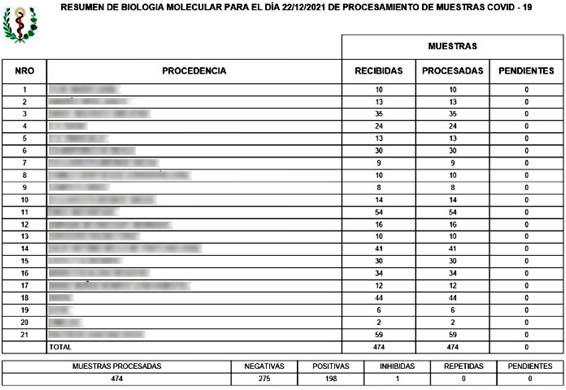
La base de datos cuenta con un número consecutivo, la fecha, el año, por quién es creado y el jefe que aprueba; además, muestra un resumen del total de muestras y cuántas de ellas fueron positivas, negativas, inhibidas y a repetir.
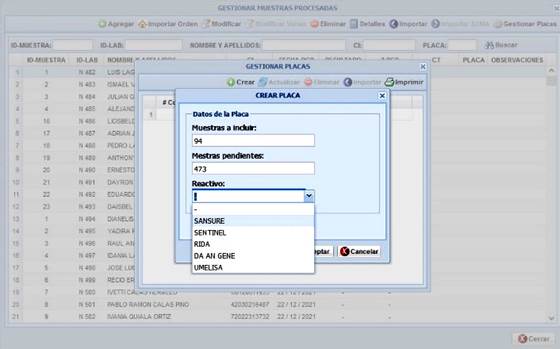
El documento creado contiene la información recibida y se formaliza en el fichero del día. El sistema de forma automática procesa la información y asigna el número de muestra correspondiente, para el tipo de análisis a realizar. En caso de que sea un paciente que ya existe, no se incluye y se registra con el mismo número. Si es nuevo, se incorpora con todos los datos. Posteriormente se crea el protocolo con el reactivo y el número de muestras a incluir. El sistema incluye en el protocolo los casos pendientes, de acuerdo con el número de muestras deseado y asigna el número de corrida correspondiente (Fig. 3).
En los casos que existan muestras con un primer resultado a repetir, por ser inhibidas, se incluirán de forma automática al final del grupo que no contiene primer resultado. Brinda la posibilidad de intercambiar a un paciente en cualquier posición. Al concluir, se imprime el protocolo y se montan las muestras en el equipo de PCR, como indica el documento generado (Fig. 4).
Terminada la corrida se exportan los resultados, en un documento en formato Excel, en dependencia del reactivo que se emplee. Se copian en una carpeta compartida entre el puesto de trabajo (PC) del termociclador y la máquina de procesamiento con el GestLab. Este procedimiento permite importar sobre el protocolo en cuestión, el fichero Excel, e incorpora de manera automática los resultados a todos los pacientes que se encuentran en la corrida analizada.
Una vez concluido el día de trabajo, el jefe de grupo procede a realizar la aprobación, aspecto que permite generar el listado con el formato establecido por el Ministerio de Salud Pública, además del listado que se incorpora al sistema de gestión documental y un cierre consolidado con los totales en cuanto a cantidad de muestras por tipo de resultado y los centros de aislamientos que envían muestras (Fig. 5).
El puesto de mando tiene acceso para ver toda la información procesada por el laboratorio de biología molecular (LBM) y el banco de sangre, de forma resumida y la posibilidad de buscar resultados de una o varias personas, aspecto que elimina la necesidad de tener ficheros de varios días abiertos para buscar resultados.
Se incorporó una aplicación móvil, a través de la cual se publica la información de los pacientes dentro del hospital (Fig. 6).
La apk permite conocer los resultados, para la movilidad dentro del hospital. Esta aplicación se encuentra disponible en las salas, para que los médicos puedan disponer del resultado de cada uno de sus pacientes; garantizando que la información que se procesa en ambos laboratorios se actualice de forma automática en la entidad superior de salud, con información inmediata.
Control de los inventarios
Para el trabajo en el proceso de análisis de las muestras se emplean insumos médicos, estos son controlados e inventariados. En el sistema se incorpora un módulo que gestiona los principales procesos de registro y control material: recepción, ajuste, consumo, despacho y submayor de inventario continuo por productos, con cálculo de cobertura por días. Este módulo brinda información de manera inmediata al personal de abastecimiento médico. Notificando de forma diaria y oportuna sobre el gasto de material durante el procesamiento de muestras.
Gestión de los ingresos
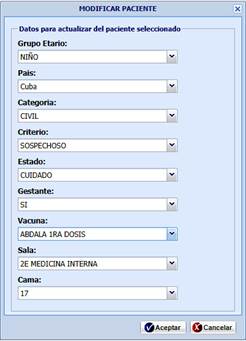
Al tener el resultado del PCR, si es positivo, se realiza una conciliación mediante el empleo de un servicio web, que brinda el sistema de ingresos del hospital y el GestLab; asigna de forma automática la sala y cama donde será ingresado el paciente. Da posibilidad de registrar otros datos adicionales como (Fig. 7). Esto favorece la generación de reportes estadísticos que se envían al Ministerio de Salud Pública, para el control de los pacientes ingresados.
Para validar el funcionamiento de la solución se aplicó una prueba funcional, mediante el método de caja negra.21 En la tabla 1 se muestra una comparación en base al tiempo de ejecución para el proceso de confección del protocolo.
DISCUSIÓN
Una vez puesto en explotación, en el centro de recepción y análisis de muestra en el Hospital Militar Central “Dr. Luis Díaz Soto” y transcurridos 8 meses de uso, se identifican los siguientes beneficios:
Reducción del tiempo en la obtención de los resultados por cada corrida en el laboratorio.
Continuidad al proceso de migración a software libre.
Incidió en la transformación del proceso organizativo del laboratorio, definiendo roles con responsabilidades específicas en cada área.
Reducción del error humano en la incorporación de los resultados, gana notablemente en confiabilidad y seguridad de los datos que se procesan.
Mejora los procesos de información estadística sobre los resultados de las muestras y los pacientes ingresados.
Disminuye el tiempo de respuesta para notificar el resultado de los pacientes.
Garantiza que la información sea actualizada al sistema de gestión documental sin problemas (nube Papyrus),22 aspecto que beneficia a la población que es atendida por el hospital, con el objetivo de que conozca su resultado por vía telefónica.
Incide sobre el registro y control de los medios materiales que se destinan para garantizar el procesamiento de las muestras en cada turno de trabajo.
Incorporación de una aplicación móvil, para que el personal que labora en las salas de ingresados, conozca el resultado de las pruebas realizadas para sus pacientes y permite al personal de dirección, conocer los datos estadísticos del procesamiento diario de los laboratorios.
Se implementó un sistema automatizado que elimina los procesos manuales de análisis, procesamiento y resultado final de las muestras de PCR, control de los suministros médicos en los laboratorios, centros de toma de muestras y logra mayor rapidez en la respuesta a los pacientes.