My SciELO
Services on Demand
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Obstetricia y Ginecología
Print version ISSN 0138-600X
Rev Cubana Obstet Ginecol vol.38 no.1 Ciudad de la Habana Jan.-Mar. 2012
OBSTETRICIA
Diagnóstico prenatal citogenético mediante la hibridación in situ con fluorescencia
Cytogenetic prenatal diagnosis by means of in situ hybridization with fluorescence
Dr. Cs. Luis Alberto Méndez Rosado,I MSc. Alfredo Nodarse Rodríguez,II MSc. Enny Morales Rodríguez,I MSc. Anduriña Barrios Martínez,I MSc. Michel Soriano Torres,I Lic. Arlay Castelvi LópezI
I Laboratorio de citogenética del Centro Nacional Genética Médica. La Habana, Cuba.
II Departamento Provincial de Genética. Hospital Ginecobstétrico "Ramón González Coro". La Habana, Cuba.
RESUMEN
La utilización de la técnica de hibridación in situ con fluorescencia aplicada al diagnóstico prenatal citogenético es una vía rápida para establecer un nexo entre los genes y los cromosomas sin necesidad de realizar cultivos celulares, permitiendo la detección de anomalías cromosómicas en células en interfase. El objetivo del presente trabajo fue evaluar los resultados obtenidos en la introducción de este método para el diagnóstico prenatal de aneuploidías en embarazos de alto riesgo. Se examinaron 40 casos prenatales de alto riesgo por la técnica de hibridación in situ, se obtuvieron resultados satisfactorios en 34. Se corroboró con el resultado obtenido de la citogenética convencional en el 97 % de los casos. Se realizó el diagnóstico de aneuploidías de los cromosomas 18, 21 y 13, el 80 % de los núcleos examinados, presentó 3 señales. En los casos normales no existieron discrepancias con la citogenética respecto a los cromosomas sexuales, el número de cromosomas autosómicos (21,13 y 18) y los marcajes observados por FISH. Esta técnica debe ser aplicada en casos de alto riego de aneuploidías de los cromosomas 21, 13, 18 y X, casos con elevada ansiedad materna, y/o cuando una determinada situación clínica así lo demande. Esta técnica debe ser complementada mediante el examen cromosómico de las células fetales.
Palabras clave: hibridación in situ fluorescente (FISH), células en interfase, diagnóstico prenatal citogenético, líquido amniótico.
ABSTRACT
Fluorescence in situ hybridization applied to the cytogenetic prenatal diagnosis is a rapid way to stablish a nexus between genes and chromosomes without celular culture and allows detection of chromosomal abnormalities on interphase cells. The aim of the present study was to evaluate this method as a tool in prenatal diagnosis of aneuploidies in high risk pregnancies. Prenatal diagnosis was carried out in 40 high-risk pregnancies using fluorescence in situ hybridization, 34 had successuful results. The 97 % the cases were confirmed by conventional cytogenetic results. The diagnosis of 18, 21 and 13 chromosome aneuploidies showed three hybridization signals in 80 % of the scored nuclei. The results of fluorescence in situ hybridization were in conformity with the results of cytogenetic analysis in all the normal cases (sex and autosomic chromosomes). This technique should be applied in high risk cases of chromosomes aneuploidies (21,18, 13 and X), high maternal anxiety, or when significant clinical situation is present. It should be employed as an adjunctive tool to the examination of fetal chromosomes.
Key words: fluorescence in situ hybridization (FISH), interfase cells, cytogenetic prenatal diagnosis, amniotic fluid.
INTRODUCCIÓN
La citogenética convencional constituye una herramienta insustituible tanto en los estudios prenatales, posnatales como en investigaciones relacionadas con el cáncer. Actualmente en nuestro laboratorio el resultado del diagnóstico prenatal citogenético (DPC) tarda entre 15 y 20 días, es necesario el cultivo celular, la obtención de cromosomas y el diagnóstico al microscopio óptico. Con la introducción de la técnica de hibridación in situ con fluorescencia (FISH por sus siglas en inglés) aplicadas a células amnióticas en interfase, un resultado puede ser obtenido en tan solo 2 días, lo cual provee una rápida y segura identificación de las aneuploidías más frecuentes en el segundo trimestre del embarazo (21,18, 13 y X) y constituye una prueba diagnóstica complementaria a la de citogenética convencional. Además en ciertas situaciones clínicas el tiempo requerido para completar el análisis cromosómico podría constituir una significativa carga emocional para la pareja y un incremento del riesgo ante la posible interrupción del embarazo.1
La técnica FISH en células en interfase ha sido exitosamente utilizada en varios países con un alto grado de concordancia con los resultados de la citogenética clásica, pero no todas las anomalías cromosómicas pueden ser detectadas, particularmente los reordenamientos estructurales y los mosaicos en baja proporción constituyen una limitante de esta técnica durante el DPC.1-4
El presente trabajo tiene como objetivo evaluar los resultados obtenidos en la introducción de la técnica FISH, aplicada a células en interfase, en el DPC de un grupo de embarazos de alto riesgo.
MÉTODOS
Obtención de las muestras
Se extrajeron 20 mL de líquido amniótico entre las 16 y 20 sem de gestación mediante la amniocentesis, las muestras en el laboratorio se dividieron tomando 5 mL para la técnica FISH y 15 mL para el cultivo celular y obtención de cromosomas por los métodos convencionales según técnica descrita por Margaret y otros5 y modificada en el laboratorio.
Las muestras para la técnica FISH fueron obtenidas de:
- Muestras de líquido amniótico conservadas a 4 °C, una vez concluido el diagnóstico por citogenética convencional del caso.
- Muestras de líquido amniótico de casos en los que aún no se había obtenido el resultado por citogenética convencional.
Los casos de diagnóstico prenatal fueron remitidos al laboratorio por pertenecer a diferentes grupos de riesgo de cromosomopatías como: embarazadas mayores de 38 años, alteraciones detectadas por ecografía fetal que implicaban un riesgo de enfermedad de origen cromosómico, alteraciones en marcadores séricos maternos y casos con antecedentes familiares de enfermedades de origen cromosómico.
Preparación de la muestra
En la etapa de estandarización de la técnica, se utilizaron muestras de líquido amniótico que tenían menos de 30 días de almacenadas a 4 °C y que ya se había concluido el diagnóstico prenatal citogenético. Una vez estandarizada en el laboratorio se realizó la técnica de FISH sin conocimiento previo del diagnóstico cromosómico. Se escogieron muestras en las que el líquido fuera de color claro, para evitar contaminación con sangre materna.
Por cada muestra se tomaron de 3-5 mL de líquido amniótico que fue centrifugado a 1 000 rpm durante 10 min. El sobrenadante fue removido y se le añadieron al pellet 4 mL de EDTA-tripsina a 37 °C durante 20 min. Se le realizó la hipotonía (solución de KCl al 0,56 %) durante 20 min a 37 °C. Se realizaron dos fijaciones con metanol-ácido acético (3:1) se guardó a 4 °C durante 1 h. La extensión se realizó en láminas limpias precalentadas y fue verificada en microscopio de contraste de fase la calidad de la preparación.
Hibridación y detección
Para la identificación de los cromosomas 18, X y Y fueron utilizados kit de sondas Aneuvision para sondas a -satélite CEP 18 (p11.1-q11.1) marcada con fluorocromo SpectrumAqua, X (p11.1-q11.1) marcada con fluorocromo SpectrumGreen y Y (p11.1-q11.1) marcada con fluorocromo SpectrumOrange.
Para la identificación de los cromosomas 13 y 21 fueron utilizados kit de sondas Aneuvision para sondas LSI 13 (13q14) marcada con fluorocromo SpectrumGreen y 21 (q22.13-q22.2) marcada con fluorocromo SpectrumOrange.
Antes de la hibridación las láminas con las células del líquido amniótico fueron incubadas en una solución 2 XSSC (stándar saline citrato) a 37 °C durante media hora. La muestra fue deshidratada en una serie de alcoholes al 70 %, 85 % y 100 %. Por cada lámina se añadieron 10 mL de la mezcla de las sondas (18, X y Y o 13 y 21) se le colocaron cubreobjetos y se sellaron con cemento de goma. La lámina fue ubicada en un sistema de desnaturalización e hibridación del ADN (Alpha Unit Block Assembly for PTC DNA Engine Systems) y se realizó la codesnaturalización a 73 °C durante 3 min, la hibridación en cámara húmeda a 37 °C durante toda la noche. Al día siguiente se retiró el cubreobjeto y se realizaron dos lavados uno con 0,4 SSC/ 0,3 % NP40 a 72 °C durante dos minutos y otro con 2X SSC/ 0,1 % NP40 a temperatura ambiente durante 10 segundos.
En todos los casos se utilizó la tinción de contraste DAPI (diamidino fenil indol ) para la identificación de los núcleos celulares en interfase.
El análisis cuantitativo
Para la evaluación de las señales fluorescentes la observación se realizó con un lente de inmersión 100X. Los núcleos libres de citoplasma donde el marcaje era evidente fueron los seleccionados para el conteo de las señales. Se analizaron aquellos que tenían 1, 2, 3 y 4 señales dentro del núcleo, se descartaron los que aparecían sobrelapados, con una intensa luminosidad o un marcaje débil. Se consideró como una señal si el marcaje estaba dividido en 2, pero la distancia entre las mismas era menor al ancho de una de estas señales.
En el presente estudio para el conteo de las señales se asumió el criterio analítico de Ward y otros.1 Sobre el análisis del resultado de la técnica FISH las muestras fueron consideradas como informativas normales o anormales (no obstante el resultado final del estudio fue realizado sobre la base del diagnóstico del cariotipo). Las muestras fueron consideradas normales/disómicas cuando como mínimo el 90 % de los núcleos examinados mostraron 2 señales para cada cromosoma autosómico analizado (21,18 y 13) y un patrón que se correspondió con la hibridación de los cromosomas sexuales (XX o XY).
La muestra se consideró anormal cuando el 70 % o más de los núcleos analizados que hibridaron con las sondas de los cromosomas autosómicos (21,18 y 13) presentaban 3 señales o un patrón de señales para los cromosomas sexuales diferente al XX o XY.
El criterio para la definición del mosaicismo cromosómico aún no está bien definido internacionalmente pero consideramos que con un 20 % o más de células con un patrón de signos diferentes, existe un alto riesgo de mosaicismo cromosómico. Este criterio fue asumido de acuerdo a lo descrito en el prospecto del Kit Aneuvision para estas sondas y la experiencia de otros autores.6,7
Captura y procesamiento de imágenes
Se utilizó el sofware Cytovision versión 3.9, sección Genus de Applied Imaging para la captura, procesamiento y análisis de las imágenes. Las imágenes fueron capturadas desde microscopios con fluorescencia OLYMPUS BX51. De cada imagen tomada se guardó una versión cruda, como evidencia del marcaje real en los núcleos. Por cada caso se analizaron los núcleos, utilizando un set de filtros individuales para DAPI, Spectrum Aqua, Spectrum Green y Spectrum Gold.
Aspectos éticos
Las muestras de líquido amniótico se obtuvieron mediante el procedimiento de rutina del DPC establecido en el país que incluye el consentimiento informado previo a la pareja, o al menos a la gestante.
RESULTADOS
En el presente estudio se utilizaron dos vías para el DPC, la forma convencional a través del estudio cromosómico y la técnica FISH en amniocitos en interfase. En una primera etapa de estandarización se analizaron 26 muestras en que ya se conocía previamente el resultado cromosómico. Las restantes muestras se realizaron sin conocimiento previo de los estudios cromosómicos y estas fueron corroboradas mediante ellos.
Se analizaron 40 casos del DPC. En 34 casos se obtuvo una hibridación informativa para las sondas CEP (18, X y Y) y LSI (13 y 21). En los restantes 6 casos no siempre se logró la hibridación o esta fue débil. Pertenecen a la etapa de estandarización de la técnica 4 casos sin resultados.
En los 34 casos de marcaje con las 5 sondas, el número de células examinadas varió desde 10 hasta 70 con un promedio de 20 células analizadas por caso.
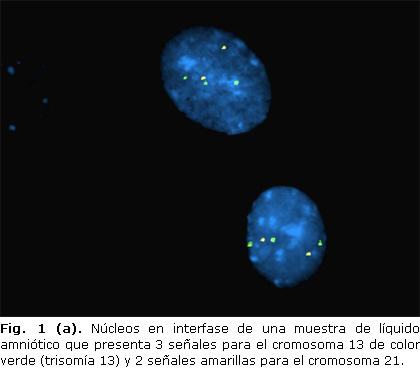
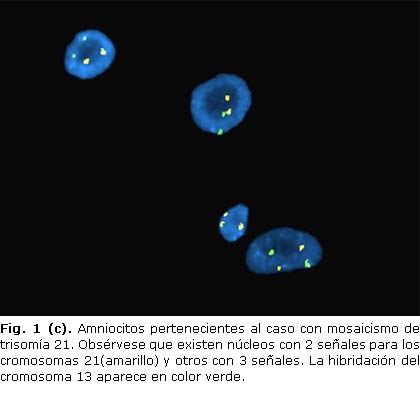
Se observaron 3 casos con aneuploidías cromosómicas, uno que correspondió a una trisomía 13, otro con una trisomía del par 18 y el tercero a un mosaico de trisomía 21 (Fig. 1 a, b y c). En cada caso se analizaron 15, 20 y 70 núcleos respectivamente, presentaron los dos primeros tres señales en el 80 % de los núcleos examinados.
En el tercer caso se presentó una metafase con trisomía 21, de 12 analizadas por citogenética convencional. Se realizó el FISH en estas láminas y de 70 núcleos examinados en 9 células aparecieron 3 señales correspondientes al cromosoma 21 para un 13 % de células con la posible línea trisómica. En los resultados de la resiembra de este caso se analizaron 100 metafases y se detectaron 3 con la trisomía 21 en diferentes frascos de cultivo.
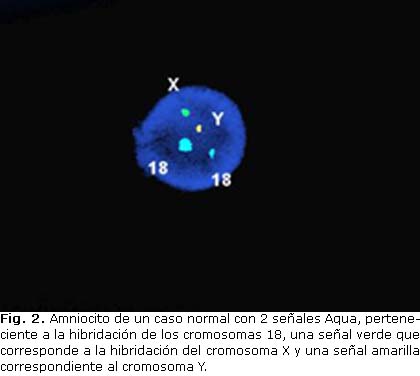
En los casos normales (Fig. 2) no existieron discrepancias respecto a los cromosomas sexuales y número de cromosomas autosómicos (21,13 y 18) observados por las señales de FISH y el resultado obtenido en el DPC por métodos convencionales. Existieron en menor proporción células que de acuerdo al número de señales observadas para cada sonda (21, 13, 18, X y Y) presentaron una señal o 3 señales. Los casos que por DPC convencional fueron normales tuvieron un predominio de dos señales (equivalente al par cromosómico específico que marcaba la sonda). Los núcleos con 1 señal y 3 señales fueron minoría en estos casos (no más del 6 % de las células analizadas).
Solo en un caso el resultado del FISH no pudo ser corroborado mediante la citogenética convencional. En una paciente de 40 años con un diagnóstico prenatal sin resultados y con 23 sem de embarazo, fue imposible extraerle sangre del cordón umbilical debido a la obesidad que mostraba. Se realizó una segunda amniocentesis y se analizaron 25 células por FISH. Se observaron 23 células con dos señales para los cromosomas 13, 21 y 18, una señal para el cromosoma X y una señal para el cromosoma Y. Una célula presentó tres señales para el cromosoma 13 y otra presentó dos señales para el cromosoma Y. El examen por ecografía de alta resolución no detectó malformaciones en el feto varón. Debido a la avanzada edad gestacional el líquido amniótico no es cultivado y no se obtienen resultados por citogenética convencional.
DISCUSIÓN
El FISH en células en interfase aplicado al DPC es una vía rápida para establecer un nexo entre los genes y los cromosomas sin necesidad de realizar cultivos celulares para examinar a estos últimos. El presente trabajo analiza resultados iniciales en la introducción de la técnica FISH al DPC en nuestro país.
La técnica FISH tiene una eficacia internacionalmente demostrada para el DPC de las más comunes aneuploidías del 2do. trimestre del embarazo. Sin embargo como toda técnica que se introduce por primera vez, la transición de la fase de estandarización a la fase de aplicación como un diagnóstico establecido requiere confiabilidad, reproducibilidad y seguridad en los resultados, la cual debe ser lograda con el diagnóstico de un significativo número de casos y su corroboración posterior mediante las técnicas citogenéticas convencionales. En este estudio de 34 casos con resultados se pudo corroborar el diagnóstico por FISH en 33 (97 %), no se obtuvieron resultados cromosómicos en 1 caso. No existió discordancia entre ambas pruebas ni para los casos normales, ni para casos con aneuploidías cromosómicas. Internacionalmente varios autores reportan un porcentaje de concordancia entre el 98-99 %.4,8,9
En la muestra que estudiamos los casos con disomías para los cromosomas 21,13 y 18 y sexualmente normales XX o XY, presentaron un rango de núcleos con 3 señales del 2-3 % e igualmente un rango de núcleos con una señal del 3 %, excepcionalmente fueron encontrados núcleos con 4 señales. Liehr y Ziegler en el análisis de 1 200 casos de FISH en células en interfase reportaron que alrededor del 7-10 % de los núcleos presentaban 3 señales en casos que eran completamente normales (disómicos).10 Reportes similares son realizados por diversos autores4-9 y es recomendable establecer en el laboratorio un punto de corte para definir el porcentaje de núcleos con 3 señales que se va a considerar normal.
Respecto a los casos que presentaron aneuplodías al menos el 80 % de los núcleos analizados presentaban 3 señales. Jobanputra y otros6 reportan en estos casos un 97 % de núcleos con 3 señales durante el análisis de 88 casos de DPC analizados mediante el FISH. Luquet y otros11 durante el análisis de 2 000 casos de FISH establecieron que la frecuencia de los núcleos con 3 señales fue del 85 %, 70 % y 86 % para las aneuploidías de los cromosomas 13, 18 y 21 respectivamente.
En el caso que presentó un 13 % (9/70) de los núcleos analizados con 3 señales del marcaje para el cromosoma 21, hubiese sido imposible descartar un mosaicismo cromosómico real por la técnica FISH. Diversos autores plantean que esta es una de las limitantes de esta técnica12-14 y otros consideran la sospecha de un mosaicismo real cuando la línea aneuploide está por encima del 20 %.6,7 En nuestro caso el mosaicismo pudo ser corroborado mediante la citogenética convencional al diagnosticarse 3 metafases con la trisomía 21(en diferentes frascos de cultivos) de 100 que fueron analizadas. No obstante este resultado obtenido por FISH unido al derivado del examen del primer cultivo (1 célula con trisomía 21 de 12 analizadas) constituyó una premisa para aumentar el conteo de metafases durante el estudio de la resiembra del líquido amniótico.
El caso imposible de corroborar por citogenética convencional debido al impedimento surgido para realizar la cordocentesis fue discutido con la pareja, que aceptó el resultado bajo la premisa de que era una técnica en introducción y que se estaba haciendo la excepción ante la imposibilidad de dar otro resultado y tomando en cuenta la ansiedad materna.
Aun cuando la muestra analizada en este trabajo es limitada en número, lo cual no ha permitido enfrentarnos a diversos problemas del DPC mediante la tecnología FISH en células en interfase, sí consideramos que estos resultados iniciales son válidos y que debemos implementar este diagnóstico en nuestro medio. Todos los resultados obtenidos por FISH, al menos por el momento, deben ser corroborados mediante la citogenética convencional. Esta tecnología debe ser aplicada en casos de alto riego de aneploidías de los cromosomas 21,13, 18 y X y/o cuando una determinada situación clínica así lo demande. Debe ser valorada cuidadosamente en aquellos casos de una elevada ansiedad materna para ofertar en un corto periodo de tiempo un resultado preliminar a la pareja, pero el resultado final y concluyente debe ser complementado mediante el examen cromosómico de las células fetales.
REFERENCIAS BIBLIOGRÁFICAS
1. Ward DC, Gersen SL, Carelli MP, McGuire NM, Dackowski WR, Weinstein M, et al. Rapid prenatal diagnosis of chromosomal aneuploidies by fluorescence in situ hybridization: clinical experience with 4500 specimens. Am J Hum Genet. 1993;52:854-65.
2. Nederlof PM, van der Flier S, Wiegant J, Raap AK, Tanke HJ, Ploem JS, et al. Multiple fluorescence in situ hybridization; Cytometry. 1990;11:126-31.
3. Philip J, Bryndorf T, Christensen B. Prenatal aneuploidy detection in interphase cells by fluorescence in situ hybridization (FISH); Prenat. Diagn. 1994;14:1203-15.
4. Eiben B, Trawicki W, Hammans W, Goebel R, Pruggmeyer M, Epplen JT. Rapid prenatal diagnosis of aneuploidies in uncultured amniocytes by fluorescence in situ hybridization. Evaluation of > 3000 cases; Fetal Diagn. Therapy. 1999;14:193-7.
5. Margaret J. Barch, Turid Knutsen, Jack L. Spurbeck. The AGT Cytogenetics Laboratory Manual. 3rd Revised Edition. Philadelphia: Lippincott Williams and Wilkins; 1997.
6. Jobanputra V, Kumar K, Kucheria K. Prenatal detection of aneuploidies using fluorescence in situ hybridization: A preliminary experience in an Indian set up J. Biosci. 2002;27(2):155-63.
7. Anne E, Wiktor BS, Van Dyke L, Stupca PJ, Ketterling R, Thorland EC, et al. Preclinical validation of fluorescence in situ hybridization assays for clinical practice. Genetics IN Medicine. 2006;8(1).
8. Tepperberg J, Pettenati MJ, Rao PN, Lese CM, Rita D, Wyandt H, et al. Prenatal diagnosis using interphase fluorescence in situ hybridization (FISH): 2-year multi-center retrospective study and review of the literature. Prenat Diagn. 2001;21:293-301.
9. Weremowicz S, Sandstrom DJ, Morton CC, Niedzwiecki CA, Sandstrom MM, Bieber FR. Fluorescence in situ hybridization (FISH) for rapid detection of aneuploidy: experience in 911 prenatal cases. Prenat Diagn. 2001;21:262-9.
10. Liehr T, Ziegler M. Rapid Prenatal Diagnosis in the Interphase Nucleus: Procedure and Cut-off Rates Journal of Histochemistry & Cytochemistry. 2005;53(3):289-91. Disponible en: http://jhc.sagepub.com/content/53/3/289.full.pdf+html
11. Luquet I, Mugneret F, Athis PD, Nadal N, Favre B, Abel C, et al. French multi-centric study of 2000 amniotic fluid interphase FISH analyses from high-risk pregnancies and review of the literature. Annales de Génétique. 2002;45:77-88.
12. American College of Medical Genetics/American Society of Human Genetics Statement, Technical and clinical assessment of fluorescence in situ hybridisation: an ACMD/ASHG position statement, 1, Technical considerations. Genet Med. 2000;2:356-61.
13. Marguerat P, Gaide AC, Thonney F, Schorderet D. Recours au diagnostic prenatal rapide par l'analyse FISH des cellules du liquid amniotique non cultivées. Rev Med Suisse Romande. 2000;120:401-7.
14. Sawa R, Hayashi Z, TanakaT. Rapid detection of chromosome aneuploidies by prenatal interphase FISH (fluorescence in situ hybridization) and its clinical utility in Japan. J Obstet Gynaecol Res. 2001;27:41-74.
Recibido: 3 de septiembre de 2011.
Aprobado: 18 de septiembre de 2011.
Luis A. Méndez Rosado. Centro Nacional Genética Médica. Calle 146 esq 31. Cubanacán, Playa. La Habana, Cuba. Correo electrónico: albermen@infomed.sld.cu