My SciELO
Services on Demand
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Medicina Tropical
On-line version ISSN 1561-3054
Rev Cubana Med Trop vol.55 no.3 Ciudad de la Habana Sept.-Dec. 2003
Artículos originales
Instituto de Medicina Tropical "Pedro Kourí"
Aplicación de la secuenciación nucleotídica de VP1 a la identificación de Enterovirus humanos
Dr. Julio Antonio Barrios Olivera,1 Lic. Luis Sarmiento Pérez,2 Lic. Odalys Valdés,3 Dr. Pedro Mas Lago4 y Téc. Rosa Palomera Puentes5
Resumen
Se describió la introducción de un método molecular para la identificación de los Enterovirus basado en la amplificación, secuenciación y análisis filogenético de la proteína VP1. Se demostró que este método reduce grandemente el tiempo requerido para la identificación de los Enterovirus aislados y resulta de gran utilidad en la caracterización de aislamientos difíciles de tipificar, con el empleo de los reactivos inmunológicos de rutina. Por la rapidez de ejecución de la técnica reviste vital importancia su uso durante epidemias, dada la rápida determinación del agente causal.
DeCS: INFECCIONES POR ENTEROVIRUS; MODELOS MOLECULARES.
Los Enterovirus constituyen un gran género dentro de la familia Picornaviridae, se describen 64 serotipos antigénicos basados en los ensayos de neutralización. Al nivel mundial, más de 500 000 000 de personas se infectan cada año, resultando en cientos de miles los casos de enfermedad febril indiferenciada, rash, enfermedades gastrointestinales, meningitis aséptica, parálisis y enfermedad neonatal severa. Más recientemente, los Enterovirus han sido implicados en algunas enfermedades crónicas como la diabetes mellitus tipo 1 y la miocardiopatía dilatada.1
La identificación de los Enterovirus es esencial en la vigilancia epidemiológica, incluidos los poliovirus salvajes, en los estudios de correlación de serotipos y enfermedades, en el reconocimiento de nuevos serotipos y en el adecuado tratamiento de las infecciones enterovirales en neonatos e inmunodeprimidos.2
Para la identificación de estos agentes causales se emplea el aislamiento viral y la posterior identificación mediante ensayos de neutralización. Este proceder resulta muy laborioso, requiere de semanas para su culminación y en ocasiones es imposible identificar el serotipo productor de la enfermedad debido a los cambios antigénicos, a las recombinaciones, al fenómeno de agregación viral o a la presencia de varios especímenes en la muestra.3
En el presente trabajo se describió la introducción de un método molecular para la identificación de los Enterovirus basados en la amplificación, secuenciación y análisis filogenético de la proteína VP1.
Métodos
Muestra: se seleccionaron un total de 12 cepas de Enterovirus aisladas en el año 1998 en el Laboratorio Nacional de Referencia de Enterovirus, Instituto de Medicina Tropical "Pedro Kourí" (IPK), Cuba. Estas cepas procedían de niños sanos, ninguna de las cuales pudo ser identificada por la prueba de neutralización con el empleo de la mezcla de Lim-Benyesh-Melnick (LBM).
Extracción de ácido ribonucleico (ARN): para la obtención del ARN viral de la muestra se utilizó el método del TRIZOL LS Reagent comercial (BRL Life Technologies) según las instrucciones del fabricante.
Síntesis del ácido dexosirribonucleico complementario (ADNc): la síntesis del ADNc se realizó con cebadores al azar, con la utilización del estuche comercial de preamplificación ThermoScript (Gibco BRL, Life Technologies).
Reacción en cadena de la polimerasa (RCP): para la amplificación de VP1 se emplearon 2 juegos de cebadores (011-012 y 011-040) que fueron diseñados por Oberste y otros4 (tabla 1). Para esto se utilizó el estuche comercial AmpliTaq Gold (Applied Biosystems), siguiendo las instrucciones del fabricante. Los tubos se colocaron en un termociclador (PTC 100TM. MJ Research, Inc. Watertown Mass.) a la temperatura de 94 ºC durante 10 min, para la activación de la enzima, a lo que siguieron 30 ciclos con temperaturas de 95 ºC por 45 s, 42 ºC por 45 s y 68 ºC durante 45 s; finalmente la mezcla de reacción se mantuvo durante 10 min a 70 ºC.
Tabla 1. Secuencia nucleotídica y posición en el genoma de los cebadores empleados para la amplificación y secuenciación de VP1
| Primer | Secuencia | Gen | Posición nucleotídica a |
| 011 | GCICCIGAYTGITGICCRAA | 2A | 3408-3389 |
| 012 | ATGTAYGTICCICCIGGIGG | VP1 | 2951-2970 |
| 040 | ATGTAYRTICCIMCIGGIGC | VP1 | 2951-2970 |
a correspondiente con la secuencia PV1-Mahoney (No. en Genbank J02281).
Para la detección del producto amplificado se analizaron 10 µL de cada tubo de reacción en una electroforesis en gel de agarosa 2 % en tampón TBE (Tris-Borato 0,09M, EDTA 2 mM, pH 8). En cada corrida se incluyó el patrón de peso molecular FX174 RF DNA/Hae III Fragments (Gibco BRL, Life Technologies). La coloración de los geles se realizó con una solución de bromuro de etidio a una concentración final de 0,1-0,2 µg/mL, y se visualizaron las bandas de amplificación en un transiluminador de luz ultravioleta (LKB).
Purificación del producto de la RCP: el producto de la RCP se purificó para su posterior secuenciación mediante la utilización del estuche comercial QIAquick (QIAGEN, Santa Clarita, CA).
Secuenciación: se realizó secuenciación manual mediante el método de terminación de la cadena empleando el estuche comercial Thermo Sequenase Radiolabeled Terminator Cycle Sequencing (USB).
Determinación del serotipo: se determinó por comparación de la secuencia amplificada de VP1 con las secuencias de todos los Enterovirus humanos y otros Picornavirus, publicadas en la base de datos GenBank. Se consideró que más de 75 % de homología en la secuencia nucleotídica (88 % en la secuencia aminoacídica) era indicativo de cepas de un mismo serotipo, según lo descrito por Oberste y otros.5
Análisis filogenético: el árbol filogenético fue construido utilizando el programa MEGA (versión 2.1)6 y se basó en el análisis de la secuencia VP1, mediante el método de Neighbor-joining (bootstrap 1000).7
Microneutralización: una vez identificado el serotipo viral por el análisis filogenético, se procedió a confirmar la identificación mediante la prueba de neutralización con el empleo de las mezclas de la J-P de LBM, siguiendo el protocolo de la OMS.8
Resultados
Amplificación de ácidos nucleicos
La secuencia nucleotídica de la proteína VP1 de los Enterovirus parece estar correlacionada con los serotipos enterovirales. Esta proteína presenta una gran variabilidad, por lo que resulta imposible la amplificación de esta parte del genoma en todos los Enterovirus, con el uso de un solo juego de cebadores. En este trabajo, cuando se emplearon los cebadores 011-012, no se obtuvo la amplificación en ninguna de las cepas del estudio. Con el empleo de los primer 011-040 se obtuvo un producto de 457 pb en las 12 cepas estudiadas.
Tipificación molecular basada en el análisis de la región VP1
Las secuencias de la región VP1 de las cepas estudiadas se compararon con todas las secuencias de Enterovirus publicadas en Genbank, encontrándose en todas entre 78 a 86 % de homología nucleotídica y más de 95 % de homología aminoacídica con algún serotipo de Enterovirus (tabla 2). Esto permitió identificar 3 cepas de Coxsackie A4, 2 Coxsackie A8 (CA8) y 7 Coxsackie A10 (CA10). En la figura se muestra el análisis filogenético de las cepas estudiadas. Como puede observarse, todas se agrupan en correspondencia con el serotipo al cual pertenecen.
Tabla 2. Porcentaje de identidad nucleotídica y aminoacídica de las cepas estudiadas
| Cepas | Serotipo identificado | Identidad nucleotídica (%) | Identidad aminoacídica (%) |
| 2-49 | CA10 | 78 | 96 |
| 13-53 | CA8 | 82 | 96 |
| 20-3 | CA4 | 86 | 95 |
| 23-5 | CA4 | 85 | 97 |
| 23-33 | CA4 | 86 | 97 |
| 23-57 | CA10 | 79 | 96 |
| 24-1 | CA8 | 81 | 96 |
| B2-38 | CA10 | 78 | 96 |
| B3-44 | CA10 | 79 | 96 |
| B5-19 | CA10 | 78 | 96 |
| B6-4 | CA10 | 78 | 96 |
| B6-17 | CA10 | 79 | 96 |
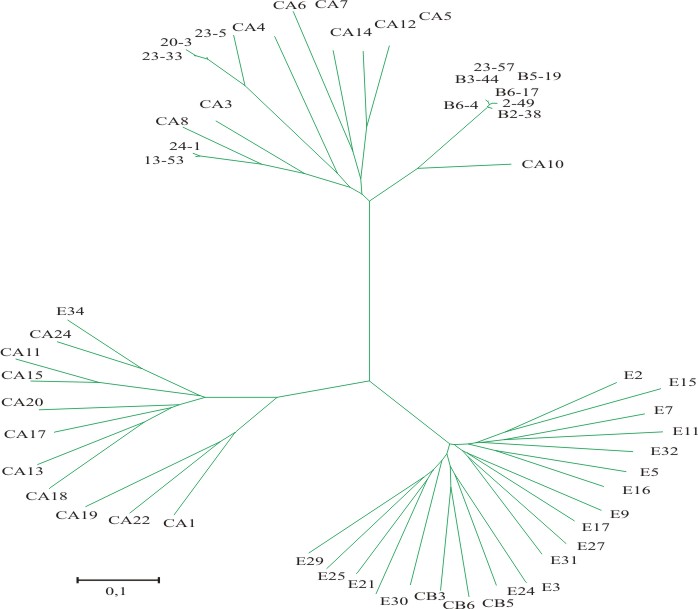
Fig. Árbol filogenético de la región VP1.
Remicroneutralización
Después de conocer el serotipo viral mediante el análisis filogenético de la región VP1, se procedió a su confirmación empleando las mezclas de J-P de LBM. Hubo una correspondencia entre los serotipos identificados por ambas técnicas.
Discusión
El estudio de la región 5' no codificante (5'NTR) de los Enterovirus mediante métodos moleculares, permite la rápida y específica detección de todos los miembros de este género viral.9 Sin embargo, por la gran variabilidad que presenta esta región dentro de un mismo serotipo no resulta útil para la identificación de los diferentes serotipos.10-12
Otros estudios filogenéticos sugirieron que el empleo de la región de unión entre las proteínas VP4-VP2 para la identificación de los distintos serotipos podía ser de más utilidad que la región 5'NTR, sin embargo, no obtuvieron resultados favorables para todos los serotipos virales.13-15
La región de unión entre VP1 y 2A ha sido ampliamente usada en los estudios de patrones de transmisión de los Poliovirus y se ha podido comprobar que las secuencias de VP1 de estos virus se agrupan en correspondencia con los serotipos homólogos.16
Los serotipos de los Enterovirus están bien definidos sobre la base de la neutralización. En la proteína VP1 se encuentran ubicados los epítopes neutralizantes de mayor importancia.17,18 Recientemente Oberste y otros describieron que la secuencia de la proteína VP1 contiene la información específica de serotipo de todos los Enterovirus. Esto ha permitido el desarrollo de métodos moleculares para la identificación, tanto de los serotipos tradicionales de los Enterovirus como el descubrimiento de otros nuevos.19
Los aislamientos incluidos en este estudio no pudieron ser identificados por la prueba de neutralización empleando las mezclas de la A-H de LBM, ni tampoco antisueros específicos contra Poliovirus. Esta mezcla contiene anticuerpos contra solo 42 serotipos de Enterovirus, de los 64 conocidos. Una de las explicaciones más sencillas para que falle la neutralización es que el serotipo aislado no esté incluido dentro de la mezcla. En este estudio específico todas las cepas pertenecieron al grupo de los Coxsackie A. Hasta hace unos años no existían sistemas celulares sensibles para el aislamiento de estos virus, por lo que sus antisueros específicos no se encuentran incluidos en la mezcla de la A-H de LBM que son los que se usan de rutina en los laboratorios.
Todas las cepas fueron amplificadas con el empleo de los cebadores 011-040, no fue así para los cebadores 011-012. Esto concuerda con lo reportado por Oberste y otros,4 quienes encuentran que la mayoría de los Coxsackie del grupo A son amplificados por los cebadores 011-040. Una de las desventajas mayores de este método es que como la VP1 es una proteína tan variable, es muy difícil amplificar todos los Enterovirus humanos con solo un juego de cebadores. Más recientemente Caro y otros describen un juego de cebadores que amplifican VP1-2C dl 92,2 % de los Enterovirus.20
Se logró identificar 100 % de las cepas siguiendo los criterios de Oberste y otros, reafirmándose de esta forma la efectividad del método para la tipificación de los Enterovirus.
La meta de la erradicación de la poliomielitis está muy cerca y la vacunación con la vacuna de Poliovirus atenuada (VPO) cesará. El nicho ecológico ocupado por los Poliovirus quedará vacante, lo que generará que otros Enterovirus no Polio ocupen el papel protagónico. El incremento de la circulación, detección, identificación y evolución de los Enterovirus no Polio, así como la detección de nuevas cepas epidémicas, obliga a contar con mejores herramientas para el diagnóstico de estos problemas de salud. El contar en Cuba con estas nuevas metodologías como complemento de las clásicas, permitirá estar en mejores condiciones para enfrentar la nueva época que se avecina.
Summary
The introduction of a mollecular method to identify the Entoviruses based on the amplification, sequencing and phylogenetic analysis of protein VP1 was described. It was proved that this method reduces significantly the time required for the identification of the isolated Entoviruses and that it is very useful in the characterization of isolates which are difficult to typify by the routine immunoloigical reagents. As it is a very fast technqiue, its use is very important during epidemics to determine the causal agent rapidly.
Subject headings: ENTEROVIRUS INFECTIONS; MODELS, MOLECULAR.
Referencias bibliográficas
- 1. Pallansch MA, Roos RP. Enterovirus: Poliovirus, Coxsackievirus, Echovirus and newer enteroviruses En: Fields BN, Knipe DM. Virology. 4nded. New York: Raven Press; 2001. p.549-609.
- Muir PU, Kammerer K, Korn MN, Mulders T, Poyry B, Weissbrich R, et al. Molecular typing of Enterovirus: current status and future requirements. Clin Microbiol Rev 1998;11:202-27.
- Kapsenberg JG, Ras A, Pallansch MA. Improvement of Enterovirus neutralization by treatment with sodium deoxycholate or chloroform. Intervirology 1980;12:329-34.
- Oberste MS, Maher K, Kilpatrick DR, Flemister MR, Brown BA, Pallansch MA. Typing of human Enterovirus by partial sequencing of VP1. J Clin Microbiol 1999;37:1288-93.
- Oberste MS, Maher K, Flemister MR, Marchetti G, Kilpatrick DR, Pallansch MA. Comparison of classic and molecular approaches for the identification of untypable enteroviruses. J Clin Microbiol 2000;38:1170-4.
- Kumar S, Tamura K, Jakobsen IB, Nei M. MEGA2: Molecular evolutionary genetics analysis software, Arizona State University, Tempe, Arizona, USA, 2001
- Saitou N, Nei M. The neighbor-joining method: a new method for recon strutting phylogenetic trees. Mol Biol Evol 1987;4:406-25.
- Melnick J, Winberly I. Lyophilized combination pools of Enterovirus equine antisera. New LBM prepared from reserves of antisera stored frozen for 2 decades. Bull WHO 1985;63:543-50.
- Rotbart HA, Romero JR. Laboratory diagnosis of enteroviral infections. En: Rotbart HA. Human Enterovirus Infections. Washington, DC: ASM Press;1995.
- Kopecka H, Brown B, Pallansch MA. Genotypic variation in Coxsackie B5 isolates from three different outbreaks in the United States. Virus Res 1995;38: 125-36.
- Diedrich S, Driesel G, Schreier E. Sequence comparison of echovirus type 30 isolates to other enteroviruses in the 5' noncoding region. J Med Virol 1995;46: 148-52.
- Bailly JL, Borman AM, Lafeuille HP, Kean KM. Natural isolates of echo virus type 25 with extensive variations in IRES sequences and different translational efficiencies. Virology 1996;215:83-96.
- Huttunen P, Santii J, Pulli T, Hyypia T. The major echovirus group is genetically coherent and related to coxsackie B viruses. J Gen Virol 1996;77:715-25.
- Poyry T, Kinnunen L, Hyypia T, Brown B, Horsnell C, Hovi T, et al. Genetic and phylogenetic clustering of Enteroviruses. J Gen Virol 1996;77:1699-717.
- Pulli T, Koskimies P, Hyypia T. Molecular comparison of Coxsackie A virus serotypes. Virology 1995 ;211:30-8.
- Kew OM, Mulders MN, Lipskaya GY, Da silva EE, Pallansch MA. Molecular Epidemiology of Polioviruses. Semin Virol 1995;6:401-14.
- Mateu MG. Antibody recognition of Picornaviruses and escape from neutralization: structural view. Virus Res 1995;38:1-24.
- Minor PD. Antigenic structure of Picornaviruses. Curr Top. Microbiol Immunol 1990;161:121-54.
- Oberste MS, Maher K, Kilpatrick DR, Pallansch MA. Molecular evolution of the human enteroviruses: correlation of serotype with VP1 sequence and application to picornavirus classification. J Virol 1999;73:1941-8.
- Caro V, Guillot S, Delpeyroux F, Crainic R. Molecular strategy for serotyping of human enteroviruses. J Gen Virol 2001;82:79-91.
Recibido: 2 de junio de 2003. Aprobado: 9 de julio de 2003.
Dr. Julio Antonio Barrios Olivera. Instituto de Medicina Tropical "Pedro Kourí". Apartado 601, Marianao 13, Ciudad de La Habana, Cuba. Correo electrónico: ciipk@ipk.sld.cu
1 Máster en Virología. Especialista de I Grado en Microbiología.
2 Máster en Virología. Licenciado en Microbiología. Aspirante a Investigador.
3 Máster en Virología. Licenciada en Microbiología. Investigadora Agregada.
4 Doctor en Ciencias Médicas. Especialista de II Grado en Microbiología. Investigador de Mérito.
5 Técnico en Microbiología.














