My SciELO
Services on Demand
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Medicina Tropical
On-line version ISSN 1561-3054
Rev Cubana Med Trop vol.63 no.2 Ciudad de la Habana May.-Aug. 2011
ARTÍCULO ORIGINAL
Diagnóstico molecular del virus influenza A (H1N1) 2009 y otros vírus respiratorios, durante la primera ola pandémica en Cuba
Molecular diagnosis of influenza A (H1N1) 2009 virus and other respiratory viruses during the first pandemic wave in Cuba
Suset Oropeza Fernández,I Belsy Acosta Herrera,II Alexander Piñón Ramos,III Odalys Valdés Ramírez,IV Clara Savón Valdés,V Amely Arencibia García,VI Elias Guilarte García,VII Grehete González Muñoz,VIII Ángel Goyenechea Hernández,IX Mayra Muné Jiménez,X Guelsys González Báez,XI Bárbara Hernández Espinosa,XII María G. Guzmán Tirado,XIII Alina Llop HernándezXIV
IEspecialista de II Grado en Microbiología. Investigador Auxiliar. Profesor Auxiliar. Instituto de Medicina Tropical ¨Pedro Tourí¨ (IPK). La Habana, Cuba.
IIEspecialista de II Grado en Microbiología. Investigador Auxiliar. Asistente. IPK. La Habana, Cuba.
IIIMáster en Virología. Investigador Agregado. Instructor. IPK. La Habana, Cuba.
IVDoctor en Ciencias de la Salud. Investigador Titular. Asistente. IPK. La Habana, Cuba.
VDoctor en Ciencias Biológicas. Investigador Titular. Profesor Titular. IPK. La Habana, Cuba.
VILicenciada en Microbiología. IPK. La Habana, Cuba.
VIIMédico Residente de Microbiología. IPK. La Habana, Cuba.
VIIIMaster en Virología. Investigador Auxiliar. Instructor. IPK. La Habana, Cuba.
IXEspecialista de II Grado en Microbiología. Investigador Titular. Profesor Titular. IPK. La Habana, Cuba.
XDoctor en Ciencias de la Salud. Investigador Titular. Profesor Titular. IPK. La Habana, Cuba.
XITécnico en Química Industrial. Asistente Técnico Docente. IPK. La Habana, Cuba.
XIITécnico en Microbiología. Asistente Técnico Docente. IPK. La Habana, Cuba.
XIIIDoctor en Ciencias. Investigador Titular. Profesor Titular. IPK. La Habana, Cuba.
XIVDoctor en Ciencias Médicas. Investigador Titular. Profesor Titular. IPK. La Habana, Cuba.
RESUMEN
Introducción: el primer virus pandémico del siglo XXI, el virus influenza A (H1N1)/2009, emergió en México, a finales del mes de abril de 2009, después de un triple reordenamiento entre virus de influenza de origen aviar, humano y porcino, y desde allí se diseminó por el mundo. Frente a ese evento, en Cuba, se adoptaron medidas determinantes antipandémicas, entre ellas, se reforzó la vigilancia virológica con todas las acciones necesarias.
Objetivos: detectar y confirmar la entrada del agente causal de la pandemia en el país, de forma rápida, oportuna y además, definir la participación de otros virus en la etiología de las infecciones respiratorias agudas.
Métodos: como consecuencia de la vigilancia de laboratorio, entre las semanas epidemiológicas 38 a la 42 de 2009 (meses de septiembre y octubre) se procesó un total de 1 063 muestras clínicas respiratorias (exudado nasofaríngeo, aspirado bronquial y muestras de necropsia de pulmón), detectándose el mayor número de casos confirmados de infección por el nuevo virus en este período, que se correspondió con la primera oleada pandémica en Cuba. El diagnóstico virológico se realizó utilizando un algoritmo de diagnóstico molecular.
Resultados: de las 1 063 muestras, 597 (56,0 %) resultaron positivas. El virus de influenza pandémica A(H1N1)2009, fue el agente etiológico detectado con mayor frecuencia en 306 casos sospechosos (51 %), seguido por el virus influenza A(H3N2) en 228 pacientes(38 %). Otros virus respiratorios se diagnosticaron en 63 muestras clínicas (11 %). El virus pandémico se confirmó en 50 gestantes. Los rinovirus se detectaron con mayor frecuencia en muestras de pacientes con diagnóstico clínico de bronconeumonía y bronquiolitis. Durante el período estudiado se reportó un incremento de la morbilidad, se notificaron 225 825 atenciones médicas por infección respiratoria aguda a mediados del mes de octubre.
Conclusión: el algoritmo de diagnóstico molecular empleado para la confirmación de los virus influenza y otros virus respiratorios demostró ser sensible, específico y efectivo para garantizar la vigilancia virológica sistemática en nuestro país durante la fase pandémica.
Palabras clave: pandemia, influenza virus, diagnóstico molecular.
ABSTRACT
Introduction: the first pandemic virus of the 21st century - the influenza A (H1N1)/2009 virus-appeared in Mexico in April 2009 after triple reassortment of influenza strains of avian, human and pig origin and from there, it was spread worldwide. With the purpose of facing up to this event, Cuba adopted anti-pandemic measures including the virology surveillance using all necessary actions.
Objectives: the detection and validation of the entry of the causative agent of pandemic into the country in a fast and timely way, in addition to the definition of involvement of other viruses in the etiology of acute respiratory infections.
Methods: as a result of the lab surveillance, from the 38th to the 42nd epidemiological weeks (September and October, 2009), 1 063 respiratory clinical samples were processed (nasopharyngeal exudates, bronchial aspirates and lung necropsy samples). The highest number of confirmed cases caused by the new virus was detected in this period that represented the first pandemic wave in Cuba. Diagnosis was based on molecular diagnosis algorithm.
Results: out of the 1 063 samples, 597 (56.0 %) were positive. The pandemic influenza A (H1N1) virus was the most commonly detected etiological agent in 306 suspected cases (51 %) followed by influenza A (H3N2) virus in 228 cases (38 %). Other respiratory viruses were diagnosed in 63 clinical samples (11 %). The pandemic virus was confirmed in 50 pregnant women. Rhinoviruses were identified more frequently in those samples from patients with clinical diagnosis of bronchial pneumonia and broncholitis. Morbidity increased during this period; 225 825 medical consultations were notified due to acute respiratory infections mid-October 2009.
Conclusions: the molecular diagnosis algorithm proved to be sensitive, specific and effective to assure the systematic virological surveillance in our country during the pandemic phase
Key words: pandemic, influenza virus, molecular diagnosis.
INTRODUCCIÓN
La influenza o gripe es una infección respiratoria aguda (IRA), cuyo agente causal es el virus de la influenza. Se asocia a elevadas tasas de morbilidad y mortalidad generalmente relacionada con la neumonía; considerables costos en los servicios de la salud pública y en la sociedad.1
La ocurrencia anual de las epidemias de influenza se mantiene por los continuos procesos de cambios antigénicos menores o drift, debidos a la acumulación de mutaciones puntuales en los genes que codifican las glicoproteínas situadas en la superficie viral: la hemaglutinina (HA) y la neuraminidasa (NA). A intervalos impredecibles ocurre la emergencia o reemergencia de una cepa pandémica, con características antigénicas y genéticas nuevas, originadas por cambios mayores o shift de la HA, la NA o ambas, para la cual la mayoría de los individuos poseen poca o ninguna inmunidad.2
Bajo estas condiciones se gestó el nuevo virus de influenza pandémico, el A (H1N1)2009, con un genoma constituido por un triple reordenamiento entre virus de influenza de origen aviar, humano y porcino.3 Se identificó por primera vez en EE. UU. y México en abril de 2009, diseminándose a todos los continentes.
La enfermedad en general se ha comportado de forma similar a la influenza estacional, pero de forma inusual ha afectado a los grupos más jóvenes y gestantes. En la actualidad se le atribuye a esta pandemia un impacto moderado en los servicios de salud.
La Organización Panamericana de la Salud (OPS), hasta el 16 de agosto de 2010, notificó 8 567 fallecidos confirmados en la región de las Américas, y de ellos 83 pertenecen a Cuba.4,5
Desde 1997, con el salto de especie del virus de influenza aviar A (H5N1),6 considerado el agente con mayores probabilidades para producir una pandemia y, posteriormente en 2003, la emergencia de un nuevo coronavirus humano, el coronavirus SARS,7 estos eventos permitieron a la comunidad científica internacional y a los sistemas nacionales de salud acelerar la preparación mundial para el enfrentamiento de una futura pandemia por virus de influenza, oportunidad sin precedentes.
El 11 de junio de 2009 fue declarada la fase pandémica por la Organización Mundial de la Salud (OMS), sin embargo, desde el 28 de abril y como parte del Plan Nacional de enfrentamiento a la influenza pandémica A(H1N1), el Laboratorio Nacional de Referencia de virus influenza (LNRI) y el Laboratorio Nacional de Referencia para otros virus respiratorios (LNRVR) del Instituto de Medicina Tropical "Pedro Kourí" (IPK), estuvieron responsabilizados con la detección rápida y oportuna de la presencia del nuevo virus en Cuba, así como el monitoreo de la circulación de este agente y otros virus respiratorios que posibilitara la alerta a las autoridades nacionales de salud para la implementación de acciones de prevención y control.
Teniendo en cuenta los resultados de la vigilancia de laboratorio, mediante un algoritmo de diagnóstico molecular, se describe en el presente trabajo, la caracterización de la circulación del virus pandémico y otros virus respiratorios en el período comprendido entre las semanas epidemiológicas 38 a la 42 de 2009 en Cuba.
MÉTODOS
Muestras clínicas
Entre el 13 de septiembre y el 17 de octubre de 2009, el LNRI procesó un total de 1 063 muestras clínicas (exudado nasofaríngeo, aspirado bronquial y muestras de necropsia de pulmón) procedentes de pacientes que cumplieron los criterios de definición de caso sospechoso de infección por el virus influenza A (H1N1) pandémico, incluido dentro del Plan Nacional de enfrentamiento a este virus, disponible en todas las unidades de los diferentes niveles de atención del Sistema Nacional de Salud. Del total de muestras procesadas, 704 provenían de pacientes con diagnóstico clínico de enfermedad tipo influenza (ETI), 328 de pacientes con infección respiratoria aguda grave (IRAG) y 31 fallecidos por IRAG.
Todas las muestras se colectaron en 3 mL de medio de transporte (universal transport medium, Copan Innovation, CA, USA) y se trasladaron al LNRI en las primeras 24 h. A su arribo al LNRI, se dispensaron y procesaron de manera inmediata. Una alícuota de cada muestra fue conservada en congelación a - 80 ºC.
Procedimiento
Todas las muestras clínicas fueron procesadas utilizando los protocolos para la detección de 17 virus respiratorios, según se detalla a continuación. El trabajo se desarrolló en gabinetes de seguridad clase II.8
Extracción de ácidos nucleicos
La extracción de ARN y ADN total se realizó a partir de las muestras clínicas (volumen inicial de 140 mL) mediante los estuches comerciales QIAamp Viral RNA (QIAGEN Ref 52906) y QIAamp Viral DNA (QIAGEN Ref 51106),9 siguiendo el protocolo descrito por el fabricante. El ARN extraído se procesó de manera inmediata cumplimentando el protocolo para la amplificación de ácidos nucleicos.
RCP en tiempo real para el diagnóstico de influenza pandémica A (H1N1) 2009
Se realizó siguiendo las instrucciones recomendadas por el Centro para el Control de Enfermedades y su Prevención (CDC, siglas en inglés), Atlanta, Georgia, Estados Unidos y publicadas por la OMS.10
Ensayos de transcripción reversa-reacción en cadena de la polimerasa (TR-RCP)
Con el objetivo de realizar el diagnóstico diferencial con virus influenza A, B y C estacionales (Inf A, Inf B e Inf C), y otros virus respiratorios (virus sincitial respiratorio humano A y B [VSRH A y B], adenovirus [AdV], virus parainfluenza humano 1,2,3,4a y 4b [1-4/ VPIH], enterovirus [EV], rinovirus [RV], metapneumovirus humano [MPVH], bocavirus humano [BoVH] y coronavirus humanos [CoVH] 229E y OC43) se emplearon varios ensayos de TR-RCP convencional publicados previamente.11-14
RESULTADOS
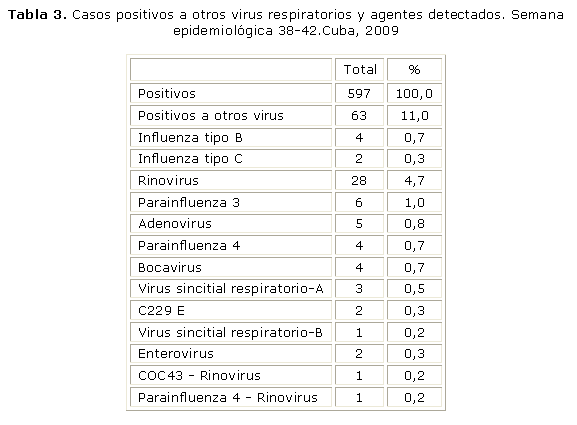
Como resultado de la vigilancia de laboratorio, entre las semanas epidemiológicas 38 a la 42 de 2009 (meses de septiembre y octubre), se procesó un total de 1 063 muestras clínicas respiratorias; 56 % (597) resultó positivo a algún agente viral. El virus de la influenza pandémica A (H1N1) 2009 fue el agente etiológico detectado con mayor frecuencia en 306 casos sospechosos (51 %), seguido por el virus influenza A (H3N2) en 228 pacientes (38 %) (tabla 1). Otros virus respiratorios se diagnosticaron en 63 muestras clínicas (11 %) (Fig.).
De los 306 casos confirmados de infección por el virus pandémico, 239 presentaron formas leves de la infección, 58 desarrollaron IRAG y 9 casos fallecidos, confirmados a partir del estudio de muestras de pulmón.
El mayor número de casos diagnosticados (86) de influenza pandémica A (H1N1) 2009 y de influenza A(H3N2) que alcanzó la cifra de 85, se detectaron en la provincia La Habana. En orden de frecuencia, la segunda más afectada fue la provincia de Las Tunas, se confirmaron 30 pacientes positivos al virus pandémico y 20 al virus influenza A (H3N2).
Como parte del estudio etiológico de brotes de IRA, se investigó un total de 223 muestras clínicas y 154 (69 %) resultaron positivas a algún agente viral; 57 % de positividad correspondió al virus pandémico (88), 51 muestras resultaron positivas a influenza A(H3N2)(33 %) y 10 % de positividad se encontró para otros virus respiratorios.15
Se procesaron 118 muestras clínicas de gestantes, en las cuales se encontró 61 % de positividad a alguno de los agentes virales investigados (72). La infección por el virus pandémico se confirmó en 69 % de las muestras positivas, de ellas, 34 % procedente de pacientes con diagnóstico clínico de IRAG. El virus influenza A (H3N2) fue detectado en 20 pacientes gestantes y solo 1 de ellas con IRAG.
En la tabla 2 se puede observar la distribución de casos confirmados al virus pandémico en los diferentes grupos de edades. El mayor número de casos se encontró en las edades comprendidas entre 15 y 24 años (102 casos), seguido por el grupo de 25 a 59 años donde se diagnosticó en 80 casos y el grupo de 10 a 14 donde se registraron 61 casos positivos.
Entre los 9 fallecidos en los que se detectó el virus pandémico, 8 estaban incluidos en las edades comprendidas entre 15 y 59 años, y 1 caso de 75 años de edad.
El diagnóstico diferencial de otros virus respiratorios posibilitó identificar al rinovirus en 28 muestras clínicas (5 %). Se identificaron otros virus, aunque en menor proporción (tabla 3).
DISCUSIÓN
En las semanas objeto de estudio se logró el monitoreo de la circulación de 17 agentes virales involucrados en la etiología de las IRA. Este resultado fue posible debido al fortalecimiento de las capacidades diagnósticas del LNRI del IPK, que hoy aplica métodos de diagnóstico molecular efectivos,10-14 de gran sensibilidad y especificidad; los cuales posibilitaron que durante 2009 se pudiera ofrecer una respuesta rápida y segura frente a un evento cuya magnitud mundial era impredecible.
Los resultados demuestran que los virus influenza son uno de los principales agentes etiológicos de las IRAs en Cuba, al confirmarse los tipos A, B o C en 90 % de las muestras positivas. Dentro de estos agentes, como era de esperar, teniendo en cuenta la fase pandémica a la que se enfrentaba el mundo, se detectó un predominio significativo de la positividad al nuevo virus pandémico (51 %), lo que confirmó la amplia circulación de este agente en el país, que ocasiona casos esporádicos, brotes, enfermedad leve, grave y muertes. Estos hallazgos están en relación con el comportamiento de un virus pandémico en una población carente de inmunidad específica para un agente emergente.2,6
No es primera vez que la humanidad se enfrenta a un evento pandémico. Durante el siglo xx, tres grandes pandemias de influenza: la "gripe española" A (H1N1) en los años 1918-1919 que causó entre 20 y 50 millones de fallecidos; la "gripe asiática" producida por el subtipo A(H2N2) en los años 1957-1958 y la de "Hong- Kong" por el subtipo A(H3N2) en los años 1968-1969, ambos causantes de 1 y 4 millones de muertes, respectivamente.2
La influenza asociada a la neumonía ha ocupado entre el cuarto y quinto lugar dentro de las causas generales de muerte15 en Cuba; en los últimos 10 años, al igual que en el resto del mundo constituye la primera causa de consultas médicas. Sin embargo, cada año el número de atenciones médicas por esta causa experimenta variaciones, las cuales se pudieron constatar en el período en que se describen los resultados.
Si se comparan los datos de morbilidad por IRA, reportados por la Subdirección de Epidemiología del IPK en la semana 35 con los reportados en la semana 38, es posible detectar un incremento significativo en el número de atenciones médicas desde 108 815 a 167 372, respectivamente. Estos valores experimentaron un aumento mayor hacia la semana 41, donde se notificaron 225 825 atenciones médicas. Como se puede apreciar en los resultados de laboratorio presentados en este trabajo, en igual período en el que fueron reportados los mayores números de atenciones médicas se confirmó el mayor número de casos de infección por el virus influenza pandémica A (H1N1) 2009, que desplazó en su totalidad de la circulación al virus de influenza estacional A (H1N1) y parcialmente al virus influenza estacional A (H3N2), subtipo dominante en la circulación en el país y que ha sido responsable de epidemias anuales y casos de IRAG.16 Al considerar los resultados clínicos, epidemiológicos y de laboratorio en el período estudiado, es posible afirmar hoy, que entre las semanas epidemiológicas 38 a la 42 de 2009 ocurrió la primera ola pandémica en Cuba (tabla 1, Fig.).
Resulta interesante que en la provincia La Habana, se observó la cocirculación del virus pandémico con rangos similares al subtipo de influenza A (H3N2), lo que no ocurrió en ninguna de las restantes provincias.
El monitoreo diario de la ocurrencia de brotes y su estudio por el laboratorio constituyó un pilar fundamental de la vigilancia de la circulación del virus pandémico en Cuba. Es de señalar que en el período durante el cual se presentan estos resultados se investigó 100 % de los brotes notificados y el estudio etiológico demostró al virus pandémico como el principal agente causal, seguido por el virus influenza estacional A(H3N2). Estos resultados reafirman la característica epidémica de los virus influenza y su rápida diseminación, con gran impacto cuando el número de susceptibles es mayor, como ocurre con la emergencia de un virus pandémico.
Desde los inicios de la circulación del virus pandémico se notificó la asociación entre embarazo y severidad de la infección por el nuevo virus.17 Teniendo en cuenta que la población de gestantes era considerada un grupo de riesgo para el desarrollo de formas graves de la infección, se llevó a cabo en Cuba una vigilancia intensificada clínica, epidemiológica y de laboratorio sobre este grupo de pacientes. Bajo esta consideración, el sistema de vigilancia estuvo encaminado a la pesquisa de 100 % de las gestantes con infección respiratoria aguda y el procedimiento personalizado por los profesionales de la salud de los diferentes niveles de atención sobre la base de un protocolo establecido con ese propósito, con el beneficio del diagnóstico y tratamiento temprano. El estudio de laboratorio de este grupo de pacientes constituyó una prioridad dentro de la vigilancia y demostró el mayor porcentaje de positividad para el virus pandémico en gestantes con IRAG, que alcanzó 34 % de positividad.
Observaciones de pasadas epidemias y pandemias de influenza han identificado a las mujeres embarazadas como un grupo vulnerable para la infección y fallecimiento por estos virus.18,19 Durante la pandemia de influenza asiática, 1957-1958, 10 % de las muertes en New York involucró a mujeres embarazadas, y en Minnesota, 50 % de los fallecimientos ocurrió en mujeres durante el embarazo.20-22
Diversos factores influyen en la vulnerabilidad de las embarazadas: los cambios inmunológicos, la demanda de ventilación aumentada, la capacidad funcional residual disminuida y la presión oncótica.23
En el desarrollo de las infecciones virales, se ha visto que tanto la inmunidad innata como la adquirida desempeñan un papel fundamental en la eliminación del virus.24 En particular un patrón de respuesta celular Th1 está asociado con el aclaramiento de las infecciones virales y el cese de la replicación. Las mujeres durante el embarazo presentan cambios hormonales relacionados con este estado fisiológico (aumento de los niveles de estrógeno y de progesterona) y consecuentemente trastornos inmunológicos. El predominio de las hormonas esteroideas pudiera incrementar la replicación viral, regulando las moléculas de la respuesta inmune que inhiben estos eventos.25 Además podrían añadirse determinantes antigénicos del virus pandémico,26 que lograran agudizar el estado de tolerancia fisiológica que presentan estas mujeres y producir una disminución notable en la respuesta Th1, con una respuesta proinflamatoria carente de control, con la consecuente liberación de mediadores inflamatorios tóxicos, en el sitio donde está ocurriendo la respuesta inmune. Diversas investigaciones son necesarias para contribuir al esclarecimiento de la patogénesis de la infección por el virus influenza pandémica A (H1N1) 2009 en el embarazo.
En contraste con las temporadas epidémicas, se observó una proporción más baja en las tasas de mortalidad en los mayores de 60 años, que resultó marcadamente alta en personas jóvenes; algunos autores plantean que estas edades constituían un factor de riesgo para fallecer,27,28 lo que concuerda con los resultados del presente trabajo donde 89 % de los fallecidos confirmados de infección por el virus pandémico durante la primera oleada en Cuba se encontraban entre las edades comprendidas entre 15 y 59 años.
Resulta importante destacar que después del virus influenza pandémico y el subtipo A (H3N2), los rinovirus constituyeron el tercer agente detectado en orden de frecuencia, durante el período de estudio. Aunque los resultados abarcan un período corto de tiempo, lo que dificulta el establecer asociaciones con reportes previos, es de destacar que a partir del empleo de las técnicas de diagnóstico molecular para la vigilancia de laboratorio de las IRA,10-14 cada vez existen más reportes sobre la importancia de los rinovirus en la etiología de este tipo de infección y se considera actualmente, que son el agente que produce hasta 50 % de todos los resfriados a lo largo del año.29,30 Además, son una causa cada vez más frecuente de infección respiratoria baja (bronquiolitis, neumonía y coinfecciones), sobre todo en edades tempranas de la vida. En un estudio realizado en Cuba, se reportó una prevalencia de infección por rinovirus en una población infantil hospitalizada de 11 %.31
Los hallazgos de este trabajo contribuyen a conocer algunas características de la circulación del virus influenza pandémico A (H1N1) 2009, durante un período de tiempo significativo, al cual se conoce como primera oleada en Cuba, sobre la base de los resultados del Laboratorio Nacional de Referencia de Influenza, que evidencia su papel importante para el enfrentamiento de situaciones emergentes de salud como la asociada a la ya finalizada pandemia de influenza por el virus A (H1N1) 2009 en el país.
AGRADECIMIENTOS
A los pacientes y trabajadores del Sistema Nacional de Salud por su generosa cooperación en todas las tareas relacionadas con la pandemia en Cuba.
REFERENCIAS BIBLIOGRÁFICAS
1. Wright PF, Webster RG. Orthomyxoviruses. In: Knipe DM, Howley PM, editors. Fields Virology. Philadelphia: Lippincott Williams and Wilkins; 2001. p. 1533-79.
2. Taubenberger JK, Morens DM. 1918 influenza: the mother of all pandemics. Emerg Infect Dis. 2006;12:15-22.
3. Newman AP, Reisdorf E, Beinemann J, Uyeki TM, Balish A, Shu B, et al. Human case of swine influenza A (H1N1) triple reassortant virus infection, Wisconsin. Emerg Infect Dis. 2008;14(9):1470-2.
4. World Health Organization. Pandemic (H1N1) 2009 - update 82. Available in: http://www.who.int/csr/don/2010_01_08/en/index.html
5. OPS. Pan American Health Organization - Organización Panamericana de la Salud - Actualización Regional. Pandemia (H1N1) 2009 (publicada 12 Ago 2010). [Citado 12 Dic 2010]. Disponible en: http://new.paho.org/hq/index.php?option=com_content&task=view&id=2929&Itemid=2295&lang=es
6. Abdel-Ghafar AN, Chotpitayasunondh T, Gao Z, Hayden FG, Nguyen DH, de Jong MD, et al. Update on avian influenza A (H5N1) virus infection in humans. N Engl J Med. 2008;358:261-73.
7. Ksiazek TG, Erdman D, Goldsmith C, Zaki S, Peret T, Emery S, et al . A novel coronavirus associated with Severe Acute Respiratory Syndrome. N Engl J Med. 2003;348(20):1953-65.
8. OMS. Laboratory biorisk management for laboratories handling pandemic influenza A (H1N1) 2009 virus. [Citado 20 Dic 2010]. Disponible en: http://www.who.int/csr/resources/publications/swineflu/LaboratoryHumanspecimensinfluenza/en/index.html
9. QIAGEN. Purification of viral RNA. QIAamp Viral RNA Mini Handbook. 2nd ed.; 2005. Available in: http://www.qiagen.com/default.aspx
10. World health Organization. CDC protocol for real time RT-PCR for swine influenza A (H1N1). Geneva. WHO, Apr 2009. Available in: http://www.who.int/csr/resources/publications/swineflu/CDCrealtimeRTPCrprotocol20090428.pdf
11. Coiras MT, Pérez-Breña P, García ML, Casas I. Simultaneous detection of influenza A, B and C viruses, respiratory syncytial virus and adenoviruses in clinical samples by multiplex reserve transcription nested-PCR assay. J Med Virol. 2003;69:32-44.
12. Tenorio Abreu A, Eiros JM, Rodríguez E, Bermejo JF, Domínguez Gil M, Vega T, et al. Influenza surveillance by molecular methods. Rev Esp Quimioter. 2009;22(4):214-20.
13. Coiras MT, Aguilar JC, Garcia ML, Casas I, Pérez-Breña P. Simultaneous detection of fourteen respiratory viruses in clinical specimens by two multiplex reverse transcription nested-PCR assays. J Med Virol. 2004;72:48495.
14. López-Huerta MR, Casas I, Acosta-Herrera B, García ML, Coiras MT, Pérez-Breña P. Two RT-PCR based assays to detect human matapneumovirus in nasopharyngeal aspirates. J Virol Methods. 2005;129:1-7.
15. Dirección Nacional de Estadísticas. Tasas de mortalidad por influenza y neumonía. Cuba: MINSAP; 2008.
16. Oropesa SI, Abreu I, Morier L, Hernández B, González Z, Goyenechea A. Neumonía hemorrágica por Influenza virus: diagnóstico virológico. Rev Cubana Med Trop. 2000;52(1):73-5.
17. Jamieson DJ, Honein MA, Rasmussen SA, Williams JL, Swerdlow DL, Siggerstaff MS, et al. H1N1 2009 influenza virus infection during pregnancy in the USA. Lancet. 2009; 374:451-8.
18. Harris JW. Influenza occurring in pregnant women. JAMA. 1919;72:978-80.
19. Mullooly JP, Barker WH, Nolan TF. Risk of acute respiratory disease among pregnant women during influenza A epidemics. Public Health Rep. 1986;101:205-11.
20. Greenberg M, Jacobziner H, Pakter J, Weisl BAG. Maternal mortality in the epidemic of Asian influenza, New York City, 1957. Am J Obstet Gynecol. 1958;76:897-902.
21. Freeman DW, Barno A. Deaths from Asian influenza associated with pregnancy. Am Obstet Gynecol. 1959;78:1172-5.
22. Creanga AA, Johnson TF, Graitcer SB. Severity of 2009 pandemic influenza A (H1N1) virus infection in pregnant women: New York City, May-June 2009. Obstet Gynecol. 2010;115:717-26.
23. Jamieson DJ, Theiler RN, Rasmussen SA. Emerging infections and pregnancy. Emerg Infect Dis. 2006;12:1638-43.
24. Joshi SR, Shaw AC, Quagliarello VJ. Pandemic Influenza H1N1 2009, Innate Immunity, and the Impact of Immunosenescence on Influenza Vaccine. Yale J Biol Med. 2009;82(4):143-51.
25. Mor G, Cardenas I. The Immune System in Pregnancy: A unique complexity. Am J Reprod Immunol. 2010;63(6):425-33.
26. Rasmussen SA, Jamieson DJ, Bresee JS. Pandemic influenza and pregnant women. Emerg Infect Dis. 2008;14(1):95-100.
27. Khiabanian H, Farrell GM, St George K, Rabadan R. Differences in patient age distribution between influenza A subtypes. PLoS One. 2009;4(8):e6832.
28. Echevarria-Zuno S, Mejia-Arangure JM, Mar-Obeso AJ, Grajales-Muniz C, Robles- Perez E, Gonzalez-Leon M. Infection and death from influenza A H1N1 virus in Mexico: a retrospective analysis. Lancet. 2009;374(9707):2072-9.
29. Hohenthal U, Vainionpää R, Nikoskelainen J, Kotilainen P. The role of rhinoviruses and enteroviruses in community acquired pneumonia in adults.Thorax. 2008;63(7):658-9.
30. Arden KE, Mackay IM. Human rhinoviruses: coming in from the cold. Genome Med. 2009;1(4):44.
31. Savón Valdés Cl, Valdés Ramírez O, Acosta Herrera B, González Muñoz G, Piñón Ramos A, González Báez G, et al. Infección por rinovirus en niños hospitalizados menores de un año. Cuba 2006. Rev Biomed. 2008;19:122-3.
Recibido: 19 de octubre de 2010.
Aprobado: 10 de marzo de 2011.
Suset Oropeza Fernández. Laboratorio Nacional de Referencia de Virus Influenza. Instituto de Medicina Tropical "Pedro Kourí". Novia del Mediodía. Km 6½. Lisa. La Habana, Cuba. Correo electrónico: s.oro@ipk.sld.cu