My SciELO
Services on Demand
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Medicina Tropical
Print version ISSN 0375-0760
Rev Cubana Med Trop vol.66 no.1 Ciudad de la Habana Jan.-Mar. 2014
Diagnóstico molecular de histoplasmosis diseminada en pacientes cubanos con sida
Molecular diagnosis of disseminated histoplasmosis in Cuban AIDS patients
MSc. Ernesto Monroy Vaca, Dr. Gerardo Martínez Machín, Dr. C. Carlos Fernández Andreu, Dra. C. María Teresa Illnait Zaragozí, Dra. Aiyalén García Rodríguez, MSc. Mayda Perurena Lancha, Dr. C. Raúl Díaz Rodríguez
Instituto de Medicina Tropical "Pedro Kourí". La Habana, Cuba.
RESUMEN
Introducción: Histoplasma capsulatum es el agente causal de la histoplasmosis. La forma diseminada de esta micosis de no ser tratada suele ser fatal.
Objetivo: evaluar el empleo de sangre periférica para el diagnóstico molecular de histoplasmosis diseminada y comparar sus resultados con el cultivo y la detección de anticuerpos específicos.
Métodos: se determinó la presencia de ADN de Histoplasma capsulatum a partir de 12 muestras de sangre de pacientes con sida y sospecha clínica de histoplasmosis diseminada, mediante dos sistemas de reacción en cadena de la polimerasa (una simple y otra anidada, que amplifican un fragmento de la región ITS y del gen Hcp100, respectivamente). Se compararon los resultados con el cultivo y la serología.
Resultados: mediante la reacción en cadena de la polimerasa simple y anidada se diagnosticó con histoplasmosis diseminada a los 12 pacientes. Por cultivo resultaron positivos 6 casos (50 %). Mientras que la presencia de anticuerpos anti-Histoplasma capsulatum se determinó en 3 casos (25 %). Los sistemas de reacción en cadena de la polimerasa empleados mostraron elevada sensibilidad, especificidad analítica y diagnóstica en la detección de Histoplasma capsulatum en esta forma clínica.
Conclusiones: los resultados del estudio sugieren que las técnicas de reacción en cadena de la polimerasa aumentan la fiabilidad del diagnóstico de la histoplasmosis cuando se utiliza en combinación con los métodos establecidos. Los sistemas de reacción en cadena de la polimerasa aplicados al diagnóstico de histoplasmosis diseminada en pacientes con sida probaron ser sistemas rápidos, específicos y sensibles.
Palabras clave: diagnóstico molecular, Histoplasma capsulatum, histoplasmosis, reacción en cadena de la polimerasa.
ABSTRACT
Introduction: Histoplasma capsulatum is the causal agent of histoplasmosis. If not treated, the disseminated form of this mycosis is often fatal.
Objective: evaluate the use of peripheral blood for the molecular diagnosis of disseminated histoplasmosis and compare the results with those obtained by culture and detection of specific antibodies.
Methods: the presence of Histoplasma capsulatum DNA was determined from 12 blood samples from patients with AIDS and clinical suspicion of disseminated histoplasmosis, using two polymerase chain reaction systems (one simple and one nested, amplifying a fragment of the ITS region and the Hcp100 gene, respectively). The results were compared with those obtained by culture and serology.
Results: all 12 patients were diagnosed with disseminated histoplasmosis by simple and nested polymerase chain reaction. Culture results were positive in 6 cases (50 %), whereas the presence of anti-Histoplasma capsulatum antibodies was determined in 3 cases (25 %). The polymerase chain reaction systems used showed high sensitivity, and analytical and diagnostic specificity for detection of this clinical form of Histoplasma capsulatum.
Conclusions: results suggest that polymerase chain reaction techniques increase the reliability of histoplasmosis diagnosis when used in combination with established methods. When used for the diagnosis of disseminated histoplasmosis in AIDS patients, polymerase chain reaction systems proved to be fast, specific and sensitive.
Key words: molecular diagnosis, Histoplasma capsulatum, histoplasmosis, polymerase chain reaction.
INTRODUCCIÓN
La histoplasmosis es la micosis endémica más común que afecta a los humanos y es causada por el hongo dimórfico Histoplasma capsulatum. Este hongo habita en suelos contaminados con guano de murciélagos y excretas de aves. La exposición inicial a H. capsulatum es por vía respiratoria y una vez que las microconidas son inhaladas llegan a los alvéolos y se diseminan fácilmente en el interior de los macrófagos por el sistema reticuloendotelial.1,2
La histoplasmosis se considera una enfermedad cosmopolita, se presenta con mayor frecuencia en ciertas áreas del continente americano.1 Las zonas de los ríos Ohio, Mississippi y Missouri en EE. UU. son las de mayor endemicidad.2 Aunque con menor frecuencia, también se han descrito aislamientos del hongo en África, Asia y Europa.3,4
En Cuba, la enfermedad se presenta en forma de brotes en todas las regiones. Se considera una enfermedad ocupacional asociada a algunos grupos de riesgo y ocupa el cuarto lugar en orden de frecuencia entre las micosis oportunistas en las personas infectadas con el virus de inmunodeficiencia humana (VIH).5 Los pacientes inmunocomprometidos desarrollan formas diseminadas de la enfermedad por infección reciente o reactivación de una infección previa; de no ser tratadas, estas formas tienen un mal pronóstico, generalmente fatal. El cuadro clínico de la histoplasmosis diseminada se asemeja al producido por infecciones de otros orígenes, lo que dificulta su diagnóstico presuntivo.6
En el laboratorio, el diagnóstico convencional de la histoplasmosis se basa en el aislamiento e identificación de su agente causal, lo que puede demorar hasta 8 semanas, debido al lento crecimiento de H. capsulatum en medios de cultivo, además, esta técnica presenta baja sensibilidad.7 La determinación de anticuerpos anti-H. capsulatum en suero presenta baja sensibilidad, porque dependerá del estado inmunológico del hospedero y del tiempo transcurrido desde el primo-contacto.6 La detección de antígeno polisacárido en suero u orina es útil en el diagnóstico de pacientes inmunocomprometidos.2,6 No obstante, esta prueba resulta de difícil adquisición y brinda resultados falsos-positivos debido a otras micosis.4,8 En Cuba, se realiza el cultivo y la detección cualitativa de anticuerpos anti-H. capsulatum.
En los últimos años se han diseñado métodos moleculares basados en la amplificación de ADN tal como la reacción en cadena de la polimerasa (PCR: polymerase chain reaction) dirigidas a diferentes genes específicos de H. capsulatum, estos han sido validados en diferentes especímenes clínicos.9-12 La PCR es una técnica sensible y rápida, útil en la detección, caracterización y diagnóstico de H. capsulatum.11,12
La implementación de dos sistemas de PCR en el Laboratorio de Micología (LM) del Instituto de Medicina Tropical «Pedro Kourí» (IPK) permitirá la instauración del diagnóstico y terapéutica oportuna y específica. Esto contribuirá directamente a la calidad de vida de los pacientes y al mejor conocimiento de esta micosis en Cuba.
El objetivo de este trabajo era evaluar el empleo de sangre periférica de pacientes con sida para el diagnóstico molecular de la histoplasmosis diseminada y comparar sus resultados con los obtenidos mediante cultivo y la detección de anticuerpos anti-H. capsulatum.
MÉTODOS
Se realizó un estudio de corte transversal a partir de muestras clínicas provenientes de 12 pacientes de sida con diagnóstico clínico de histoplasmosis diseminada,1,2 ingresados en el Hospital del IPK entre abril de 2011 y octubre de 2012. Los especímenes clínicos fueron enviados al Laboratorio de Micología para su estudio micológico.
Especímenes clínicos
Se recibieron muestras de sangre periférica, suero, escamas de piel y fragmentos de tejidos obtenidos por biopsia cuando fue posible, estas se mantuvieron según la muestra a 4 y -20 °C hasta su procesamiento (Procedimientos normativos operacionales-PNO: manejo y conservación de muestras, LM-IPK).
Diagnóstico convencional
Las muestras de sangre y los fragmentos de tejido se sembraron por duplicado en tubos con agar Sabouraud dextrosa (ASD), ASD-cloranfenicol (ASC) y agar infusión de cerebro y corazón; las muestras de escamas de piel se inocularon en ASD y ASC-cicloheximida, los cuales se incubaron a 28 y 37 ºC según requerimientos hasta 8 semanas. La identificación microscópica de los cultivos se hizo mediante la observación de las características culturales y de preparaciones con lactofenol azul de algodón (PNO: manipulación e identificación de muestras, LM-IPK).
Diagnóstico serológico
Para determinar anticuerpos anti-H. capsulatum en las muestras de suero se utilizó un sistema comercial de inmunodifusión doble (IDD) (ID fungal antibody system; immuno-mycologics, Inc, Norman, EE. UU.).
Condiciones de bioseguridad
Todos los trabajos se realizaron en un laboratorio de bioseguridad para patógenos del grupo de riesgo 2, conforme a las normas cubanas establecidas (Resolución No. 38/2006, CITMA).
Diagnóstico molecular
Reacción en cadena de la polimerasa anidada
En la PCR anidada se utilizaron los cebadores y el protocolo diseñados por Bialek y otros,9 que amplifica un fragmento de 210 pb del gen Hcp100. Para la primera amplificación se utilizaron los cebadores HcI y HcII, para la segunda ronda de amplificación los oligonucleotidos HcIII y HcIV (tabla 1). Ambas mezclas de reacción (50 µL volumen final) se prepararon en Tris/HCl (pH 8,3) 10 mM, KCl 50 mM, MgCl2 2 mM, dNTP 200 µM, iniciadores 0,5 µM, 1,5 U de HotStarTaq ADN polimerasa (Qiagen, Hilden, Alemania) y 5 µL de ADN molde. Para la segunda ronda de PCR se partió de 2,5 uL del producto amplificado de la primera ronda. Como programa de amplificación para ambas rondas se utilizaron: 95 °C por 10 min, 35 ciclos de 1 min a 94 °C, 1 min a 65 °C y 1 min a 72 °C, con una extensión final de 5 min a 72 °C.
Reacción en cadena de la polimerasa simple
En la PCR simple se siguió la metodología descrita por Buitrago y otros,13 la cual tiene como diana un fragmento de 182 pb de la región multicopia ITS1-5,8S-ITS2 del ARNr. Esta región se amplificó utilizando los cebadores Hcits1-1 y Hcits1-2 (tabla 1). Las mezclas de reacción (50 µL) se prepararon en Tris/HCl (pH 8,3) 10 mM, KCl 50 mM, MgCl2 1,75 mM, dNTP 200 µM, iniciadores 0,5 µM, 1,5 U de HotStarTaq ADN polimerasa (Qiagen, Hilden, Alemania) y 5 µL de ADN molde. Como programa de amplificación se utilizó: 95 °C por 10 min, 45 ciclos de 1 min a 94 °C, 1 min a 60 °C y 1 min a 72 °C, con una extensión final de 5 min a 72 °C.
Para ambos sistemas de PCR se utilizaron como control positivo ADN de la cepa H. capsulatum ATCC 26029 (donado por el Broad Institute, EE. UU.) y como control negativo agua MiliQ (Sigma, EE. UU.).
Determinación de límite de detección y especificidad analítica de los sistemas de reacción en cadena de la polimerasa
A partir del ADN genómico (ADNg) de la cepa H. capsulatum ATCC 26029 se realizaron diluciones seriadas en base 10 en una solución de ADN control negativo (desde 100 ng/µL hasta 0,1 fg/µL). Se añadieron 3 réplicas de cada una de las diluciones del ADNg en viales que contenían 45 µL de las mezclas de PCR simple y anidada, descritas antes. Los parámetros del ciclaje fueron los mismos previamente descritos para ambas PCR. Una vez determinada la concentración de la última dilución en la que el sistema es capaz de detectar el ADN diana en el total de las réplicas, se calculó el valor en genomas equivalentes por microlitros (geq/mL).14 Asimismo, para evaluar la especificidad analítica de las PCR ensayadas se utilizó ADN de 25 especies de hongos con taxonomía afín o que están comúnmente involucrados en infecciones sistémicas, y 3 especies de bacterias que pueden compartir hábitat común (tabla 2). Además, se utilizaron 25 muestras de ADN de aislamientos de H. capsulatum de origen clínico y ambiental. Los ácidos nucleicos utilizados pertenecen a la colección de ADN del LM-IPK y se conservaron en una solución de TE (10 mM TRIS, 1 mM EDTA, pH 8,0) a -20 °C hasta su utilización.
Tabla 2. Especies bacterianas y fúngicas utilizadas para verificar la especificidad
de los cebadores en este estudio
| Microorganismos | Reacción en cadena de la polimerasa | |
| Anidada | Simple | |
| Candida albicans | - | - |
| Candida krusei | - | - |
| Candida parapsilopsis | - | - |
| Cryptococcus neoformans | - | - |
| Cryptococcus gatti | - | - |
| Rhodotorula sp. | - | - |
| Geotrichum candidum | - | - |
| Cladophialophora bantiana | - | - |
| Cladophialophora arxii | - | - |
| Aspergillus fumigatus | - | - |
| Aspergillus terreus | - | - |
| Aspergillus nidulans | - | - |
| Aspergillus fermentans | - | - |
| Aspergillus versicolor | - | - |
| Cladosporium herbarum | - | - |
| Curvularia lunata | - | - |
| Penicillium deumbens | - | - |
| Acremonium alabamense | - | - |
| Alternaria sp. | - | - |
| Bipolaris australiensis | - | - |
| Bipolaris spicifera | - | - |
| Trichophyton rubrum | - | - |
| Fonsecae pedrosoi | - | - |
| Rhizopus oryzae | - | - |
| Mycobacterium avium | - | - |
| Mycobacterium chelonei | - | - |
| Pseudomonas aeruginosa | - | - |
| Blastomyces dermatitidis | - | - |
| Histoplasma capsulatum (25 aislamientos) | + | + |
Todos los productos amplificados se sometieron a una electroforesis en gel de agarosa 2 %, siguiendo protocolos establecidos.15 La corrida electroforética se ajustó a 90 V por 60 min. Los resultados fueron documentados (UVIsave D-55/20M, UVItec, Inglaterra) para su posterior análisis.
Detección de Histoplasma capsulatum mediante reacción en cadena de la polimerasa en muestras clínicas
El ADN de las muestras de sangre fue extraído utilizando el estuche comercial high pure pcr template preparation kit (Roche, Alemania). Se utilizaron 5 µL de solución de ADN para cada PCR descrita antes. Asimismo, para evidenciar que no ocurrió inhibición de los sistemas de PCR en los casos que el resultado fue negativo, se efectuó un ensayo de PCR simple empleando los cebadores Bac1 y Bac2 (tabla 1), que amplifican un fragmento de 202 pb del gen humano â-actina.16 La mezcla de reacción (50 µL volumen final) se prepararon en Tris/HCl (pH 8.3) 10 mM, KCl 50 mM, MgCl2 1,5 mM, dNTP 200 µM, cebadores 0,5 µM, 1,5 U de HotStarTaq ADN polimerasa y 5 µL de ADN molde. Como perfil de amplificación se utilizaron: 94 °C por 10 min, 35 ciclos de 30 s a 94 °C, 30 s a 51 °C y 30 s a 72 °C, con una extensión final de 5 min a 72 °C. Cada vez que se procesó una muestra de sangre se incluyó un control negativo (sangre periférica libre de levaduras) y un control positivo (sangre periférica con 10 ng de ADN de H. capsulatum).
Análisis de los resultados
Los datos y resultados de laboratorio de los pacientes se introdujeron en una planilla elaborada en Excel y se realizó un análisis descriptivo de los resultados. Se calculó la sensibilidad y la especificidad diagnóstica de los métodos evaluados con relación al diagnóstico clínico.
RESULTADOS
En la tabla 3 se presentan las muestras estudiadas, los métodos de diagnóstico utilizados y el resultado obtenido en cada análisis por paciente.
Tabla 3. Resultados de los estudios micológicos realizados para cada paciente
| Paciente | IDD | Material clínico | Cultivo | Reacción en cadena de la polimerasa | |
| Anidada | Simple | ||||
| 1 | - | Sangre periférica Escamas de piel | + + | + | + |
| NR | NR | ||||
| 2 | - | Sangre periférica Biopsia de piel | + + | + | + |
| NR | NR | ||||
| 3 | - | Sangre periférica | - | + | + |
| 4 | + | Sangre periférica | + | + | + |
| 5 | - | Sangre periférica | - | + | + |
| 6 | - | Sangre periférica | + | + | + |
| 7 | + | Sangre periférica | - | + | + |
| 8 | - | Sangre periférica Biopsia de piel | + + | + | + |
| NR | NR | ||||
| 9 | - | Sangre periférica | - | + | + |
| 10 | - | Sangre periférica | - | + | + |
| 11 | - | Sangre periférica | + | + | + |
| 12 | + | Sangre periférica | - | + | + |
IDD: anticuerpos anti-Histoplasma capsulatum detectados por inmunodifusión doble
en muestras de suero;-: negativo; +: positivo; NR: no realizado.
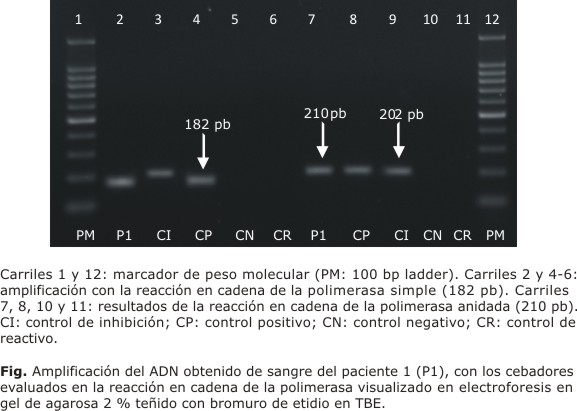
Tanto las reacciones de PCR simple como anidada resultaron positivas en el total de las muestras de sangre analizadas (Fig.), en las que se observó una banda correspondiente a 182 y 210 pb,9,13 respectivamente. Similares resultados se obtuvieron cuando se añadió ADN de los 25 aislamientos de H. capstulatum. Asimismo, las PCR utilizadas para este estudio resultaron negativas cuando se les añadió ADN de hongos diferentes de H. capsulatum y bacterias (tabla 2). Estos resultados permiten determinar que las PCR utilizadas tienen una sensibilidad y especificidad analítica de 100 %.
El límite de detección para la PCR simple y anidada resultó de 10 fg (15 geq/mL) y 1 fg (5 geq/mL), respectivamente.
La sensibilidad diagnóstica se estableció sobre la base del resultado de las PCR, a partir de sangre de los 12 pacientes con sospecha clínica de histoplasmosis diseminada, lo que resultó en una sensibilidad diagnóstica de 100 %; este valor es importante si se considera que los pacientes analizados son inmunocomprometidos, en los cuales esta metodología tiene mayor aplicación.
Solo en 6 pacientes (50 %) se logró aislar H. capsulatum a partir de sangre utilizando medios de cultivo micológicos, los cuales mostraron características culturales y microscópicas propias de este patógeno. En 3 pacientes se recogieron biopsias o escamas de piel, y se obtuvo crecimiento característico del patógeno. La prueba de IDD resultó positiva en 3 casos (25 %).
DISCUSIÓN
La histoplasmosis diseminada es una forma rara de presentación de la enfermedad y generalmente se observa en pacientes con compromiso del sistema inmunitario; en Cuba ocupa el cuarto lugar en orden de frecuencia entre las micosis que afectan a los pacientes seropositivos al VIH, después de la neumocistosis, candidiasis y criptococosis. En estos individuos las infecciones son a menudo severas, de rápida progresión y difícil diagnóstico; puede ser fatal si no se trata a tiempo.17 En este grupo de pacientes la histoplasmosis diseminada progresiva se presenta en 4,2 % de los casos en Cuba; este valor dependerá de la región geográfica.18
El diagnóstico de la histoplasmosis se basa en el análisis integral de las manifestaciones clínicas, los estudios radiológicos y datos epidemiológicos de los pacientes. Su confirmación se efectúa a través de exámenes de laboratorio,1 entre ellos se pueden citar el examen directo coloreado de la muestra, cultivo e identificación del agente, las pruebas serológicas y moleculares.19 La positividad de cada prueba variará con la forma clínica, extensión y severidad de la infección.20
Las muestras clínicas útiles para el diagnóstico de la histoplasmosis pueden ser muy variadas como aspirado bronquial, lavado broncoalveolar, esputo, orina, sangre, lesiones en piel y mucosas, biopsias de ganglios y aspirados de médula ósea.19,21 La sangre periférica constituye la muestra ideal en los pacientes con sospecha de histoplasmosis diseminada, debido a su fácil obtención y transporte. Este tipo de muestra es útil para obtener crecimiento fúngico en medios de cultivo y la realización de pruebas de PCR específicas para su determinación.
El diagnóstico convencional (cultivo e identificación de H. capsulatum) puede demorar más de 4 semanas, lo cual disminuye su valor práctico en la determinación de esta entidad. Esos métodos tienen una baja sensibilidad y requieren de infraestructura y equipamiento adecuado para manipular los cultivos de H. capsulatum en condiciones de bioseguridad.11 Según Wheat7 el cultivo produce de 20 a 50 % de falsos negativos según las formas clínicas. En este trabajo se logró cultivo compatible con H. capsulatum en 6 pacientes (50 %), a partir de 4 semanas como promedio de incubación, lo que sugiere una alta concentración de este hongo en las muestras analizadas.
Existen también métodos alternativos como la detección de antígenos y anticuerpos. En los casos de pacientes infectados con VIH e histoplasmosis diseminada, la prueba que más contribuye al diagnóstico es la detección de antígenos (96 % de sensibilidad y 85 % de especificidad) de H. capsulatum a partir de muestras de orina o suero.7 Desafortunadamente, este método tiene elevado costo y presenta reacciones inespecíficas.4 La detección de anticuerpos mediante IDD en pacientes con sida presenta baja sensibilidad debido al deterioro del sistema inmunitario de estos,14 lo que podría estar influyendo en los resultados de esta prueba (25 %) en las muestras analizadas en este estudio. Sin embargo, esta técnica es útil en personas inmunocompetentes que viven en zonas no endémicas de H. capsulatum, en las cuales esta prueba tiene un valor diagnóstico más elevado.18 Muestra de ello es que en 2012 se reportó el diagnóstico de 3 casos de histoplasmosis pulmonar basado en los resultados de la IDD y los antecedentes epidemiológicos en un grupo de franceses que visitaron cuevas cubanas.21
Dentro de las pruebas utilizadas para el diagnóstico rápido de la histoplasmosis se encuentra la descrita por Bialek y otros9 la cual utiliza una PCR anidada que amplifica un fragmento del gen unicopia Hcp100. La misma se ha utilizado ampliamente en el diagnóstico rápido de esta micosis a partir de disímiles muestras clínicas. La adaptación de la PCR anidada a las condiciones del LM-IPK demostró tener alta sensibilidad y especificidad analítica y diagnóstica. Estos resultados coinciden con lo publicado por Mauboun y otros,10 así como Toranzo y otros,11 quienes informaron 100 % y 89 %, respectivamente, de sensibilidad al analizar diversos materiales clínicos.
Utilizando la PCR anidada se determinó un límite de detección igual a 5 geq/mL, similar al valor determinado por sus autores.9 Esta PCR resulta más sensible que la PCR simple, porque cuenta con una doble ronda de amplificación que aumenta el límite de detección y especificidad. No obstante, la PCR anidada es una fuente potencial de contaminación, debido a la generación de amplicones en la apertura de los tubos entre las rondas amplificadas, lo que podría favorecer la aparición de resultados falsos-positivos.19 En este sentido, la PCR simple utilizada en el diagnóstico de la histoplasmosis que amplifica un fragmento de la región ITS disminuye la probabilidad de contaminación por manipulación y su empleo se justifica porque este locus posee varias copias en el genoma del patógeno (mayor o igual que 100 copias), lo que aumenta la sensibilidad de la técnica, además de ser un locus informativo que brinda la posibilidad de caracterizar al microorganismo.22 Utilizando esta metodología se obtuvo amplificación efectiva en los 12 casos analizados (100 %), con valores idénticos de sensibilidad, especificidad analítica y diagnóstica para un límite de detección igual a 15 geq/mL, la cual se muestra útil como técnica diagnóstica rápida de la histoplasmosis diseminada. No se observaron amplificaciones inespecíficas cuando se ensayaron las PCR antes mencionadas con el ADN extraído de especies de hongos diferentes de H. capsulatum y bacterias, lo que determina 100 % de especificidad.
A pesar del gran impacto de las técnicas moleculares en el campo de la micología médica, estas no eliminan el empleo rutinario de las técnicas clásicas, sino que las complementan al mejorar la sensibilidad general del diagnóstico y aumentar su rapidez, sobre todo en aquellos pacientes que desarrollan las formas graves de la enfermedad.20
Con este trabajo quedaron implementados 2 protocolos de PCR en el LM-IPK, lo que permite contar con herramientas sensibles y específicas que se encuentran disponibles para el diagnóstico de la histoplasmosis diseminada en el sistema nacional de salud, y de esta forma contribuye a un aumento de la calidad de vida de los pacientes gracias a un diagnóstico rápido y oportuno.
Los resultados en este trabajo permiten sugerir la utilidad de la PCR como herramienta complementaria de los métodos microbiológicos convencionales, aplicados en el diagnóstico de la histoplasmosis diseminada en pacientes con sida. Este trabajo constituye el primer reporte en Cuba de diagnóstico molecular de histoplasmosis diseminada en pacientes con sida.
AGRADECIMIENTOS
Al Dr. C. Yaxsier de Armas y al MSc. Brian Mondeja por la revisión crítica de este manuscrito.
REFERENCIAS BIBLIOGRÁFICAS
1. Kauffman CA. Histoplasmosis. Clin Chest Med. 2009;30:217-25.
2. Wheat LJ. Histoplasmosis: a review for clinicians from non-endemic areas. Mycoses. 2006;49:274-82.
3. Ashbee HR, Evans EG, Viviani MA, Dupont B, Chryssanthou E, Surmont I, et al. The ECMM Working Group on Histoplasmosis. Histoplasmosis in Europe; report on an epidemiological survey from the European Confederation of Medical Mycology Working Group. Med Mycol. 2008;46:57-65.
4. Buitrago MJ, Gómez-López A, Monzón A, Rodríguez-Tudela JL, Cuenca-Estrella M. Evaluación de una técnica de PCR cuantitativa para el diagnóstico clínico de la histoplasmosis importada. Enferm Infecc Microbiol Clin. 2007;25:16-22.
5. Fernández-Andreu C, Illnait-Zaragozí M, Martínez-Machín G, Perurena-Lancha M, Monroy-Vaca E. Una actualización acerca de histoplasmosis. Rev Cubana Med Trop. 2011;63:188-205.
6. Negroni R. Micosis sistémicas tropicales asociadas al sida. Enf Emerg. 2003;5:27-40.
7. Wheat LJ. Current diagnosis of histoplasmosis. Trends Microbiol. 2003;11:488-94.
8. Ohno H, Tanabe K, Umeyama T, Kaneko Y, Yamagoe S, Miyazaki Y. Application of nested PCR for diagnosis of histoplasmosis. J Infect Chemother. 2013;19:999-1003.
9. Bialek R, Feucht A, Aepinus C, Just-Nubling G, Robertson VJ, Knobloch J. Evaluation of two nested PCR assays for detection of Histoplasma capsulatum DNA in human tissue. J Clin Microbiol. 2002;40:1644-7.
10. Maubon D, Simon S, Aznar C. Histoplasmosis diagnosis using a polymerase chain reaction method. Application on human samples in French Guiana, South America. Diag Microbiol Infect Dis. 2007;58:441-4.
11. Toranzo AI, Tiraboschi IN, Fernández N, Ibarra-Camou B, Rivas MC, Lee W, et al. Diagnóstico molecular de histoplasmosis humana en muestras de sangre entera. Rev Argent Microbiol. 2009;41:20-6.
12. Muñoz C, Gómez BL, Tobón A, Arango K, Restrepo A, Correa MM, et al. Validation and clinical application of a molecular method for identification of Histoplasma capsulatum in human specimens in Colombia, South America. Clin Vaccine Immunol. 2010;17:62-7.
13. Buitrago MJ, Berenguer J, Mellado E, Rodríguez-Tudela JL, Cuenca-Estrella M. Detection of imported histoplasmosis in serum of HIV-infected patients using a real-time PCR-based assay. Eur J Clin Microbiol Infect Dis. 2006;25:665-8.
14. Applied Biosystems. Creating standard curves with genomic DNA or plasmid DNA templates for use in quantitative PCR; 2003. p. 1-9.
15. Sambrook KJ, Fritsch EF, Maniatis T. Molecular cloning. A laboratory manual. New York: Cold Spring Harbour Laboratory; 1989.
16. Ghossein RA, Ross DG, Salomon RN, Rabson AR. A search for mycobacterial DNA in sarcoidosis using the polymerase chain reaction. Am J Clin Pathol. 1994;101:733-7.
17. Arteaga E, Capó V, Fernández ML. Micosis oportunistas invasivas en el sida. Un estudio de 211 autopsias. Rev Ibero Micol. 1998;15:33-5.
18. Fernández CM. Histoplasma capsulatum. En: Llops HA, Valdés Dapena Vivanco MM, Zuazo SJ, editores. Microbiología y Parasitología Médica. La Habana: Editorial Ciencias Médicas; 2001. p. 509-15.
19. Simon S, Veron V, Boukhari R, Blanchet D, Aznar C. Detection of Histoplasma capsulatum DNA in human samples by real-time polymerase chain reaction. Diagn Microbiol Infect Dis. 2010;66:268-73.
20. Frías de León MG, Taylor ML, Hernández-Ramírez A, Reyes-Montes R. Utilidad de las técnicas moleculares en el diagnóstico de la histoplasmosis. Rev Mex Micol. 2007;25:83-90.
21. Senechal A, Ferry T, Boibieux A, Brion JP, Epaulard O, Chidiac C, et al. Imported pulmonary histoplasmosis in three French cavers after a trip to Cuba. J Travel Med. 2012;19:64-5.
22. Lau A, Sorrell TC, Chen S, Stanley K, Iredell J, Halliday C. Multiplex tandem PCR: a novel platform for rapid detection and identification of fungal pathogens from blood culture specimens. J Clin Microbiol. 2008;46:3021-7.
Recibido: 30 de septiembre de 2013.
Aprobado: 21 de octubre de 2013.
Gerardo Martínez Machín. Instituto de Medicina Tropical "Pedro Kourí". Autopista Novia del Mediodía km 6½. Marianao 13. La Habana, Cuba. CP 601. Teléf.: (53-7) 2553524. Correo electrónico: gerardo@ipk.sld.cu