Introducción
El diagnóstico de infecciones virales ha evolucionado desde los métodos convencionales, en los que se necesita conservar la capacidad infectiva viral, hasta los métodos moleculares. Estos últimos tienen una sensibilidad y especificidad elevada, pero dependen de la estabilidad del material genético (ARN o ADN). Con el propósito de reducir los riesgos de contagio por virus u otros microorganismos, varias compañías biomédicas están desarrollando medios de colección y transporte de muestras clínicas seguros y con propiedades desnaturalizantes.1
Existen métodos clásicos para inactivar microorganismos como la luz ultravioleta, radiaciones gamma, detergentes y el calor, que demostraron su efectividad como agentes desnaturalizantes de virus desnudos y envueltos, como los Coronavirus. Esto permite disminuir su nivel de contención de bioseguridad, no obstante, la inactivación resultante debe ser segura y certificada por técnicas de laboratorio. En el caso de los medios de colecta y transporte de muestra con capacidad de desnaturalizar virus, se requiere que estos no afecten la integridad del ácido nucleico viral y el desempeño de la reacción en cadena de la polimerasa (PCR). Con este objetivo, se emplean el TRIzol, detergentes, soluciones desnaturalizantes como el AVL y el RTL; estos últimos presentes en los estuches de extracción de ARN (QIAGEN).2 Sin embargo, la eficacia de estas soluciones depende de la concentración del patógeno, de los agentes inactivantes y del tiempo de contacto.
Desde que comenzó la pandemia de la enfermedad por Coronavirus 2019 (COVID-19), el personal de los laboratorios encargados del diagnóstico molecular del Coronavirus 2, causante del síndrome respiratorio agudo severo (SARS-CoV-2), ha manipulado y procesado gran número de muestras clínicas respiratorias altamente infecciosas. Con la finalidad de disminuir los riesgos en el transporte, manipulación y procesamiento de las muestras, así como garantizar la detección oportuna del SARS-CoV-2, el Centro Nacional de Biopreparados (BIOCEN, Cuba) desarrolló un medio de transporte viral inactivante denominado TAN (acrónimo de transporte de ácidos nucleicos). Este se compone por hidrocloruro de guanidina (GHCl), presevantes del ARN, agentes quelantes y reductores. El GHCl como agente caotrópico inhibe la acción de las ribonucleasas, lo que contribuye a estabilizar el ARN viral.
Según las recomendaciones de la Organización Mundial de la Salud (OMS), las muestras clínicas potencialmente infectadas con SARS-CoV-2 deben ser manipuladas y procesadas en laboratorios con nivel 2 de bioseguridad (BSL2), mientras que los ensayos que requieran multiplicación viral deben realizarse en BSL3.3 En ambos casos, el personal encargado de la realización de estos debe utilizar equipo de protección personal, guantes y máscara de protección facial.
El propósito de este trabajo fue demostrar la capacidad del medio TAN de inactivar la infectividad del virus Zika (VZk), como modelo de virus envuelto, y que a su vez se trabaja en BSL2 para cultivo celular. Posteriormente, se evaluó el desempeño clínico de este medio, con muestras clínicas de exudado nasofaríngeo (ENF) colectadas para el diagnóstico del SARS-CoV-2.
Métodos
Diseño del estudio
Se realizó un estudio analítico de tipo experimental, para evaluar la capacidad del medio TAN de inactivar la infectividad del VZk. El GHCl, agente caotrópico y desnaturalizante del medio se empleó a una concentración de 2,5 M.
Células y virus Zika
La cepa viral que se utilizó fue VZk 9661 orina/2016 aislada de un paciente cubano, con un título viral de 4,66 × 106 unidades formadoras de placa por mililitro (ufp/mL), y dos pases en la línea celular Vero, proveniente de riñón de mono verde africano (ATCC, CCL-81). Esta cepa fue aislada y propagada en el Laboratorio de Arbovirus, del Instituto de Medicina Tropical Pedro Kourí (IPK). Para esta investigación se utilizó la línea celular Vero, crecidas a 37 °C en medio esencial mínimo (MEM), con 100 g/mL de penicilina y estreptomicina; suplementado con 10 % de suero fetal bovino (SFB). Como medio de mantenimiento se empleó el mismo medio de crecimiento con un 2 % de SFB.
Siguiendo el protocolo descrito por Blow y otros, los inóculos para evaluar la inactivación de la infectividad viral se prepararon a partir de una relación medio TAN y VZk inicial de 4:1. Esta suspensión fue homogenizada por vórtex y constituyó el inóculo puro; a partir de este se prepararon diluciones seriadas en base 10 desde 10-1 hasta 10-4 en medio de mantenimiento de células.4 Después de obtener la relación inicial medio TAN:VZk (4:1), se evaluaron tres tiempos de contacto en minutos (2 min, 15 min y 30 min), antes de hacer las diluciones referidas e infectar las células. Este mismo procedimiento se empleó para los inóculos que fueron utilizados como controles del ensayo: medio TAN: medio de mantenimiento (control de citotoxicidad) y medio de mantenimiento: VZk (control de multiplicación viral), además, del control de células Vero (células + medio de mantenimiento). Las células fueron infectadas con VZk a una multiplicidad viral de 0,001, cada inóculo se dispensó por duplicado y el experimento se repitió en dos ocasiones.
Para el estudio se emplearon placas de 24 pocillos (Falcon®), que contenían 105células/pozos, las que se incubaron en 5 % de CO2 a 37 ºC por 7 días. Para añadir los inóculos, se procedió a retirar el medio de crecimiento y se añadió 0,1 mL del inóculo correspondiente. Este se mantuvo en contacto con la monocapa celular durante 60 min., y luego se completó hasta 1 mL con medio de mantenimiento. Las células fueron observadas diariamente en un microscopio óptico Olympus, para evaluar la viabilidad celular y el efecto citopático (ECP). Al culminar el periodo de propagación, se calculó el porcentaje de células vivas para cada inóculo tiñendo las células con tripán azul, y realizando el conteo en cámara de Neubauer. La visualización del ECP que provoca el VZk durante la multiplicación en las células Vero permitió estimar la replicación viral, y fue verificada mediante la transcripción reversa-PCR en tiempo real (RT-PCR). Con los valores que se obtuvieron del ciclo umbral (Ct), se realizó la detección cualitativa del VZk y la semicuantificación del virus, ya que su valor es inversamente proporcional a la cantidad de ARN presente en la muestra.
RT-PCR para la detección del virus Zika
Al concluir el tiempo de multiplicación viral, se colectó el sobrenadante y se centrifugó a 1500 rpm por 10 min a temperatura ambiente (TA), para recuperar el botón de células desprendidas. Luego, estas fueron lavadas con solución fosfato salina (PBS) 1X, y resuspendidas en 0,2 mL de esta misma solución. La extracción del ARN viral se realizó a partir del sobrenadante y de las células colectadas, con el estuche QIamp viral RNA mini kit (QIAGEN, Alemania), siguiendo las instrucciones del fabricante. Luego se procedió con la RT-PCR con cebadores y sonda específica para el VZk diseñados por Lanciotti y otros, los que amplificaron un fragmento del gen de la proteína E del genoma viral.5 La corrida del RT-PCR se realizó en el equipo Applied Biosystem 7500 y en cada ensayo se incluyeron controles negativos y positivos, para fiscalizar el desempeño de la técnica. Las muestras fueron consideradas positivas cuando se obtuvo una curva sigmoidal con valores de Ct < 35. La RT-PCR se utilizó para demostrar la inactivación de la infectividad del VZk, al comparar los valores del Ct del inóculo medio TAN: VZk, con los controles del ensayo.
Evaluación del desempeño clínico del medio TAN en muestras de ENF colectadas para diagnóstico molecular de SARS-CoV-2
Se seleccionaron al azar 19 ENF positivos y 11 negativos a la RT-PCR para SARS-CoV-2, colectados en el medio comercial CITOSWAB® VTM (China), en marzo de 2021. El medio TAN se empleó para diluir estos ENF en una proporción de 4:1, por lo que se definieron dos grupos ENF sin tratar (ENF1 en CITOSWAB® VTM) y tratados con medio TAN (ENF2). En ambos grupos las muestras se mantuvieron a TA del laboratorio (22-24 ºC), durante 24 horas. Posteriormente, se realizó la extracción del ARN viral de ambos grupos de ENF, con el estuche de QIamp viral RNA mini kit. La pureza e integridad del ARN extraído se evaluó comparando la relación de la absorbancia 260/280 (A260/280) medida en el espectrofotómetro QIAxpert, entre los ENF1 y 2.
Los valores A260/280 > 1,7 se correspondieron con muestra de ARN de pureza aceptable, mientras que una razón A260/280 < 1,7 indicaba contaminación con compuestos aromáticos.6 La RT-PCR para el SARS-CoV-2 se realizó con el estuche comercial STAT-NAT® COVID-19 MULTI (SENTINEL, Italia), que detecta simultáneamente genes del marco abierto de lectura 1b (ORF1b) y de la ARN polimerasa ARN dependiente (RpRd), y para su validez se detectó como control interno el gen constitutivo humano de la RNAse P. Para la RT-PCR se empleó el termociclador de Roche LightCycler 480-II/96 y en cada ensayo se incluyeron controles negativos y positivos, como controles de calidad. Las muestras fueron consideradas positivas cuando se obtuvo una curva sigmoidal, con valores de Ct < 37.
Análisis estadístico
Se realizó un estudio experimental, y se emplearon estadígrafos descriptivos como media y desviación estándar (DS). La viabilidad celular se clasificó como conservada, cuando se observó que más del 50 % de las células cultivadas estaban viables al concluir el periodo de observación, y no conservada cuando no se cumplió esta condición. Para estimar las diferencias entre las medias se empleó la prueba T de students pareada y los valores de p < 0,05 fueron considerados estadísticamente significativos. Además, como indicadores del desempeño del medio TAN se evaluó la sensibilidad, especificidad clínica y concordancia (índice Kappa) para el diagnóstico molecular del SARS-CoV-2.
Los resultados se presentaron en porcentajes e intervalos de confianza al 95 %, tomando como referencia los resultados de la RT-PCR realizada a partir de la extracción de ARN de los ENF1. Asimismo, se empleó el coeficiente de correlación de Pearson, para evaluar la correlación entre los Ct obtenidos por ENF1 y 2, pareado por muestra. El paquete estadístico GraphPad Prism 6 se utilizó para realizar los análisis.
Resultados
Inicialmente, al evaluar la viabilidad de las células Vero se detectó la muerte celular masiva desde los primeros días (2-3 días) de propagación, al comparar con el control de células, en las células que se pusieron en contacto con el medio TAN: VZk y medio TAN: medio de mantenimiento (relación 4:1, sin diluir). Sin embargo, la viabilidad celular observada a través del microscopio óptico, se conservó a partir de las diluciones empleadas hasta culminar el periodo de observación. Por ende, la reducción de la infectividad viral se evaluó a partir de la dilución 10-1 del inóculo medio TAN: VZk.
En todas las diluciones del medio en estudio que contenían virus, así como en el control de propagación del VZk se observó ECP, el cual estuvo caracterizado por cambios de la morfología celular (a redondeada) y disrupción de la monocapa en grado variable. Este efecto incrementó notablemente su expresión desde la dilución 10-1 del medio TAN: VZk, hasta la dilución 10-4. En la dilución 10-1 del inóculo medio TAN: VZk se observó desprendimiento celular de grado variable al finalizar el periodo de observación, similar al apreciado en el control de medio TAN libre de VZk (medio TAN: medio de mantenimiento), de esta misma dilución.
Según la RT-PCR para el VZk, se obtuvieron resultados no detectables en el sobrenadante y en las células infectadas con el inóculo medio TAN: VZk sin diluir y a la dilución 10-1, independientemente del tiempo de contacto medio TAN: virus. Mientras que en las células inoculadas con medio TAN: VZk a diluciones de 10-2 a 10-4 se detectó el fragmento del genoma viral. Específicamente, en las diluciones 10-3 a 10-4 del medio TAN: VZk, los valores de Ct fueron muy cercanos al control de multiplicación del VZk del ensayo (Tablas 1 y 2). Los Ct tuvieron un comportamiento similar en el sobrenadante y en las células recuperadas, según las diluciones evaluadas y los tiempos de contacto medio TAN: virus.
Tabla 1 - Comportamiento de la viabilidad celular e inactivación de la infectividad del virus Zika con el medio TAN, expresado en valores de Ct
|
|
|
10-3 |
Dilución 10-4 |
|||
|---|---|---|---|---|---|---|
|
Ct Células/sobrenadante (viabilidad celular por tripán azul) | ||||||
| Control de propagación de VZk |
24,05/23,15 (C) |
NA | NA | NA | NA | NA |
| Control de células Vero |
ND/ND (C) |
NA | NA | NA | NA | NA |
| Medio TAN: VZk | NA | ND/ND (NC) |
ND/ND (C) |
30,39/32,00 (C) | 24,15/24,90 (C) | 23,15/23,90 (C) |
Leyenda: VZk: virus Zika; Ct: ciclo umbral; NA: no aplica; ND: no detectable; NC: no conservada; C: conservada.
Tabla2 - Inactivación de la infectividad del virus Zika por el medio TAN en dependencia del tiempo de contacto
|
|
|
|
|
|||
|---|---|---|---|---|---|---|
| Ct Células/medio | 2 | ND/ND | ND/ND | 30,39/32,00 | 24,15/23,90 | 23,12/22,33 |
| 15 | ND/ND | ND/ND | 31,18/32,30 | 23,21/23,40 | 23,00/21,10, | |
| 30 | ND/ND | ND/ND | 30,18/32,40 | 22,15/24,33 | 22,15/22,00 | |
Leyenda: VZk: virus Zika; Ct: ciclo umbral; ND: no detectable.
Al evaluar el desempeño clínico del medio en estudio con el diagnóstico molecular del SARS-CoV-2 por RT-PCR, previamente se estimó la pureza del ARN extraído de los ENF 1 y 2. Los valores de A260/280 de ambos grupos mostraron una pureza aceptable, y al comparar las medias (ENF1:3,44 vs. ENF2:3,27) no se obtuvo diferencia estadísticamente significativa (p = 0,2286).
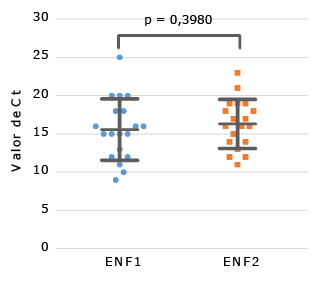
Posteriormente, se evaluaron los valores de Ct comparando los ENF1 con los ENF2 en las muestras positivas. Se constató una correlación positiva y estadísticamente significativa entre los valores de Ct de los ENF1 y 2 (r =0,5405; p =0,0169). Esto se corroboró, ya que no se identificó diferencia significativa entre las medias de los de Ct generados en ambos grupos (15,58 ± 4,00 vs. 16,32 ± 3,19; p = 0,3980) (Fig.). La sensibilidad de la RT-PCR empleando la extracción de los ENF2 fue del 100 % (IC 95 %:82,35-100), y un comportamiento similar se observó con la especificidad del 100 % (IC 95 %:71,51-100). La concordancia entre los resultados obtenidos con el diagnóstico molecular con los ENF1 y 2, fue excelente (kappa =1.0).
Discusión
La RT-PCR constituye la prueba de oro para el diagnóstico molecular de la mayoría de los virus que infectan a los humanos, y su aplicación se extiende a la caracterización genómica de estos microorganismos. Previo al diagnóstico, es importante disminuir los riesgos en el manejo y traslado de muestras biológicas potencialmente infecciosas. Por eso, en muchos laboratorios se ha extendido el uso de medios de transporte que inactivan la infectividad viral, pero al mismo tiempo garantizan la detección del ácido nucleico viral.2,7 Cabe destacar que estos no se recomiendan para las técnicas convencionales que utilizan virus viables (cultivo celular, ELISA), ya sea para el diagnóstico o la investigación.
El TRIzol es uno de los reactivos más empleados como agente desnaturalizante de virus, este se utiliza en la extracción de ácidos nucleicos, y está compuesto básicamente por fenol y tiocianato de guanidina (TCG), que tiene propiedades caotrópicas. Los compuestos que contiene la guanidina permiten estabilizar el ARN e inhibir las proteasas que lo degradan, siendo este el principal ingrediente del medio TAN.8) Teniendo en cuenta estos datos, las comparaciones del medio TAN en estudio se hicieron básicamente con el TRIzol, ya que del resto de los reactivos referidos en la literatura (AVL, RTL) se desconoce su composición.
Para evaluar la reducción de la infectividad viral en cultivos celulares por compuestos desnaturalizantes, es imprescindible eliminar o reducir el efecto citotóxico de estas sustancias, preservando las concentraciones del virus infectante. En este estudio se empleó el método de dilución del medio en estudio para reducir su citotoxicidad. Como se refiere en los resultados, el efecto citotóxico masivo del medio TAN se constató en las células Vero expuestas al inóculo sin diluir, a diferencia de los inóculos preparados a partir de las diluciones seriadas. No obstante, se constató cierto grado de toxicidad residual en la dilución 10-1, donde se observó un desprendimiento celular tanto en el control de medio TAN como en las células infectadas con VZk (medio TAN: VZk). Blow y otros identificaron en un ensayo de inactivación de Flavivirus, que el TRIzol y el AVL (QIAGEN) causaron muerte masiva de las células Vero hasta la dilución de 10-2.4 Probablemente, la concentración de guanidina o la presencia de otros reactivos no relacionados con el medio TAN, justifiquen los resultados encontrados por estos investigadores. La filtración con columnas de Sephadex LH-20 y Amicon Ultra 50 kDa es otro método que se emplea para eliminar la citotoxicidad de soluciones desnaturalizantes, las que eliminaron el 100 % de la citotoxicidad de medios de transportes de muestras basados en sustancias inactivantes como el TCG.2 Los gradientes de centrifugación con sacarosa al 20 % y 50 % han sido igualmente utilizados para eliminar el TRIzol como agente desnaturalizante, con un buen porcentaje de recuperación de virus.7
Existen varios métodos para evaluar la multiplicación viral, tales como la reducción en el número de placas en los virus que lisan las células al infectarla; el ECP que se produce en el 50 % de los cultivos infectados (TCID50); visualización de virus por microscopía electrónica, y la detección cualitativa o cuantitativa de proteínas virales y el ácido nucleico (ARN o ADN).2,7 Estas últimas macromoléculas pueden ser identificadas en el sobrenadante o en el interior de las células infectadas.
Los resultados sugieren la capacidad del medio TAN de inactivar el VZk a la dilución de 10-1, donde la viabilidad celular estaba conservada al concluir el periodo de propagación. Chan y otros observaron en un experimento con Filovirus en células 293T un desprendimiento celular significativo en ausencia de muerte celular, debido a la carencia de señales de fosforilación de regiones extracelulares de proteínas transmembrana.9 Los resultados sugieren que las muestras clínicas con una concentración de VZk ≤ 4,66 × 106ufp/mL pueden ser inactivadas al ser tratadas con medio TAN hasta una dilución de 10-1, independientemente del tiempo de contacto virus: medio.
No obstante, el desprendimiento de células observado al finalizar el ensayo a esta dilución, tanto en el inóculo de medio TAN con y sin VZk, genera la incertidumbre de que la inhibición de la replicación viral podría atribuirse a la citotoxicidad residual del medio, y no a la inactivación de la capacidad infectiva viral. Kochel y otros identificaron que una relación inicial de TRIzol: virus de 4:1 con 10 min de contacto, similar a la utilizada en este estudio, neutralizó la infectividad viral de Flavivirus (virus Dengue,106,32 TCID50/mL) y Coronavirus (MERS-CoV, 107,13 TCID50/mL). Los autores eliminaron la citotoxicidad del TRIzol por gradiente de sacarosa, a diferencia del presente estudio donde se utilizó la dilución.7
El VZk se detectó por RT-PCR a la dilución del medio TAN de 10-2 a 10-4, lo cual fue avalado por la presencia del ECP característico de la multiplicación viral, a estas diluciones. Al comparar el método de cuantificación viral por ECP con la RT-PCR, se identificó que este último tiene mayor sensibilidad, especificidad, precisión y permite una semicuantificación relativa del virus. No obstante, no se puede descartar que en estas diluciones (10-2-10-4) exista virus inactivo, y que la RT-PCR pudiera estar sobreestimando la presencia de virus infeccioso. Dado que esta técnica no distingue entre virus inactivado e infectivo, para revertir esta incertidumbre algunos autores plantean que la detección del material antigenónico, pudiera ser un marcador subrogado para evaluar la inactivación viral.10
En el contexto de la pandemia de COVID-19, Jureka y otros inactivaron el SARS-CoV-2 (106 ufp) al tratar el virus con TRIzol a una concentración final del 10 %.11 Los autores confirmaron esta inactivación al no detectar el ECP en células infectadas con el virus tratado, por tanto, concluyeron que esto permite reducir el nivel de bioseguridad para trabajar muestras biológicas potencialmente positivas al SARS-CoV-2.
Al analizar la pureza del ARN extraído a partir de la muestra de ENF en el medio CITOSWAB® VTM e inactivada con el medio TAN, los resultados obtenidos fueron similares, lo que descarta la existencia de contaminación con proteínas en los ENF2 caracterizada por una razón A260/A280 baja (< 1,7).6 Los resultados sugieren que el medio TAN, en la relación empleada en el ENF2 no interfiere en la pureza e integridad del ARN extraído con el método de QIAamp mini kit. Esto demuestra la estabilidad del ARN a las 24 horas de contacto, por lo que es apropiado para las técnicas de amplificación de biología molecular. Sin embargo, otros autores han identificado un decrecimiento en la pureza del ARN, empleando la extracción de TRIzol a partir de ENF en VTM para el diagnóstico de SARS-CoV-2.12
En cuanto al diagnóstico molecular (RT-PCR) del SARS-CoV-2, se identificó una correlación estadística entre los valores de Ct obtenidos con los ENF1 y 2. A pesar de que se estudiaron un reducido número de muestras, se obtuvo una sensibilidad, especificidad y concordancia excelente con la RT-PCR realizada a partir del ENF2, tomando como referencia el ENF1. Un resultado similar fue obtenido por Carvalho y otros, quienes utilizaron una solución desnaturalizante con una concentración de guanidina de 2,5 y 4 M para inactivar al virus SARS-CoV-2, cuya detección molecular no difirió al compararse con el medio tradicional de colecta de muestra no desnaturalizante.13 Otros autores han descrito que la capacidad de inactivar SARS-CoV-2 de medios de transporte comerciales basados en guanidina supera los 5 logs, entre ellos el primer medio de este tipo PrimeStore MTM, desarrollado originalmente para el diagnóstico de Mycobacterium tuberculosis y evaluado posteriormente para virus incluyendo SARS-CoV-2.14
En el contexto de la detección de la infección por SARS-CoV-2 por técnicas moleculares, este tratamiento de las muestras reduciría el riesgo de contaminación durante la manipulación. Sin embargo, hay que destacar que, en el caso de muestras respiratorias como los lavados broncoalveolares, se puede producir una dilución del medio TAN ≥ 10-2, lo que anularía su capacidad de inactivar la infectividad viral. No obstante, es necesario evaluar el desempeño analítico y clínico del medio TAN como transportador, desde el punto de toma de muestra, lo cual reduce los riesgos en el traslado. Asimismo, resulta de interés la posibilidad de prolongar la estabilidad de las muestras prescindiendo de la cadena de frío.
Recientemente, se validó el uso del medio de transporte desnaturalizante Copan eNAT (Italia), en la colecta y transporte de muestras respiratorias para la detección del SARS-CoV-2. Los autores constataron que este medio eliminó la infectividad viral, fue compatible con el diagnóstico molecular empleado y mantuvo la estabilidad de las muestras hasta 14 días, incluso sin cadena de frío. Esto sugiere la utilidad de este medio en países de ingresos bajos, que necesitan trasladar los especímenes clínicos desde zonas apartadas. Inclusive, esto se pudiera extender a muestras que necesiten ser trasladadas debido al colapso de laboratorios, que sobrepasaron su capacidad de procesamiento muestral.1,15
Conclusiones
Los resultados sugieren la eliminación de la infectividad viral a la dilución del medio TAN: VZk ≤ 10-1, y su compatibilidad para la detección molecular del SARS-CoV-2 propone que este medio podría ser potencialmente aplicable en la inactivación de muestras clínicas sospechosas de SARS-CoV-2; dado que es un virus envuelto similar al VZk. Sin embargo, se necesita evaluar si este medio puede garantizar de forma segura la pérdida de la infectividad viral en diferentes tipos de muestras y en situaciones donde exista una elevada concentración viral, así como definir su estabilidad para el diagnóstico virológico de la COVID-19.