My SciELO
Services on Demand
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Cubana de Hematología, Inmunología y Hemoterapia
On-line version ISSN 1561-2996
Rev Cubana Hematol Inmunol Hemoter vol.16 no.3 Ciudad de la Habana Sept.-Dec. 2000
Instituto de Hematología e Inmunología
Estudio del reordenamiento molecular de los genes TEL/AML1 en la leucemia linfocítica aguda. Resultados preliminares
Lic. Niubys Cayado Gutiérrez, Lic. Adriana Muñiz Fernández, Dr. Alejandro González Otero, Dra. Eva Svarch, Dra. Gisela Martínez AntuñaRESUMEN
La leucemia linfocítica aguda (LLA) representa aproximadamente el 80 % dentro de las leucemias pediátricas. Recientemente se ha demostrado por biología molecular la existencia de una translocación críptica, la t (12;21) (p12;q22) en la LLA de tipo B, que no se detecta por las técnicas citogenéticas convencionales e involucra los oncogenes TEL y AML1. Esta alteración es actualmente la más común en esta leucemia y se observa aproximadamente en el 25 % de los casos. Diversos investigadores han planteado que dicha translocación identifica a un subgrupo de pacientes con una evolución muy favorable, por lo que se considera un indicador de buen pronóstico. La determinación de la t (12;21) en el estudio de LLA tiene importancia pronóstica, además de servir como marcador para la detección de la enfermedad mínima residual. En nuestro trabajo estandarizamos la técnica de RT-PCR para la detección de la t (12;21) y además analizamos muestras de 20 pacientes pediátricos con LLA tipo B, que en el momento del estudio se encontraban en la fase de diagnóstico inicial o en recaída. En el estudio, 5 de los 20 pacientes mostraron reordenados los genes TEL/AML 1, lo que representa el 25 % de los casos. Esta frecuencia concuerda con lo comunicado en la literatura hasta el momento.Descriptores DeCS: LEUCEMIA LINFOCÍTICA AGUDA/diagnóstico; ONCÓGENES; NIÑOS; TRANSLOCACIÓN (GENÉTICA).
Con el descubrimiento del cromosoma Filadelfia (Ph) realizado en 1960 por Nowell y Hungerford en la leucemia mieloide crónica, se demostró la existencia de una alteración cromosómica específica en un proceso maligno humano.1 Posteriormente se identificaron otras alteraciones cromosómicas en las leucemias, en particular algunas translocaciones, que se vincularon con determinados tipos de estas enfermedades.2 Algunos investigadores consideraron que estas alteraciones eran consecuencia del proceso oncogénico. Sin embargo, este criterio cambió completamente, pues las técnicas de biología molecular pusieron en evidencia la existencia de genes específicos comprometidos en las translocaciones cromosómicas y que son activados por ellas. Esto dio lugar al establecimiento de los conceptos de protooncogenes y de oncogenes.
Se ha comprobado que algunas de estas anormalidades genéticas tienen importancia pronóstica y terapéutica en distintos tipos de leucemias. En la leucemia linfocitica aguda (LLA), que es el tipo de hemopatía maligna más frecuente en la infancia, se conocen varias alteraciones cromosómicas con importancia pronóstica. Entre ellas tenemos la t(4;11) y la t(9;22) que son indicadores de mal pronóstico, mientras que la hiperdiploídia se asocia con buen pronóstico.3
Estudios moleculares recientes demostraron que la t(12;21), que no se detecta por las técnicas citogenéticas convencionales, es la alteración más frecuente en la LLA de tipo B (LLA-B) y se observa aproximadamente en el 25 % de los casos.4,5 En esta translocación el gen TEL, localizado en la región telomérica del cromosoma 12, se fusiona con el gen AML 1, localizado en el cromosoma 21. El gen TEL codifica para un factor de transcripción de la familia ETS y el gen AML 1 codifica una proteína que forma parte del complejo transcripcional AML1/CBFb (core binding factor). Como consecuencia de la translocación se origina un gen híbrido que da lugar a la síntesis de una proteína también híbrida, cuyo extremo N-terminal es un segmento del gen TEL y el C-terminal es un segmento del gen AML 1. Esta proteína híbrida TEL-AML 1 tiene propiedades anormales y por lo tanto, afecta la función normal del complejo transcripcional AML 1/CBFb y por ende, la síntesis de las proteínas que este complejo controla.6
Recientemente un gran número de investigaciones clínicas han demostrado que la t(12;21) identifica a un subgrupo de pacientes clínicamente diferentes con una evolución muy favorable, por lo que se considera esta translocación un indicador de buen pronóstico.7-9 En estos casos la técnica utilizada fue RT-PCR (reverso transcripción y reacción en cadena de la polimerasa), que ha contribuido extraordinariamente a obtener un diagnóstico más preciso y ha permitido también la detección de la enfermedad mínima residual (EMR) con una mayor sensibilidad en otras leucemias.
El objetivo de nuestro trabajo fue la introducción del estudio molecular de la t(12;21) en nuestra institución, teniendo en cuenta la importancia que el hallazgo de esta alteración tiene en el diagnóstico, pronóstico y seguimiento de los pacientes con LLA-B. Se muestran los resultados preliminares obtenidos.
MÉTODOS
Pacientes
Se estudió un total de 20 pacientes pediátricos con diagnóstico de LLA-B, en edades que oscilaron entre 1 y 14 años, con un promedio de 7,3 años. En el momento del estudio, 18 pacientes se encontraban en la etapa de diagnóstico inicial y 2 en recaída hematológica.Técnica de reverso transcripción-reacción en cadena de la polimerasa (RT-PCR)
El aislamiento de leucocitos de muestras de médula ósea o sangre periférica se realizó mediante un gradiente de Ficoll-Telebrix 38 según el método convencional. Posteriormente se procedió a la purificación del ARN por el método de fenolcloroformo.10 La integridad del ARN se comprobó con una electroforesis en gel de agarosa al 1,5 %.La síntesis del ADNc con la enzima reverso transcriptasa y la técnica de PCR para la amplificación del ADNc de la fusión TEL/AML 1 se hizo según el protocolo de BIOMED.9 Las reacciones de PCR fueron llevadas a cabo en una máquina MJ-Research, INC. Versión 1.2. Las condiciones de los ciclos utilizados para la PCR fueron las siguientes: una desnaturalización inicial a 94 ° C de 1 min; posteriormente se repitieron un total de 35 ciclos de desnaturalización a 94 ° C, 1 min; alineación a 65 ° C, 1 min; elongación a 72 ° C, 1 min, y finalmente las muestras se enfriaron a 5 ° C. Se realizó una segunda reacción de PCR (anidado o nested) con las mismas condiciones descritas, cambiando solamente los oligonucleótidos.
En todas las reacciones se utilizó como control positivo una línea celular establecida que tiene reordenados los genes TEL/AML 1 y un control negativo que contiene todos los reactivos de PCR menos ADNc. Como control interno se utilizó el gen de la cadena a del ácido retinoico (RARa). Los oligonucleótidos empleados fueron sintetizados por Amersham Pharmacia Biotech. Las secuencias se muestran a continuación:
Para la fusión TEL-AML 1
Primer PCR:
TEL-A TGC ACC CTC TGA TCC TGA AC
AML-B AAC GCC TCG CTC ATC TTG C
PCR anidado:
TEL-C AAG CCC ATC AAC CTC TCT CAT C
AML-1-D TGG AAG GCG GCG TGA AGC
Para el control normal (RAR).
R2 GCT CTG ACC ACT CTC CAG CA
R5 CCA CTA GTG GTA GCC TGA GGA CT
El patrón de la electroforesis se observó en gel de agarosa al 2 %. El marcador de peso molecular utilizado fue Phi X 174/Hae III.
RESULTADOS
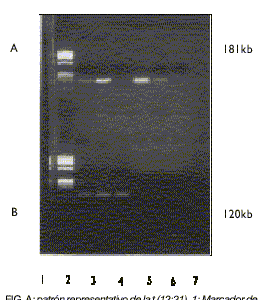
Los resultados obtenidos en el presente trabajo fueron los siguientes: de los 20 pacientes estudiados, 5 (25 %) mostraron la banda de reordenamiento de los genes TEL/AML 1 (fig.). Los 15 casos restantes resultaron negativos para la t(12; 21).
FIG. A: patrón representativo de la t (12;21). 1: Marcador de peso molecular Phi X-174-Haell. 2 y 3: Pacientes positivos para la t (12;21). 4: Paciente negativo para la t (12;21). 5 y 6: Controles positivos. 7: Control negativo. B: patrón representativo del gen RAR. 1: Igual que A-1. 2-4: Banda de amplificación del control interno (RAR) de los pacientes.
DISCUSIÓN
Los estudios moleculares realizados por diversos investigadores han demostrado que la t(12; 21), que fusiona los oncogenes TEL y AML 1, es la más frecuente en la LLA-B infantil, y ocurre aproximadamente en el 25 % de los casos.4 Adicionalmente, los estudios de seguimiento han demostrado que esta translocación se encuentra asociada con buen pronóstico, aunque el mecanismo mediante el cual actúa el gen híbrido TEL-AML 1 se desconoce aún.7En nuestro estudio el 25 % de los pacientes mostraron esta anormalidad, por lo que nuestros resultados coinciden con lo descrito en la literatura.
Consideramos que haber introducido esta técnica en nuestro laboratorio, para el estudio molecular de las leucemias es de gran importancia, pues la t(12;21), por su frecuencia e importancia pronóstica, es el marcador molecular más importante que tiene en la actualidad la LLA-B infantil. Su detección en un paciente al diagnóstico de la enfermedad, también permite utilizarla posteriormente para determinar la EMR por técnicas moleculares de gran sensibilidad.
El estudio de la t(12;21) constituye un primer paso en la clasificación genética de la LLA-B y representa el primer ejemplo en el cual una anormalidad genética está asociada con un pronóstico favorable en la leucemia infantil. La caracterización molecular de otras alteraciones cromosómicas permitirá con toda seguridad identificar nuevos marcadores genéticos con importancia pronóstica y contribuirá a mejorar el diagnóstico, tratamiento y seguimiento de los pacientes con leucemia.
SUMMARY
Acute lymphocytic leukemia (ALL) represents approximately 80% of the pediatric leukemias.The existance of a cryptic translocation, the t (12;21) (p12;q22) in the type B ALL, which is not detected by the conventional cytogenetic techniques and involve the TEL and AML1 oncogenes, has been recently shown by molecular biology. This alteration is at present the most common in this leukemia and it is observed approximately in 25% of the cases. Some authors have stated that such translocation identifies a group of patients with a very favorable evolution and that's why it is considered as an indicator of good prognosis. The determination of the t (12;21) in the study of ALL has a prognostic importance and it also serves as a marker for the detection of the minimal residual disease. In our paper, we standardized the RT-PCR technique for the detection of the t (12;21) and we also analized samples from 20 pediatric patients with type B ALL, which at the time of the study was in the phase of initial diagnosis or on relapse. In the study, 5 of the 20 patients showed rearranged TEL/AML l genes, which accounted for 25% of the cases. This frequency agrees with what is reported in literature up to now.Subject headings: LEUKEMIA, LYMPHOCYTIC, ACUTE/diagnosis; ONCOGENES; CHILD; TRANSLOCATIONS (GENETIC).
REFERENCIAS BIBLIOGRÁFICAS
1. Nowell PC, Hungerford DA. A minute chromosome in human chronic granulocytic leukemia. Science 1960;132:1497-9.
2. Rowley JD. Recurring chromosome abnormalities in leukemia and lymphoma. Semin Hematol 1990;27:122-36.
3. Rubnitz JE, Behm FG, Pui CH, et al. Genetic studies of childhood acute lymphoblastic leukemia with emphasis on p16, MLL and ETV6 gene abnormalities: results of St Jude total therapy study XII. Leukemia 1997;11:1201-6.
4. Shurtleff SA, Bujis A, Behm FG, et al. TEL/AML 1 fusion resulting from a cryptic t (12;21) is the most common genetic lesion in pediatric ALL and defines a subgroup of patients with an excellent prognosis. Leukemia 1995;9:1985-9.
5. Borkhardt A, Cazzaniga G, Viehmann S, et al: Incidence and clinical relevance of TEL/AML 1 fusion genes in children with acute lymphoblastic leukemia enrolled in the German and Italian multicenter therapy trials. Blood 1997;90:571.
6. Liang DC, Chou TB, Chen JS, et al. High incidence of TEL/AML 1 fusion resulting from cryptic t (12;21) in childhood B-lineage acute lymphoblastic leukemia in Taiwan. Leukemia 1996;10:991-3.
7. Rubnitz JE, Downing JR, Pui CH, et al. TEL gene rearrangement in acute lymphoblastic leukemia: a new genetic marker with prognostic significance. J Clin Oncol 1997;15:1150-7.
8. McLean TW, Ringold S, Neuberg D, Stegmainer KR, Lantravalní R, Ritz J, et al. TEL/AML 1 dimerizes and is associated with a favorable outcome in childhood acute lymphoblastic leukemia. Blood 1996;88:4252-8.
9. Seeger K, Adams HP, Buchwald D, et al. TEL-AML 1 fusion transcript in relapsed childhood acute lymphoblastic leukemia. Blood 1998;91:1716-22.
10. Maniatis T, Fritsch EF, Sambrook J. Molecular cloning. A laboratory manual. New York: Cold Spring, 1982.
Lic. Niubys Cayado Gutiérrez. Instituto de Hematología e Inmunología.Apartado 8070, CP 10800, Ciudad de La Habana, Cuba. Teléf.: (537) 578268. Fax: (537) 338979. e-mail: ihidir@hemato.sld.cu