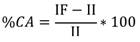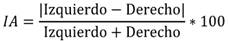1.-Introducción
Las mediciones de resonancia magnética estructural (RME) para monitorear la progresión de la Enfermedad de Alzheimer (EA) se están volviendo fundamentales en la práctica clínica, y más en el contexto de los estudios longitudinales. Se ha demostrado que la reducción del volumen del hipocampo tiene una fuerte asociación con la progresión de la enfermedad. Además, tiende a ser registrado de manera más consistente en todos los estudios epidemiológicos, tanto de forma transversal como longitudinal, en relación a otros marcadores. En un trabajo anterior se describió la metodología utilizada para el procesamiento cross-sectional de las imágenes de resonancia magnética (IRM) [1]. Sin embargo, su utilización está pensada para estudios transversales, y no para estudios evolutivos. El desarrollo de ensayos clínicos en Cuba necesita de biomarcadores para documentar el efecto de los medicamentos. Por ello, esta investigación describe y valida un método para procesar de forma longitudinal y automática, el volumen del hipocampo como biomarcador imagenológico de la EA. El trabajo compara los resultados reportados (derivatives) del procesamiento cross-sectional con FreeSurfer 6.0 de las IRM de un grupo de sujetos sanos de la base de datos internacional ADNI 3 (del inglés, Alzheimer's Disease Neuroimaging Initiative) [2] con los obtenidos en este trabajo. Esta comparación permite validar el pipeline implementado en CNEURO y comprobar si existe similitud entre ambos resultados. Queda por tanto establecida la secuencia de pasos para el procesamiento longitudinal de las IRM. Esta metodología se utiliza para estudiar la variación temporal del volumen del hipocampo y el índice de asimetría de una muestra de sujetos cubanos diagnosticados con EA.
2.- Metodología
2.1.- Sujetos participantes
En este estudio participaron dos grupos de sujetos: sanos y diagnosticados con EA. El grupo de sujetos enfermos estuvo conformado por 20 individuos cubanos (71.35 ± 6.35 años) diagnosticados siguiendo los criterios del Instituto Nacional de Envejecimiento- Asociación de Alzheimer (NIA-AA, del inglés, National Institute on Aging - Alzheimer's Association) del año 2011 [3] y los criterios de grado leve/moderado según GDS (del inglés, Global Deficit Score), apoyados además en el diagnóstico clínico-neuropsicológico. Se obtuvo el consentimiento informado de los sujetos incluidos y la aprobación del Comité de Ética de la Investigación Clínica (CEIC) de las instituciones involucradas. El otro grupo estuvo formado por 20 individuos sanos (71.25 ± 5.562 años), pertenecientes a la base de datos internacional ADNI 3. Se seleccionaron los sujetos de esta base de datos porque se reportan los derivatives; y además constituye una referencia a nivel mundial para investigaciones en neuroimágenes y marcadores biológicos de la EA.
2.2.- Adquisición y procesamiento de las imágenes
Las secuencias de pulso utilizadas para adquirir las imágenes son las recomendadas por la ADNI 3. Estas son optimizadas para usarse en el escáner de IRM Siemens Magnetom Allegra 3.0 T, instalado en el Centro de Neurociencias de Cuba (CNEURO). Para realizar la segmentación automática del hipocampo en este trabajo se utilizó la secuencia T1-3D MP-RAGE (tamaño de vóxel [mm3]: 0.8 x 0.8 x 1; tiempo [ms]: TE = 2.6; TR = 2000, TI = 900; duración [min]: 9:19). La secuencia 3D se adquirió con orientación sagital y se reconstruyeron axial y coronal.
El software de segmentación automática FreeSurfer 6.0 (http://surfer.nmr.mgh.harvard.edu/), fue el utilizado para el procesamiento longitudinal de las imágenes. Esta herramienta es altamente demandante de recursos computacionales y del empleo de técnicas de paralalelización de procesos. Por tanto, es impracticable su ejecución en una PC convencional para procesar múltiples sujetos. Por ello, este programa fue ejecutado sobre una plataforma de hardware de altas prestaciones con la siguiente arquitectura: Clúster Linux de 5 nodos de cómputo con 2 CPU Intel Xeon 12 Core a 2.4Ghz, 48GB RAM, dos HDD de 300GB de almacenamiento cada uno, teniendo un performance pico teórico de 4 TFLOPS. Esta arquitectura permite realizar el despacho de hasta 24 hilos de ejecución por nodo.
Para el análisis longitudinal de cada sujeto de ambos grupos se utilizan imágenes en dos instantes de tiempo espaciados por un año. Las variables a medir son el volumen y la asimetría del hipocampo. Para calcular el prociento de cambio anual (%CA) del volumen del hipocampo se usa la siguiente ecuación:
En este trabajo se normalizan los volúmenes del hipocampo respecto al volumen total intracraneal eTIV (del inglés, estimated total intracraneal volume) porque esto facilita la interpretación y la comparación de los resultados con otros estudios [4]. El índice de asimetría (IA) fue calculado a partir de las diferencias de los valores absolutos de los volúmenes del hipocampo izquierdo y derecho normalizados por el eTIV, utilizando la siguiente fórmula:
2.3.- Métodos estadísticos
Se realizaron los siguientes procesamientos estadísticos con el programa IBM SPSS Statistics (versión 26):
Se comprueba la normalidad de los datos con una prueba de Kolmogorov-Smirnof. Si estos poseen una distribución normal, entonces se realiza la comparación entre las mediciones de ADNI 3 y el pipeline:
Para comparar los resultados del procesamiento cross-sectional de los sujetos de ADNI 3 con los obtenidos por el pipeline descrito, fue utilizada la prueba t-student no pariada de dos colas (p < 0.05)
Para evaluar la concordancia general entre las mediciones realizadas por ADNI 3 y las obtenidas con el pipeline, se utilizó el gráfico de Bland-Altman con 95% de intervalo de confianza.
Para calificar la fuerza del acuerdo de ambas mediciones se utilizó el coeficiente de correlación intraclase (CCI) con un 95% de intervalo de confianza [Modelo: Alpha, Mixto de dos factores, tipo: acuerdo absoluto]
Análisis de los resultados del procesamiento longitudinal a ambos grupos de sujetos:
Gráfico de violín para estudiar el comportamiento de los valores medios del porciento de cambio anual (%CA) del volumen del hipocampo.
Prueba t-student no pariada de una cola (p < 0.05) para conocer si existe diferencias significativas entre ambos grupos.
Si los datos no poseen una distribución normal, se utilizará la estadística no paramétrica correspondiente a las anteriormente mencionadas.
2.4.- Fundamentación del uso del procesamiento longitudinal
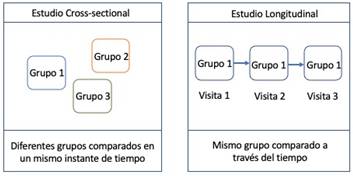
La elección del procesamiento cross-sectional o longitudinal depende del diseño de la investigación. En un estudio transversal, se recopilan datos de una población en un momento específico; en un estudio longitudinal, se recopilan repetidamente datos de la misma muestra durante un período de tiempo prolongado (figura 1). Los estudios longitudinales permiten investigar los cambios de las variables en el tiempo usando al sujeto como su propio control. Por tanto, los cambios en los biomarcadores son atribuibles a efectos del tratamiento o a cambios biológicos y no a diferencias casuales entre individuos. La utilización del procesamiento longitudinal de FreeSurfer en estudios evolutivos tiene disímiles ventajas, entre ellas: mayor sensibilidad a cambios estructurales y mayor precisión en la clasificación estructuras cerebrales [5].
Esto se facilita con la creación de una plantilla intermedia con las imágenes tomadas en diferentes instantes de tiempo para cada sujeto. Se disminuyen de esta forma errores de procesamiento por la presencia de artefactos en la imagen, cambios de posición del paciente y otros problemas no atribuibles al fenómeno biológico. Además, el procesamiento longitudinal facilita el análisis grupal de los biomarcadores incluidos en un estudio. Investigaciones previas han demostrado la superioridad del procesamiento longitudinal de FreeSurfer [6].
2.5.- Secuencia de pasos del procesamiento longitudinal con freesurfer 6.0
Tras una revisión del estado del arte para la realización de estudios longitudinales [5, 6] de al menos dos IRM cerebrales con 12 meses de diferencia, se usan tres pasos para el procesamiento. El primero es el procesamiento transversal (cross-sectional), el segundo es la conformación de la plantilla base y el tercero es el procesamiento longitudinal que utiliza los resultados de los pasos anteriores. La línea de comando es:
Paso 1.
recon-all -s subject_v1.cross -i subject_v1.nii -all -hippocampal-subfields-T1 -brainstem-structure -3T
recon-all -s subject_v2.cross -i subject_v2.nii -all -hippocampal-subfields-T1 -brainstem-structure -3T
siendo:
<recon-all> comando que inicia la segmentación de las estructuras cerebrales,
<subject_v1.cross> y <subject_v2.cross> carpetas de salida como resultado del procesamiento cross-sectional de cada visita,
<subject_v1.nii> y <subject_v2.nii> ficheros de entrada que contienen las imágenes estructurales del paciente en formato nifti de ambas visitas,
<-hippocampal-subfields-T1> flag (indicador) específico para realizar la segmentación de los subcampos del hipocampo,
<-brainstem-structure> flag específico para realizar la segmentación de las estructuras del tronco encefálico,
<-3T> flag específico para corregir la intensidad de las imágenes tomadas con equipos de 3T.
<-all > flag para indicar que se realizarán todas las operaciones del procesamiento.
Es una sola línea de comando que se repetirá tantas veces como visitas tenga cada sujeto. En este trabajo sólo fueron tomadas dos instantes de tiempo (visitas), por ello solo se ejecutó el comando dos veces por sujeto.
Al finalizar el procesamiento, se comprueba que “terminó sin errores”. Si hay un error llamado: "error en la transformación de Talairach", se volverá a ejecutar la línea de comando anterior agregando al comando recon-all el flag: - notalairach. Al agregarlo, FreeSurfer ignorará la verificación de transformación de Talairach y continuará con la ejecución del resto de instrucciones. Otro error probable es cuando se ejecuta la misma línea de comando dos veces. Entonces en consola aparecerá el error: "este comando todavía se está ejecutando en segundo plano". Para solucionarlo, se vuelve a ejecutar la línea de comando agregando el flag: - no-isrunning[5].
El segundo paso consiste en la creación de la plantilla llamada “base”. Esta es creada a partir de los resultados obtenidos del procesamiento de todas las visitas del sujeto, explicadas en el paso 1. Para ello se debe ejecutar el siguiente comando:
Paso 2.
recon-all -base subject.base -tp subject_v1.cross -tp subject_v2.cross -all
siendo:
<-base> flag indicador del cálculo de la plantilla base,
<-tp> flag indicador de time point,
<subject.base> la carpeta de salida con los resultados del procesamiento de la plantilla “base” y
<subject_v1.cross> y <subject_v2.cross> las salidas del procesamiento del Paso 1.
El tercer y último paso es el procesamiento longitudinal, que lleva a un espacio común la plantilla “base” con los diferentes procesamientos de cada visita. Esto permite ver cambios en los diferentes puntos de tiempo. Se realiza usando la siguiente línea de comando [5]:
Paso 3.
recon-all -long subject_v1.cross subject.base -all
recon-all -long subject_v2.cross subject.base -all
Al igual que el paso 1, este comando se ejecutará la misma cantidad de veces que visitas tenga el sujeto. Como resultado se obtendrá una carpeta de salida con el nombre estructurado de la siguiente forma: subject_vn.cross.long.subject.base
La ejecución de los pasos anteriormente descritos se puede automatizar mediante la programación de un script que procese todos los casos del estudio automáticamente. De ocurrir fallas en la segmentación de algún sujeto, su correción se realiza manualmente y se recalcula lanzando el comando del paso correspondiente.
2.6.- Control de calidad de las segmentaciones y corrección de errores
Cuando todos los pasos terminan, se hace el control de calidad (CC) de las segmentaciones comenzando con la inspección visual del archivo "aseg.presurf.mgz" de la carpeta "mri" para asegurar que es correcta. Se recomienda hacer el CC comenzando por el último paso hasta el primero, evaluando en cada caso si es necesario realizar correcciones para recalcular los valores. Si se observa un error, se debe corregir la segmentación editando el archivo "aseg.presurf.mgz" usando Freeview. Los siguientes comandos recalculan las estadísticas del archivo "aseg.stat" para cada paso:
Cross-sectional [5]:
recon-all -autorecon2-noaseg -autorecon3 -subjid subject_vn.cross
Base:
recon-all -base subject.base -tp subject_v1.cross -tp subject_v2.cross -tp subject_vn.cross -autorecon2 -autorecon3
Longitudinal:
recon-all -autorecon2-noaseg -autorecon3 -subjid subject_vn.cross.long.subject.base -long subject_vn.cross subject.base
Si se usa esta parte del procesamiento no se usa el flag <-all> pues se realizaron solo las operaciones <-autorecon2> y -<autorecon3>.
Al finalizar el procesamiento se utiliza el comando "aseg2table" de FreeSurfer para recopilar las estadísticas en un archivo ".csv" e iniciar el análisis estadístico.
3.- Resultados y discusión
3.1.- Validación del procesamiento cross-sectional
Para validar el pipeline descrito anteriormente se compararon los resultados obtenidos del procesamiento cross-sectional en dos instantes de tiempos de 20 sujetos sanos de la base de datos ADNI 3 con los derivatives reportados por dicho estudio. La principal ventaja de trabajar con volúmenes sin normalizar, es su uso en comparaciones de estudios cruzados y el potencial para crear valores volumétricos normativos para diferentes intervalos de edad [4].
Como los datos cumplen una distribución normal (p> 0.1000) las comparaciones se realizan con la t-student. El resultado de esta prueba no mostró diferencias significativas entre ambas mediciones (tabla 1), realizadas en dos instantes de tiempos diferentes.
Tabla 1 Comparación de los resultados usando la prueba t-student
| Visita 1 | Visita 2 | |||
|---|---|---|---|---|
| Procesamiento | Hipocampo Izquierdo | Hipocampo Derecho | Hipocampo Izquierdo | Hipocampo Derecho |
| Pipeline (CNEURO) vs ADNI derivatives | p= 0.8814 | p= 0.9759 | p= 0.9773 | p=0.9156 |
Para evaluar de forma visual la concordancia general entre ambas mediciones, se realizó el análisis del gráfico de Bland-Altman para la visita 1 (figura 2). La diferencia fue de 25.78 mm3; DE = 106.5 (línea central continua) para el hipocampo izquierdo, y para el hipocampo derecho la diferencia fue de 5.28 mm3; DE = 87.62 (línea central continua). Las líneas de concordancia están expresadas como M ± 1.96* DE y corresponden a las líneas exteriores discontinuas. A pesar de ser la medición con menor valor de p, para ambos hipocampos el gráfico mostró buena concordancia de los resultados. Las diferencias se encontraron cercanas a la media, dentro del intervalo de confianza (95%) y sin describir una tendencia relacional.
El cálculo del CCI mostró buena fiabilidad, con CCI > 0.97 para ambos hipocampos en ambas visitas como se muestra en la tabla 2.
Estos resultados demuestran que las mediciones realizadas utilizando el pipeline (CNEURO) concuerdan con las reportadas en el estudio ADNI 3. Por tanto, la metodología de trabajo descrita es válida para la cuantificación volumétrica del hipocampo.

Figura 2 Análisis de concordancia mediante el gráfico Bland-Altman de los valores de volumen de hipocampo izquierdo y derecho, calculados por ADNI y los obtenidos por el pipeline. DE: desviación estándar.
Tabla 2 Valores del CCI entre las mediciones reportadas por ADNI 3 y las obtenidas con el pipeline en ambas visitas
| Visita 1 | Visita 2 | |||
|---|---|---|---|---|
| Hip Izq | Hip Der | Hip Izq | Hip Der | |
| ADNI 3 vs Pipeline (CNEURO) | CCI= 0.990 LS= 0.996 LI= 0.976 | CCI= 0.994 LS= 0.998 LI=0.985 | CCI= 0.992 LS= 0.997 LI= 0.980 | CCI= 0.997 LS= 0.999 LI= 0.993 |
LS: límite superior, LI: límite inferior [Intervalo de confianza del 95%]
3.2.- Resultados del procesamiento longitudinal
Los dos grupos de sujetos estudiados estuvieron balanceados en edad y sexo, con promedio de edad de 71 años y mayor cantidad de mujeres que hombres. Esta distribución es común en muestras de sujetos con EA. Reportes previos de la literatura afirman una mayor incidencia en mujeres que en hombres y con edades superiores a los 65 años de edad [7, 8].
Los resultados del procesamiento longitudinal realizado a cada grupo siguiendo el pipeline descrito se muestran en la tabla 3. Tras realizar el control de calidad de las segmentaciones, no fue necesario realizar correcciones.
Las mediciones longitudinales del volumen del hipocampo muestran, para ambos grupos, una disminución respecto al valor inicial. Dicha disminución es parte del envejecimiento normal, pero en el caso de los enfermos es mucho mayor. Se conoce que en individuos sanos, el volumen del hipocampo disminuye de 1.2-1.4% anual y en 4.3% o superior en pacientes con EA [9].
Al realizar el cálculo del %CA de cada hipocampo de cada grupo, se obtuvo para el grupo sano un 0.98% para el hipocampo izquierdo y 0.63% para el hipocampo derecho. Y para el grupo enfermo un 2.51% y 2.41% respectivamente. Estos resultados se encuentran en el rango de los valores esperados para cada grupo. Dicho valor ha sido reportado a partir del estudio de grandes bases de datos. A pesar de ello, el grupo enfermo reporta un %CA que es 2-3 veces mayor que el correspondiente al grupo de sujetos sanos, encontrándose diferencias significativas entre el hipocampo izquierdo (p= 0.0156) y derecho (p= 0.0157) entre ambos grupos (figura 3a). Estos resultados cohinciden con reportes previos de la literatura que afirman un mayor grado de atrofia en sujetos enfermos respecto a sanos [10].
Tabla 3 Resultados de las mediciones longitudinales realizadas a cada grupo
| ADNI3 (CN) | Cubanos (EA) | ||||
|---|---|---|---|---|---|
| Cantidad de sujetos | 20 | 20 | |||
| Edad (Media ± DE) (años) | 71.25 ± 5.56 | 71.35 ± 6.35 | |||
| [Edad mínima, Edad máxima](años) | [61, 81] | [62, 82] | |||
| 25% Percentil (años) | 66.25 | 64.25 | |||
| Mediana (años) | 71 | 71 | |||
| 75% Percentil (años) | 76.75 | 77.75 | |||
| Sexo, % de mujeres | 70 % | 80 % | |||
| Visita 1 | Volumen hipocampo izquierdo (Media ± DE) mm3 | 3636 ± 518.2 | 2949 ± 485.7 | ||
| Volumen hipocampo derecho (Media ± DE) mm3 | 3792 ± 559.7 | 3048 ± 444.6 | |||
| Vol normalizado hipocampo izquierdo (Media ± DE) | 0.00241 ± 0.00030 | 0.00212 ± 0.00028 | |||
| Vol normalizado hipocampo derecho (Media ± DE) | 0.00252 ± 0.00034 | 0.00219 ± 0.00027 | |||
| Índice de asimetría, % de los hipocampos (Media ± DE) | 2.69 ± 1.8 | 4.81 ± 4.0 | |||
| Visita 2 | Volumen hipocampo izquierdo (Media ± DE) mm3 | 3608 ± 548.7 | 2884 ± 516.5 | ||
| Volumen hipocampo derecho (Media ± DE) mm3 | 3769 ± 552.4 | 2991 ± 506.0 | |||
| Volumen norma hipocampo izquierdo (Media ± DE) | 0.00240 ± 0.00032 | 0.00207 ± 0.00029 | |||
| Volumen norma hipocampo derecho (Media ± DE) | 0.00250 ± 0.00034 | 0.00215 ± 0.00031 | |||
| Índice de asimetría, % de los hipocampos (Media ± DE) | 2.61 ± 2.0 | 5.15 ± 4.2 | |||
| %CA Volumen del Hipocampo (Media ± DE) | Hipocampo Izquierdo | 0.98 ± 2.9 | 2.51 ± 3.3 | ||
| Hipocampo Derecho | 0.63 ± 1.9 | 2.41 ± 3.7 | |||
Vol normalizado: volumen normalizado con el eTIV, CN: controles normales, EA: Enfermedad de Alzheimer
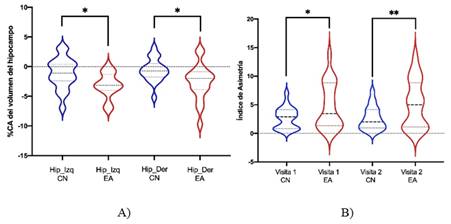
Fig. 3 Gráficos de violin de %CA e IA de ambos hipocampos para sujetos sanos (color azul) y enfermos (color rojo). En ella se observa la existencia de diferencias significativas entre los sujetos sanos y los enfermos.
Otra medición que confirma lo aterior, es el IA (figura 3b). Las comparaciones entre grupos mostraron diferencias significativas de un nivel significación (p< 0.0196) entre grupos para la visita 1, y dos niveles de significación (p< 0.0099) para la visita 2. Estos resultados indican una mayor la asimetría del hipocampo en los sujetos enfermos respecto a los sanos, con una prevalencia de asimetría lateral derecha. Nuevamente esto cohincide con reportes previos de la literatura [11, 12]. Estos resultados confirman el diagnóstico de las pruebas neuropsicológicas y demuestran la robustez del método de medición.
4.- Conclusiones
En este trabajo quedó descrito y validado un procedimiento longitudinal para la cuantificación volumétrica automática de estructuras cerebrales. Quedó demostrada su utilidad y fiabilidad al mostrar resultados similares a los reportados en bases de datos internacionales. Con esto se pone a disposición de los investigadores cubanos la secuencia de pasos para usar el volumen del hipocampo como biomarcador imagenológico de la EA en ensayos clínicos.